DNA polymeráza I - DNA polymerase I
| DNA polymeráza I | |||||||
|---|---|---|---|---|---|---|---|
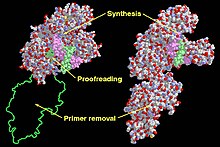 Funkční domény v Klenowově fragmentu (vlevo) a DNA polymerázy I (vpravo). | |||||||
| Identifikátory | |||||||
| Organismus | |||||||
| Symbol | polA | ||||||
| Entrez | 948356 | ||||||
| PDB | 1 DPI | ||||||
| RefSeq (Prot) | NP_418300.1 | ||||||
| UniProt | P00582 | ||||||
| Další údaje | |||||||
| EC číslo | 2.7.7.7 | ||||||
| Chromozóm | genom: 4,04 - 4,05 Mb | ||||||
| |||||||
DNA polymeráza I (nebo Pol I) je enzym který se účastní procesu prokaryotiky replikace DNA. Objevil Arthur Kornberg v roce 1956,[1] bylo to první známé DNA polymeráza (a první známý z jakéhokoli druhu polymeráza ). To bylo původně charakterizováno v E-coli a je všudypřítomný v prokaryoty. v E-coli a mnoho dalších bakterií, gen který kóduje Pol I je známý jako polA. The E-coli forma enzymu se skládá z 928 aminokyselin a je příkladem a procesivní enzym - může postupně katalyzovat více polymerací bez uvolnění jednovláknového templátu.[2] Fyziologickou funkcí Pol I je hlavně opravit jakékoli poškození pomocí DNA, ale také slouží k propojení Okazaki fragmenty odstraněním RNA primerů a nahrazením řetězce DNA.
Objev
V roce 1956 Arthur Kornberg a jeho kolegové objevili Pol I pomocí Escherichia coli (E-coli) extrakty k vývoji testu syntézy DNA. Vědci dodali 14C označený thymidin, aby bylo možné získat radioaktivní polymer DNA, nikoli RNA. Vědci přidali, aby zahájili čištění DNA polymerázy streptomycin sulfát do E-coli extrakt, který vytvořil sraženinu, která se skládala ze supernatantu neobsahujícího nukleové kyseliny (S-frakce) a sraženiny obsahující nukleovou kyselinu (P-frakce). Bylo zjištěno, že P-frakce obsahovala Pol I a tepelně stabilní faktory, které byly nezbytné pro to, aby reakce syntézy DNA byly vystaveny extrémním teplotám. Tyto faktory byly identifikovány jako nukleosid trifosfáty, stavební kameny nukleových kyselin. S-zlomek obsahoval více deoxynukleosid kinázy.[3] V roce 1959 byla Nobelova cena za fyziologii nebo medicínu udělena Arthurovi Kornbergovi a Severo Ochoa „za objev mechanismů zapojených do biologické syntézy Ribonukleová kyselina a Deoxyribonukleová kyselina."[4]

Struktura a funkce
Obecná struktura
Pol I funguje hlavně při opravách poškozené DNA. Pol I je součástí třídy proteinů nadrodiny alfa / beta, která se skládá z alfa a beta segmentů, které jsou rozptýleny po daném proteinu. E-coli DNA Pol I se skládá ze čtyř domén se dvěma samostatnými enzymatickými aktivitami. Čtvrtá doména se skládá z exonukleáza který korektuje produkt DNA Pol I a je schopen odstranit všechny chyby spáchané Pol I. Další tři domény spolupracují na udržení aktivity DNA polymerázy.[5]
E-coli bakterie obsahuje 5 různých DNA polymeráz: DNA Pol I, DNA Pol II, DNA Pol III, DNA Pol IV a DNA Pol V. Eukaryotické buňky obsahují 5 různých DNA polymeráz: α, β, γ, δ a ε.[6] Eukaryotická DNA polymeráza β je nejpodobnější E-coli DNA Pol I, protože jeho hlavní funkce je spojena spíše s opravou DNA než s replikací. DNA polymeráza β se používá hlavně při bazální excizi-opravě a nukleotidové excizi.[7] Bylo identifikováno celkem 15 lidských DNA polymeráz.[8]
Strukturní a funkční podobnost s jinými polymerázami

Při replikaci DNA se přední řetězec DNA kontinuálně prodlužuje ve směru pohybu vidlice replikace, zatímco řetězec zaostávající DNA běží diskontinuálně v opačném směru jako Okazaki fragmenty.[9] DNA polymerázy také nemohou iniciovat řetězce DNA, takže musí být iniciovány krátkými segmenty RNA nebo DNA známými jako primery.[5] Aby mohla polymerace DNA proběhnout, musí být splněny dva požadavky. Nejprve musí mít všechny DNA polymerázy jak vlákno templátu, tak vlákno primeru. Na rozdíl od RNA nemohou DNA polymerázy syntetizovat DNA z řetězce templátu. Syntéza musí být zahájena krátkým segmentem RNA, známým jako RNA primer, syntetizoval Primase ve směru 5 'až 3'. Syntéza DNA pak probíhá přidáním dNTP k 3 'hydroxylové skupině na konci již existujícího řetězce DNA nebo primeru RNA. Zadruhé, DNA polymerázy mohou pouze přidat nové nukleotidy k již existujícímu řetězci pomocí vodíkové vazby.[6] Vzhledem k tomu, že všechny DNA polymerázy mají podobnou strukturu, všechny sdílejí mechanismus polymerázy katalyzovaný dvěma kovovými ionty. Jeden z kovových iontů aktivuje 3 'hydroxylovou skupinu primeru, který potom napadá primární 5' fosfát dNTP. Druhý kovový iont stabilizuje záporný náboj zbývajícího kyslíku a následně chelatuje dvě vystupující fosfátové skupiny.[10]
Říká se, že rentgenové struktury polymerázové domény všech DNA polymeráz připomínají struktury lidské ruky. Všechny DNA polymerázy obsahují tři domény. První doména, která je známá jako „doména prstů“, interaguje s dNTP a základnou spárované šablony. „Doména prstů“ také interaguje se šablonou tak, aby byla správně umístěna na aktivním webu.[11] Druhá doména, známá jako „palmová doména“, katalyzuje reakci přenosu fosforylové skupiny. A konečně, třetí doména, která je známá jako „doména palce“, interaguje s dvouvláknovou DNA.[12] Exonukleázová doména obsahuje své vlastní katalytické místo a odstraňuje spárované báze. Mezi sedmi různými rodinami DNA polymeráz je „palmová doména“ zachována v pěti z těchto rodin. „Prstová doména“ a „doména palce“ nejsou konzistentní v každé rodině kvůli různým prvkům sekundární struktury z různých sekvencí.[11]
Funkce
Pol I má čtyři enzymatické aktivity:
- A 5'→3' (dopředu) aktivita DNA polymerázy závislá na DNA, vyžadující 3 ' primer web a řetězec šablony
- A 3 '→ 5' (vzad) exonukleáza činnost, která zprostředkovává korektura
- Zprostředkování aktivity exonukleázy 5 '→ 3' (dopředu) překlad nicku v době Oprava DNA.
- Aktivita DNA polymerázy závislá na 5 '→ 3' (dopředné) RNA. Pol I pracuje na šablonách RNA se značně nižší účinností (0,1–0,4%) než na šablonách DNA a tato aktivita má pravděpodobně jen omezený biologický význam.[13]
Aby se zjistilo, zda se Pol I primárně používá k replikaci DNA nebo k opravě poškození DNA, byl proveden experiment s deficitním Pol I mutantním kmenem E-coli. Mutantní kmen, kterému chyběl Pol I, byl izolován a ošetřen mutagenem. U mutantního kmene se vyvinuly bakteriální kolonie, které pokračovaly v normálním růstu a také jim chyběl Pol I. To potvrdilo, že Pol I nebyl pro replikaci DNA vyžadován. Mutantní kmen však také vykazoval charakteristiky, které zahrnovaly extrémní citlivost na určité faktory, které poškozovaly DNA, podobně UV světlo. To tedy znovu potvrdilo, že Pol I se pravděpodobněji bude podílet na opravě poškození DNA než na replikaci DNA.[6]
Mechanismus
V procesu replikace RNáza H odstraní RNA primer (vytvořil primase ) z zaostávající vlákno a poté polymeráza I vyplní potřebné nukleotidy mezi Okazaki fragmenty (vidět replikace DNA ) ve směru 5 '→ 3', korektury chyb. Je to enzym závislý na templátu - přidává pouze nukleotidy správně základní pár s existujícím řetězcem DNA působícím jako templát. Je zásadní, aby tyto nukleotidy byly ve správné orientaci a geometrii k párování bází s řetězcem DNA templátu tak DNA ligáza mohou spojit různé fragmenty dohromady do souvislého vlákna DNA. Studie polymerázy Potvrdil jsem, že různé dNTP se mohou vázat na stejné aktivní místo na polymeráze I. Polymeráza I je schopna aktivně rozlišovat mezi různými dNTP pouze poté, co podstoupí konformační změna. Jakmile k této změně došlo, Pol I zkontroluje správnou geometrii a správné zarovnání páru bází, vytvořeného mezi vázaným dNTP a odpovídající bází na pramenu šablony. Správná geometrie párů bází A = T a G≡C je jediná, která se vejde do Aktivní stránky. Je však důležité vědět, že každý desátý4 do 105 nukleotidy jsou přidány nesprávně. Pol I však může tuto chybu v replikaci DNA opravit pomocí své selektivní metody aktivní diskriminace.[5]
Přes jeho časnou charakterizaci se rychle ukázalo, že polymeráza I není enzym odpovědný za většinu syntézy DNA - replikace DNA v E-coli probíhá rychlostí přibližně 1 000 nukleotidů za sekundu, zatímco rychlost syntézy párů bází polymerázou I je v průměru pouze mezi 10 a 20 nukleotidy za sekundu. Navíc jeho buněčné množství přibližně 400 molekul na buňku nekorelovalo se skutečností, že jsou obvykle jen dva replikační vidlice v E-coli. Navíc to není dostatečné procesivní zkopírovat celý genom, protože odpadá po začlenění pouze 25–50 nukleotidy. Jeho role v replikaci byla prokázána, když v roce 1969 John Cairns izoloval životaschopnou polymerázu I mutant které postrádaly aktivitu polymerázy.[14] Cairnsova laboratorní asistentka, Paula De Lucia, vytvořila tisíce bezbuněčných výtažků z E-coli kolonií a testoval je na aktivitu DNA-polymerázy. 3 478. klon obsahoval polA mutant, kterého Cairns pojmenoval pro připisování „Paule“ [De Lucia].[15] To nebylo až do objevu DNA polymeráza III že byla nakonec identifikována hlavní replikativní DNA polymeráza.
Výzkumné aplikace

DNA polymeráza I získaná z E-coli se hojně používá pro molekulární biologie výzkum. Avšak aktivita 5 '→ 3' exonukleázy ji činí nevhodnou pro mnoho aplikací. Tuto nežádoucí enzymatickou aktivitu lze jednoduše odstranit z holoenzymu a zanechat užitečnou molekulu zvanou Klenowův fragment, široce používaný v molekulární biologie. Ve skutečnosti byl Klenowův fragment použit během prvních protokolů z polymerázová řetězová reakce (PCR) amplifikace do Thermus aquaticus, zdroj tepelně tolerantní Taq Polymeráza Byl jsem objeven v roce 1976.[17] Vystavení DNA polymerázy I proteáze subtilisin štěpí molekulu na menší fragment, který si zachovává pouze aktivity DNA polymerázy a korektury.
Viz také
Reference
- ^ Lehman IR, Bessman MJ, Simms ES, Kornberg A (červenec 1958). „Enzymatická syntéza deoxyribonukleové kyseliny. I. Příprava substrátů a částečné čištění enzymu z Escherichia coli". The Journal of Biological Chemistry. 233 (1): 163–70. PMID 13563462.
- ^ Voet D, Voet JG, Pratt CW (1999). Základy biochemie. New York: Wiley.[stránka potřebná ]
- ^ Lehman IR (září 2003). „Objev DNA polymerázy“. The Journal of Biological Chemistry. 278 (37): 34733–8. doi:10,1074 / jbc.X300002200. PMID 12791679.
- ^ „Nobelova cena za fyziologii nebo medicínu 1959“. www.nobelprize.org. Citováno 2016-11-08.
- ^ A b C Cox MM, Doudna J (2015). Molekulární biologie (2. vyd.). New York: W.H. Freemane.[stránka potřebná ]
- ^ A b C Cooper, Geoffrey M. Geoffrey (01.01.2000). "Replikace DNA". Citovat deník vyžaduje
| deník =(Pomoc) - ^ Wood RD, Shivji MK (duben 1997). „Které DNA polymerázy se používají pro opravu DNA u eukaryot?“. Karcinogeneze. 18 (4): 605–10. doi:10.1093 / carcin / 18.4.605. PMID 9111189.
- ^ Biertümpfel C, Zhao Y, Kondo Y, Ramón-Maiques S, Gregory M, Lee JY, Masutani C, Lehmann AR, Hanaoka F, Yang W (červen 2010). "Struktura a mechanismus lidské DNA polymerázy eta". Příroda. 465 (7301): 1044–8. Bibcode:2010Natur.465.1044B. doi:10.1038 / nature09196. PMC 2899710. PMID 20577208.
- ^ Hübscher U, Spadari S, Villani G, Maga G (2010). DNA polymerázy. doi:10.1142/7667. ISBN 978-981-4299-16-9.[stránka potřebná ]
- ^ „DNA Polymerase I: Enzymatic Reactions“.
- ^ A b „MBIO.4.14.5“. bioscience.jbpub.com. Citováno 2017-05-14.
- ^ Loeb LA, Monnat RJ (srpen 2008). "DNA polymerázy a lidská nemoc". Genetika hodnocení přírody. 9 (8): 594–604. doi:10.1038 / nrg2345. PMID 18626473.
- ^ Ricchetti M, Buc H (únor 1993). "E-coli DNA polymeráza I jako reverzní transkriptáza ". Časopis EMBO. 12 (2): 387–96. PMC 413221. PMID 7679988.
- ^ De Lucia P, Cairns J (prosinec 1969). "Izolace E-coli kmen s mutací ovlivňující DNA polymerázu ". Příroda. 224 (5225): 1164–6. Bibcode:1969 Natur.224.1164D. doi:10.1038 / 2241164a0. PMID 4902142.
- ^ Friedberg EC (únor 2006). „Enzym heuréka: objev DNA polymerázy“. Nature Reviews Molecular Cell Biology. 7 (2): 143–7. doi:10.1038 / nrm1787. PMID 16493419.
- ^ EMBL-EBI. „EMBL Evropský bioinformatický institut“. www.ebi.ac.uk. Citováno 2016-11-08.
- ^ van Pelt-Verkuil E, van Belkum A, Hays JP (2008). "Taq a další termostabilní DNA polymerázy". Principy a technické aspekty amplifikace PCR. 103–18. doi:10.1007/978-1-4020-6241-4_7. ISBN 978-1-4020-6240-7.
