POLD1 - POLD1
Gen polymerázy delta 1 (POLD1) kóduje velkou katalytickou podjednotku POLD1 / p125 Delta DNA polymerázy (Polδ) komplex.[5][6] Polδ enzym je zodpovědný za syntézu zaostávající vlákno DNA a podílí se také na některých činnostech v přední vlákno (Obrázek 1). Podjednotka POLD1 / p125 kóduje DNA polymerující i exonukleáza domény, které poskytují proteinu důležitou druhou funkci v korekturách zajišťujících přesnost replikace během syntézy DNA, a v řadě typů spojených s replikací Oprava DNA po poškození DNA. Germinální mutace zhoršující aktivitu POLD1 podílejí se na několika typech dědičných nádorových onemocnění, na některých sporadických rakovinách a na vývojovém syndromu předčasného stárnutí, Dolní čelist hypoplázie, Hluchota a Progeroid funkce a Lipodystrofie (MDPL /MDP syndrom ). Studie POLD1 zdůrazňují důležitost udržování genomické stability na hranici tumorigeneze. V současné době není jasné, zda je zvýšená tumorigeneze spojena s POLD1 defekty jsou výsledkem zvýšených substitucí základny nebo kvůli zhroucení vidlice a výroba DNA dvouvláknové zlomy (DSB).[6][7] Nedávné recenze se zabývaly důležitými funkcemi POLD1 a Polδ.[6][7]
Objev
První DNA polymeráza, DNA polymeráza I, byl objeven uživatelem Arthur Kornberg a jeho kolegové v roce 1956,[8] zkontrolováno v.[9] V roce 1976 Byrnes et al. objevili třetí aktivitu DNA polymerázy v buňkách savců, která se nazývala delta polymerázy (δ).[10] Byl purifikován z králičího erytroidu hyperplastické kostní dřeň a popsána jako DNA polymeráza, která má vlastní 3 'až 5' exonukleázovou aktivitu. Funkce korektury exonukleáz 3—5 ‘pro DNA polymerázy (E-coli ) poprvé popsali Kornberg a Brutlag před 4 lety,[11] zkontrolováno v.[12] Lidská DNA Polδ je a heterotetramer. Čtyři podjednotky jsou: (POLD1 / p125), (POLD3/ p66), (POLD2/ p50) a (POLD4/ p12), přičemž alternativní názvy odrážejí molekulové hmotnosti vyjádřené v kilodaltonů (kDa). Polymerázová katalytická podjednotka byla identifikována jako 125 kDa polypeptid aktivním barvením v roce 1991.[13] Několik skupin nezávisle klonovalo lidské a myší cDNA POLD1.[5][14][15] Po jeho čištění z různých zdrojů, včetně brzlíku, lidské placenty a buněk HeLa,[16][17][18][19][20] jeho aktivita byla zapojena do opravy DNA.[21][22]
Gen
Polymeráza (DNA) delta 1, katalytická podjednotka a POLD1 jsou název a symbol genu schválené Výborem pro genovou nomenklaturu (HGNC) organizace Human Genome Organization (HUGO).[23] POLD1 je také známý jako CDC2, MDPL, POLD, a CRCS10), je ~ 34 kb dlouhý a jeho cytogenetická poloha je chromozom 19[24] q13,33.[25] Přesné umístění v sestavě GRCh38.p2 je od páru bází 50 384 290 do páru bází 50 418 018 na chromozomu 19.[26] Myší ortolog se mapuje na myší chromozom 7.[27] U lidí obsahuje hlavní transkript POLD1 (NM_002691.3) 27 exonů a překládá se do 1107 aminokyselin podjednotky p125 nebo A. Byla hlášena delší izoforma s vložením 26 aminokyselin do rámce po aminokyselině 592 (NP_001295561.1). A pseudogen (LOC100422453) byla hlášena na dlouhém rameni chromozomu 6.[26] Tabulka 1 uvádí názvy genů a chromozomální umístění různých podjednotek Polδ u lidí, myší, začínajících kvasinek (S. cerevisiae ) a štěpné droždí (S. pombe ).
The POLD1 promotor genů je regulován prostřednictvím aparátu buněčného cyklu a exprese mRNA POLD1 dosáhne vrcholu v pozdní fázi G1 / S během replikace DNA.[28] Promotér POLD1 je bohatý na G / C a nemá žádné TATA box. Transkripce tohoto promotoru obsahujícího GC box je regulována Sp1 a transkripční faktory související se Sp1, jako je Sp3, s jejich vazbou zprostředkovanou opakovacími vazebnými sekvencemi 11 bp.[29][30] The POLD1 promotor obsahuje E2F -jako sekvence umístěná poblíž majora místo zahájení transkripce.[30] Další regulační prvek, prvek buněčného cyklu / oblast homologie genů buněčného cyklu (CDE / CHR), umístěný za startovacím místem, je důležitý pro POLD1 přepis v G2 / M postupně E2F1 a p21 bílkoviny.[31][32] P53 reguluje POLD1 transkripce nepřímou aktivací závislou na p21 dráhy p53-p21-DREAM-CDE / CHR.[33] Jedna studie uvádí, že nádorový supresorový protein p53 soutěží se Sp1 o vazbu na POLD1 promotér.[29] A mikroRNA (miR), miR-155, downreguluje POLD1 nepřímo potlačením transkripčního faktoru FOXO3a,[34] který má domnělá vazebná místa v POLD1 promotor (RTMAAYA; prvek odpovědi).[35]
Protein
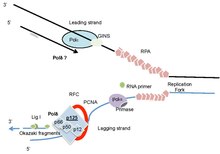
POLD1 / p125 má společný záhyb rodiny B, podobně jako jiné DNA polymerázy (Polα a ε).[37] Lidský POLD1 / p125 má domnělý údaj signál jaderné lokalizace na N-terminál konec (zbytky 4-19).[24] Zbytky 304-533 obsahují exonukleázovou doménu (obrázek 2), zatímco zbytky 579-974 obsahují polymerázovou doménu. Exonukleázová doména je doména typu DnaQ typu DnaQ společná pro B-DNA polymeráza rodina.[38] Tato doména má vlásenka beta struktura, která pomáhá při přepínání mezi aktivními místy polymerázy a exonukleázy v případě nesprávné inkorporace nukleotidů.
Motivy A a C, které jsou nejkonzervovanější z polymerázové domény. Ty mají 2 katalytické aspartáty, v motivu A (DXXLYPS, D602) a motivu C (DTDS, D757), které váží vápník v aktivním místě. Motiv A má 11 aminokyselin, které jsou důležité pro inkorporaci a tvorbu nukleotidů fosfodiesterová vazba.
Tyrosin Y701 funguje podobně jako tyrosin Y567 v RB69 bakteriofág ortolog jako cukrová sterická brána, která brání začlenění ribonukleotidů.[39] Motiv LXCXE (711 až 715) zprostředkovává vazbu na pRB Během Fáze G1 buněčného cyklu.[40] Polymerázová doména má také vysoce konzervativní motiv KKRY (zbytky 806 až 809), který je důležitý pro vazebnou a katalytickou funkci.[41] POLD1 lze cílit na jádro po okyselení pomocí motivu nukleolární zadržovací sekvence (NoDS) představovaného malými sekvenčními motivy rozptýlenými po celé oblasti kódující protein.[42][43][44] The C-terminál doména má dva konzervované cystein - potřebné motivy pro vázání kovů (CysA a CysB) (od 1012 do 1083) Proliferující buněčný jaderný antigen (PCNA) vázání a nábor doplňkových podjednotek.[45] CysB koordinuje klastr [4Fe-4S] přidaný prostřednictvím Cytosolic Iron-Sulphine Protein Assembly (CIA), který vyžaduje funkci mitochondriálního montážního aparátu Iron Sulphur Cluster (ISC).[46] Proces zrání je zprostředkován jádrem zaměřeným na komplex CIA1-CIA2B / FAM96B-MMS19, který interaguje s apoproteinem, aby zajistil specifickou inzerci klastru Fe-S.[47][48]

Vazebné a asociační studie ukázaly, že POLD2 je úzce spojen s POLD1; POLD3 a POLD2 interagují navzájem a POLD4 interagují s POLD1 i POLD2.[51][52] Heterotetramer Polδ rekonstituovaný koexpresí podjednotek v buňkách Sf9 měl vlastnosti podobné jako Polδ purifikovaný z brzlíku tele a kompletní holoenzym byl velmi silně stimulován PCNA.[53] Četné studie ukázaly, že zatímco POLD1 má jak aktivitu polymerázy, tak 3'-5 ‘exonukleázovou korekturu, ostatní podjednotky zvyšují tyto aktivity, schopnosti vázat DNA a funkčně důležité interakce s PCNA a jejím upínacím zavaděčem Faktor replikace C. (RFC). O holoenzymu DNA Polδ se často uvažuje tak, že zahrnuje PCNA a RFC a také čtyři podjednotky komplexu polymerázy (obrázek 1).
Řada dalších studií a šetření identifikovala další interakční partnery související s funkcemi replikace a opravy DNA. Obrázek 3 ukazuje matici zavedených a domnělých interakcí během replikace a opravy, ke které lze dále přistupovat[54] a.[55] Webové stránky na Vanderbiltově univerzitě poskytují další interakci s důležitou proteinovou strukturou POLD1 a různými třídami interakcí genů a proteinů na základě kritérií, jako je společný výskyt v komplexu, přímá fyzická interakce, regulační vztah a společná exprese.[56]
| Polymeráza delta podjednotky | Název proteinu v člověku | Homo sapiens | Mus musculus | Saccharomyces cerevisiae | Schizosaccharomyces pombe |
|---|---|---|---|---|---|
| A (katalytický) | p125 | POLD1-Chr 19q13.3 | Pold1-Chr 7B4 | POL3-Chr IV | cdc6-Chr II |
| B (příslušenství) | p50 | POLD2-Chr 7p13 | Pold2-Chr 11A2 | POL31-Chr X | cdc1-Chr I |
| C (příslušenství) | p66 | POLD3-Chr 11q14 | Pold3-Chr 7F1 | POL32-Chr X | cdc27-Chr II |
| D (příslušenství) | p12 | POLD4-Chr 11q13 | Pold4-Chr 19A | - | cdm1-Chr II |
| Tabulka 1: Genové názvy a umístění chromozomů pro různé podjednotky delta polymerázy v lidských, myších, nadějných a štěpných kvasinkách. | |||||
Vyjádření a regulace

Protein POLD1 / P125 je všudypřítomně exprimován v panelu lidských tkání s vysokými hladinami v tkáních srdce a plic.[60] Subcelulární lokalizace POLD1 / p125 je převážně v jádro a nukleoplazma.[61]
Snížení POLD1 / p125 bylo pozorováno u stárnoucí lidské kožní fibroblasty a v lymfocytech od starší populace.[62][63] Výraz POLD1 / p125 je epigeneticky regulováno v reakci na poškození DNA.[64] Další studie také ukázaly, že exprese POLD1 / p125 je regulována miR-155,[34] p53[29] a podle dlouhá nekódující RNA, PVT1.[65] V přítomnosti poškození DNA nebo replikačního stresu (UV světlo, methylmethansulfonát, hydroxymočovina nebo afidikolin ), podjednotka POLD4 / p12 se rychle degraduje. Katalytické aktivity p125 se liší, ať už je to v heterotetrameru (Polδ4, s p12[66][67]) nebo v heterotrimeru (Polδ3, bez p12).[68] Produkce heterotrimeru závisí na degradaci p12 působením E3 ligáza RNF8, protein zapojený do opravy DSB a možná homologní rekombinace (HR).[69] Kromě toho E3 ligáza CRL4Cdt2 může degradovat POLD4 / p12 během normální replikace DNA a v přítomnosti poškození DNA.[70] POLD4 / p12 může být také degradován proteáza µ-calpain, který se podílí na aktivaci vápníku apoptóza.[71][72]
POLD1 / p125 má doménu NoDS, která reguluje transport do nukleolu v reakci na acidózu.[44] Nukleolární transport vyžaduje přímou interakci mezi podjednotkou p50 a WRN protein.[73] Během reakce na poškození DNA se WRN pohybuje ven z nukleolu a uvolňuje tak Polδ.[74][75] Ukázalo se také, že POLD1 / p125 interaguje s PDIP46 / SKAR[76] a LMO2.[77][78]
Funkce
replikace DNA
Replikace DNA je vysoce organizovaný proces, který zahrnuje mnoho enzymů a proteinů, včetně několika DNA polymeráz. Hlavní replikativní aktivita v S fáze buněčného cyklu závisí na třech DNA polymerázách - Polymeráza alfa (Polα), delta polymerázy (Polδ) a Polymeráza epsilon (Polε). Po zahájení syntézy DNA Polα, Polδ nebo Polε provedou syntézu zpoždění a vedoucí řetězce.[79] Tyto polymerázy udržují velmi vysokou věrnost, kterou zajišťuje Watson-Crickovo párování základen a aktivita 3'-exonukleázy (nebo korektury).[80] Nedávné studie tvrdí, že Polδ může syntetizovat hlavní řetězec.[80][81][82][83][84] Jak tyto polymerázy fungují ve vztahu k dalším faktorům podílejícím se na replikaci, je velmi zajímavé, protože to pravděpodobně vysvětluje mutační prostředí, které produkují, když jsou defektní. Udržování věrnosti replikace je jemná rovnováha mezi jedinečnými chybami polymerázami δ a ε,[85] rovnováha mezi korekturami a MMR a rozdíl ve zpracování ribonukleotidů mezi těmito dvěma řetězci.[36] Rozsáhlé studie na modelech kvasinek ukázaly, že mutace v doméně exonukleáz homologů Polδ a Polε mohou způsobit fenotyp mutátoru, přezkoumáno v.[86] Jednovláknová (ss) DNA syntetizovaná během syntézy zaostávajícího vlákna může být cílena látkami poškozujícími ss-DNA a je také selektivním cílem pro APOBEC mutace.[87] Proteiny vázající DNA, které rychle reasociují post-replikaci, zabraňují Polδ v opravách chyb produkovaných Polα ve zralém zaostávajícím řetězci.[88] Studie kvasinek ukázaly, že Polδ dokáže korigovat chyby Polε na vedoucím řetězci.[89]
Oprava DNA
Aktivita POLD1 přispívá k více evolučně konzervované Procesy opravy DNA, včetně Neshoda opravy (MMR), Translesová syntéza (TLS), Oprava základní excize (BER), Oprava nukleotidové excize (NER) a dvouvláknový zlom (DSB) oprava.[6] POLD1 zprostředkovává kroky po řezu v BER, NER a MMR.[6] Polδ interaguje s MMR aparátem na podporu korektury po replikaci nově syntetizované DNA,[90] s buňkami nesoucími mutace, které inaktivují složky POLD1 a MMR zažívající zvýšené rychlosti mutací.[91][92] Jak je uvedeno výše, heterotrimer Polδ (Polδ3) se stává dominantní oligomerní formou POLD1 a je aktivní během přítomnosti poškození DNA. Polδ3 je méně náchylný k chybám než (Polδ4) a může lépe rozlišovat mezi neodpovídajícími páry, spojené s lepší aktivitou korektury: má však sníženou schopnost obejít některé základní léze.[74][93] Místo toho přechází polymeráza Polδ na specializovanou polymeráza zeta (Polζ) je důležitý pro TLS, protože substituce p125 za katalytickou podjednotku Polζ, p353, umožňuje lepší bypass aktivitu.[6] V tomto procesu vysoce konzervovaná C-koncová doména (CTD) POLD1 / p125 interaguje s CTD doménou Polζ a železné shluky v každé CTD zprostředkovávají interakce zahrnující vazbu na POLD2, které umožňují přepínání polymerázy během TLS.[94] Některé nedávné studie naznačují, že přechod z Polδ na Pol lambda (λ) také podporuje TLS a opravu poškození oxidační DNA jako 7,8-dihydro-8-oxoguanin léze.[95]
Vyčerpání POLD1 může zastavit buněčný cyklus ve fázích G1 a G2 / M v lidských buňkách.[96] Blokování buněčného cyklu v těchto fázích obvykle indikuje přítomnost poškození DNA a aktivaci kontrolních bodů poškození DNA. POLD1 vyčerpané buňky jsou citlivé na inhibici kináz kontrolního bodu poškození DNA ATR a CHK1.[97] v S. pombe, HR mechanismy by mohly restartovat zastavené replikační vidlice využitím aktivity syntézy řetězců Polδ, ale takový neallelický HR zprostředkovaný restart je velmi náchylný k chybám, což potenciálně vede ke zvýšené genomové nestabilitě.[98] Polδ strukturálně a funkčně interaguje s proteinem WRN a WRN rekrutuje Polδ do jádra.[73] The WRN gen je mutován Wernerův syndrom (an autosomální recesivní porucha) vedoucí ke zrychlenému stárnutí a zvýšené genetické nestabilitě. Interakce s WRN zvyšuje procesivitu Polδ způsobem nezávislým na PCNA.[99] Prostřednictvím těchto interakcí WRN přímo ovlivňuje replikaci a opravu DNA a pomáhá při syntéze zprostředkované Polδ.
Klinický význam
Rakovina
Ukázalo se, že proteiny pro opravu DNA jsou důležité u lidských onemocnění včetně rakoviny. Například zárodečné mutace v DNA opravných proteinech zapojených do MMR (MSH2, MLH1, MSH6 a PMS2) byly popsány v Lynchův syndrom (LS), který je charakterizován přítomností nestabilita mikrosatelitu (MSI).[100] Nověji byly zárodečné mutace hlášeny v doménách exonukleázy POLD1 a PÓL, katalytická podjednotka Polε. Tyto mutace jsou spojeny s oligoadenomatózní polypózou s časným nástupem kolorektální karcinom (CRC), rakovina endometria (EDMC), rakovina prsu, a mozkové nádory.([101][102][103][104][105] zkontrolováno v[7]) Většina z hlášených POLD1 mutace spojené s rakovinou jsou přítomny v doméně exonukleázy.[7][101][102][106][107][108] Na rozdíl od LS POLD1 mutované nádory jsou mikrosatelitní stabilní. Některá data naznačují myšlenku, že POLD1 nádory jsou spojeny s řidičskými mutacemi v genech včetně APC a KRAS.[101] The POLD1 missense mutace str. S478N v doméně exonukleázy byl ověřen jako škodlivý a patogenní.[101] jiný POLD1 klinicky byly identifikovány varianty, u nichž se předpokládá, že budou škodlivé, a jsou v současné době předmětem dalšího zkoumání (např. s. D316H, s. D316G, s. R409W, s. L474P a s. P327L).[102][103][104]
U pediatrických pacientů došlo u dvojitě zasažených mutací POLD1 nebo PÓL a nedostatek opravy bialelického nesouladu (bMMRD), vede k ultrahypermutovaným fenotypům nádoru.[109][110][111] Takové fenotypy jako ultrahypermutace v nádorech mohou naznačovat lepší reakci na vyvíjející se novější protinádorová léčiva, i když to vyžaduje přímé hodnocení pro POLD1.[112][113][114][115][116][117] Bouffet a kol. nahlásit dva sourozence pomocí bMMRD- glioblastoma multiforme kteří mají somatické mutace v PÓL (P436H v jednom, S461P v druhém) a vykazovaly trvalou odpověď na klinickou studii s anti-programovaná smrt-1 inhibitor nivolumab. POLD1 mutace byly studovány v buněčných liniích[118][119][120][121] a modely myší. Například a homozygotní Mutace Polδ u myší, která narušuje enzymatické funkce, vede k vysoce zvýšené incidenci rakoviny.[122]
MDPL
Poškozující mutace v POLD1 byly také pozorovány u pacientů se syndromem známým jako mandibulární hypoplázie, hluchota a progeroidní rysy se syndromem lipodystrofie (MDPL / MDP) (# 615381 v databázi OMIM (Online Mendelian Inheritance in Man)).[60][123][124] Toto je velmi vzácný syndrom a bylo popsáno několik studií popisujících mutace. Pozorované mutace jsou v oblastech, které ovlivňují doménu exonukleázy a domény polymerázy.[60][123] Pět nesouvisejících de novo případy byly popsány stejným způsobem heterozygotní varianta, c.1812_1814delCTC p.Ser605del (rs398122386). S605 je ve vysoce konzervativním motivu A aktivního místa polymerázy. Tato varianta neinhibuje aktivitu vázání DNA, ale ovlivňuje katalýzu. Další varianta byla hlášena u samostatného pacienta (p.R507C).[123] Tato varianta se nachází ve vysoce konzervované doméně ExoIII a dosud nebyla zcela charakterizována.
Varianty POLD1 Ser605del a R507C byly také identifikovány u podskupiny pacientů s atypickým Wernerovým syndromem (AWS). Po molekulárním testování byli tito pacienti reklasifikováni jako pacienti s MDPL / MDP. MDPL / MDP, AWS a Wernerův syndrom progeria.[125] První příklad přenosu zárodečné linie byl pozorován u matky a syna s mutací Ser605del.[126] Nedávno dvě nezávislé studie identifikovaly pacienty se stejnou homozygotní variantou sestřihu v POLE1, katalytická podjednotka Polε. Jeden s fenotypem obličejový dysmorfismus, imunodeficience, liveso a nízký vzrůst (také známý jako FILS syndrom).[127] Druhý způsobil závažnější příznaky.[128] Tyto případy se připojují k rostoucímu počtu vývojových vad spojených s dědičnými mutacemi zaměřenými na funkci polymerázových genů.
Věková závislá downregulace POLD1 bylo pozorováno.[63] ačkoli dosud nebyl s tímto fenotypem spojen žádný klinický význam. Probíhají také studie, jejichž cílem je pochopit, zda existuje vztah mezi těmito patologiemi nebo těmito mutacemi a predispozicí k rakovině. V současné době navrhované mechanismy, kterými jsou defekty POLD1 patogenní, se zaměřují na myšlenku replikačních defektů vedoucích k genomové nestabilitě a aktivaci kontrolního bodu, což v konečném důsledku vede k buněčné smrti nebo stárnutí buněk. Alternativně je Polδ spojen s lamináty a jaderný obal během zadržení G1 / S nebo rané fáze S; mutace v laminách způsobují lipodystrofie související s jadernou obálkou s fenotypy podobnými MDPL / MDP a Wernerovým syndromem.[129]
Hodnocení rizika rakoviny a komerční testování
Dědičné rakoviny tlustého střeva a konečníku (CRC) spojené s mutacemi schopnosti korektury POLD1 a PÓL jsou někdy označovány jako „korektury spojené s polymerázou polypóza “(PPAP), (ačkoli byla identifikována alespoň jedna studie POLD1 mutace spojené s nepolypózním CRC).[101][102][104][106][107] POLD1 mutace byly také spojeny se zvýšenou predispozicí k rakovině rakovina endometria.[101][104][105] Nedávná studie navrhla pokyny pro genetické testování na POLD1 mutace, které zahrnují: 1) Výskyt 20–100 adenomů a 2) Rodinná anamnéza, která splňuje kritéria Amsterdam II pro rakovinu kolorektálního karcinomu a endometria.[103] Aktuální pokyny pro klinické testování pro rodiny s mutacemi v POLD1 / PÓL zahrnout kolonoskopie (každé 1–2 roky), gastroduodenoskopie (každé 3 roky) začínající brzy (20–25), možnost mozkové nádory a screening rakoviny endometria (začátek u 40 u ženských nosiček).[103] V současné době probíhají studie k určení přesného rizika rakoviny z konkrétních POLD1 mutace. Aktuální data naznačují, že mutace v tomto genu jsou vysoce penetrant. Další nedávná studie ukázala, že mutace ovlivňující mutace Polδ a Polε se mohou vyskytovat společně s mutacemi MMR.[110] To naznačuje, že testování panelových genů by mělo zahrnovat geny MMR a Pol iu pacientů s MSI.
Existuje několik možností komerčního diagnostického testování mutací v POLD1.[130] Genetické testování obvykle zahrnuje POLD1 kódující exony (26) a alespoň 20 bází do sousedních nekódujících oblastí. Pro rodiny se známými mutacemi je k dispozici také testování na jednom místě k potvrzení přítomnosti mutace.[130] Dostupnost těchto genetických testů otevřela nové možnosti pro rakoviny dříve klasifikované jako geneticky nedefinované kolorektální karcinomy nebo kolorektální karcinom typu „X“.[105] Rovněž byly vyvinuty zdroje pro klinické testování MDPL / MDP.[131]
Poznámky
Verze tohoto článku z roku 2016 byla aktualizována externím odborníkem na základě modelu dvojího publikace. Korespondence akademický recenzent článek byl publikován v Gen a lze jej citovat jako: Emmanuelle Nicolas; Erica Golemis; Sanjeevani Arora (16. června 2016), „POLD1: Ústřední mediátor replikace a opravy DNA a implikace pro rakovinu a jiné patologické stavy“, Gen, 590 (1): 128–141, doi:10.1016 / J.GENE.2016.06.031, ISSN 0378-1119, PMC 4969162, PMID 27320729, Wikidata Q38870074 |
Reference
- ^ A b C GRCh38: Vydání souboru 89: ENSG00000062822 - Ensembl, Květen 2017
- ^ A b C GRCm38: Vydání souboru 89: ENSMUSG00000038644 - Ensembl, Květen 2017
- ^ „Human PubMed Reference:“. Národní centrum pro biotechnologické informace, Americká národní lékařská knihovna.
- ^ „Myš PubMed Reference:“. Národní centrum pro biotechnologické informace, Americká národní lékařská knihovna.
- ^ A b Chung DW, Zhang JA, Tan CK, Davie EW, So AG, Downey KM (prosinec 1991). "Primární struktura katalytické podjednotky delta lidské DNA polymerázy a chromozomální umístění genu". Sborník Národní akademie věd Spojených států amerických. 88 (24): 11197–201. Bibcode:1991PNAS ... 8811197C. doi:10.1073 / pnas.88.24.11197. PMC 53101. PMID 1722322.
- ^ A b C d E F Prindle MJ, Loeb LA (prosinec 2012). „Delta DNA polymerázy v replikaci DNA a udržování genomu“. Environmentální a molekulární mutageneze. 53 (9): 666–82. doi:10,1002 / em.21745. PMC 3694620. PMID 23065663.
- ^ A b C d Rayner E, van Gool IC, Palles C, Kearsey SE, Bosse T, Tomlinson I, Church DN (leden 2016). „Soubor chyb: mutace doménové korekce polymerázy v rakovině“. Recenze přírody. Rakovina. 16 (2): 71–81. doi:10.1038 / nrc.2015.12. PMID 26822575. S2CID 9359891.
- ^ Kornberg A, Kornberg SR, Simms ES (duben 1956). "Syntéza metafosfátu enzymem z Escherichia coli". Biochimica et Biophysica Acta. 20 (1): 215–27. doi:10.1016/0006-3002(56)90280-3. PMID 13315368.
- ^ Friedberg EC (únor 2006). „Enzym heuréka: objev DNA polymerázy“. Nature Reviews Molecular Cell Biology. 7 (2): 143–7. doi:10.1038 / nrm1787. PMID 16493419. S2CID 39605644.
- ^ Byrnes JJ, Downey KM, Black VL, So AG (červen 1976). "Nová savčí DNA polymeráza s aktivitou 3 'až 5' exonukleázy: delta DNA polymerázy". Biochemie. 15 (13): 2817–23. doi:10.1021 / bi00658a018. PMID 949478.
- ^ "Enzymatická syntéza deoxyribonukleové kyseliny. XXXVI. Funkce korektury aktivity 3 5 'exonukleázy v polymeráze deoxyribonukleové kyseliny". ResearchGate. Citováno 2016-04-25.
- ^ Reha-Krantz LJ (květen 2010). „Korektury DNA polymerázy: Více rolí udržuje stabilitu genomu“. Biochimica et Biophysica Acta (BBA) - bílkoviny a proteomika. 1804 (5): 1049–63. doi:10.1016 / j.bbapap.2009.06.012. PMID 19545649.
- ^ Lee MY, Jiang YQ, Zhang SJ, Toomey NL (únor 1991). "Charakterizace delty lidské DNA polymerázy a její imunochemické vztahy s DNA polymerázou alfa a epsilon". The Journal of Biological Chemistry. 266 (4): 2423–9. PMID 1703528.
- ^ Yang CL, Chang LS, Zhang P, Hao H, Zhu L, Toomey NL, Lee MY (únor 1992). "Molekulární klonování cDNA pro katalytickou podjednotku delta lidské DNA polymerázy". Výzkum nukleových kyselin. 20 (4): 735–45. doi:10.1093 / nar / 20.4.735. PMC 312012. PMID 1542570.
- ^ Cullmann G, Hindges R, Berchtold MW, Hübscher U (prosinec 1993). "Klonování myší cDNA kódující delta polymerázu DNA: upřesnění homologických polí". Gen. 134 (2): 191–200. doi:10.1016 / 0378-1119 (93) 90093-i. PMID 8262377.
- ^ Lee MY, Tan CK, So AG, Downey KM (květen 1980). „Čištění delty polymerázy deoxyribonukleové kyseliny delta z brzlíku tele: částečná charakterizace fyzikálních vlastností“. Biochemie. 19 (10): 2096–101. doi:10.1021 / bi00551a015. PMID 7378348.
- ^ Lee MY, Tan CK, Downey KM, So AG (duben 1984). "Další studie na deltě DNA DNA telecího brzlíku purifikované do homogenity novým postupem". Biochemie. 23 (9): 1906–13. doi:10.1021 / bi00304a003. PMID 6426510.
- ^ Crute JJ, Wahl AF, Bambara RA (leden 1986). „Čištění a charakterizace dvou nových forem delta DNA polymerázy s vysokou molekulovou hmotností“. Biochemie. 25 (1): 26–36. doi:10.1021 / bi00349a005. PMID 3954990.
- ^ Wahl AF, Crute JJ, Sabatino RD, Bodner JB, Marraccino RL, Harwell LW, Lord EM, Bambara RA (prosinec 1986). "Vlastnosti dvou forem delta DNA polymerázy z brzlíku tele". Biochemie. 25 (24): 7821–7. doi:10.1021 / bi00372a006. PMID 3099836.
- ^ Lee MY, Toomey NL (únor 1987). "Lidská placentární DNA polymeráza delta: identifikace 170-kilodaltonového polypeptidu aktivním barvením a imunoblotováním". Biochemie. 26 (4): 1076–85. doi:10.1021 / bi00378a014. PMID 2436659.
- ^ Dresler SL, Kimbro KS (květen 1987). „2 ', 3'-Dideoxythymidin 5'-trifosfátová inhibice replikace DNA a ultrafialová indukovaná syntéza opravy DNA v lidských buňkách: důkazy o zapojení delty DNA polymerázy“. Biochemie. 26 (10): 2664–8. doi:10.1021 / bi00384a002. PMID 3606985.
- ^ Nishida C, Reinhard P, Linn S (leden 1988). „Syntéza opravy DNA v lidských fibroblastech vyžaduje delta DNA polymerázy“. The Journal of Biological Chemistry. 263 (1): 501–10. PMID 3335506.
- ^ "HGNC databáze jmen lidských genů | Výbor pro genovou nomenklaturu HUGO". www.genenames.org. Citováno 2016-04-25.
- ^ A b Chung DW, Zhang JA, Tan CK, Davie EW, So AG, Downey KM (prosinec 1991). "Primární struktura katalytické podjednotky delta lidské DNA polymerázy a chromozomální umístění genu". Sborník Národní akademie věd Spojených států amerických. 88 (24): 11197–201. Bibcode:1991PNAS ... 8811197C. doi:10.1073 / pnas.88.24.11197. PMC 53101. PMID 1722322.
- ^ Kemper RR, Ahn ER, Zhang P, Lee MY, Rabin M (září 1992). "Gen delta lidské DNA polymerázy se mapuje na oblast 19q13.3-q13.4 hybridizací in situ". Genomika. 14 (1): 205–6. doi:10.1016 / s0888-7543 (05) 80311-8. PMID 1427831.
- ^ A b „POLD1 polymeráza (DNA) delta 1, katalytická podjednotka [Homo sapiens (člověk)] - gen - NCBI“. www.ncbi.nlm.nih.gov. Citováno 2016-04-25.
- ^ Goldsby RE, Singh M, Preston BD (leden 1998). "Myší DNA polymerázový delta gen (Pold1) se mapuje na chromozom 7". Savčí genom. 9 (1): 92–3. doi:10,1007 / s003359900693. PMID 9434960. S2CID 42967770.
- ^ Mjelle R, Hegre SA, Aas PA, Slupphaug G, Drabløs F, Saetrom P, Krokan HE (červen 2015). „Regulace buněčného cyklu genů pro opravu lidské DNA a remodelaci chromatinu“. Oprava DNA. 30: 53–67. doi:10.1016 / j.dnarep.2015.03.007. PMID 25881042.
- ^ A b C Li B, Lee MY (srpen 2001). "Transkripční regulace lidského DNA polymerázového delta genu pro katalytickou podjednotku POLD1 supresorem p53 a Sp1". The Journal of Biological Chemistry. 276 (32): 29729–39. doi:10,1074 / jbc.M101167200. PMID 11375983.
- ^ A b Zhao L, Chang LS (únor 1997). "Lidský gen POLD1. Identifikace upstream aktivační sekvence, aktivace pomocí Sp1 a Sp3 a regulace buněčného cyklu". The Journal of Biological Chemistry. 272 (8): 4869–82. doi:10.1074 / jbc.272.8.4869. PMID 9030545.
- ^ Müller GA, Wintsche A, Stangner K, Prohaska SJ, Stadler PF, Engeland K (01.01.2014). „Stránka CHR: definice a identifikace transkripčního prvku buněčného cyklu v celém genomu“. Výzkum nukleových kyselin. 42 (16): 10331–50. doi:10.1093 / nar / gku696. PMC 4176359. PMID 25106871.
- ^ Song N, Zhu X, Shi L, An J, Wu Y, Sang J (červen 2009). "Identifikace a funkční analýza prvku CDE / CHR v promotoru POLD1". Science in China Series C: Life Sciences. 52 (6): 551–9. doi:10.1007 / s11427-009-0077-5. PMID 19557333. S2CID 19278457.
- ^ Fischer M, Quaas M, Steiner L, Engeland K (leden 2016). „Dráha p53-p21-DREAM-CDE / CHR reguluje geny buněčného cyklu G2 / M“. Výzkum nukleových kyselin. 44 (1): 164–74. doi:10.1093 / nar / gkv927. PMC 4705690. PMID 26384566.
- ^ A b Czochor JR, Sulkowski P, Glazer PM (duben 2016). „Nadměrná exprese miR-155 podporuje genomovou nestabilitu snížením výrazu delta polymerázové věty s vysokou věrností a aktivací opravy DSB náchylné k chybám“. Výzkum molekulární rakoviny. 14 (4): 363–73. doi:10.1158 / 1541-7786.MCR-15-0399. PMC 5021065. PMID 26850462.
- ^ Chen X, Ji Z, Webber A, Sharrocks AD (únor 2016). „Studie vazby v celém genomu odhalují mechanismy specificity vazby DNA a funkční souhru mezi transkripčními faktory Forkhead“. Výzkum nukleových kyselin. 44 (4): 1566–78. doi:10.1093 / nar / gkv1120. PMC 4770209. PMID 26578569.
- ^ A b Lujan SA, Williams JS, Kunkel TA (2016-02-01). „Nestabilita eukaryotického genomu ve světle asymetrické replikace DNA“. Kritické recenze v biochemii a molekulární biologii. 51 (1): 43–52. doi:10.3109/10409238.2015.1117055. PMC 4922258. PMID 26822554.
- ^ Doublié S, Zahn KE (01.01.2014). „Strukturální pohledy na replikaci eukaryotické DNA“. Hranice v mikrobiologii. 5: 444. doi:10.3389 / fmicb.2014.00444. PMC 4142720. PMID 25202305.
- ^ „NCBI CDD Conserved Protein Domain DNA_polB_delta_exo“. www.ncbi.nlm.nih.gov. Citováno 2016-04-25.
- ^ Brown JA, Suo Z (únor 2011). „Odemknutí cukru“ sterická brána „DNA polymeráz“. Biochemie. 50 (7): 1135–42. doi:10.1021 / bi101915z. PMC 3040255. PMID 21226515.
- ^ Krucher NA, Zygmunt A, Mazloum N, Tamrakar S, Ludlow JW, Lee MY (listopad 2000). „Interakce proteinu retinoblastomu (pRb) s katalytickou podjednotkou delta DNA polymerázy (p125)“. Onkogen. 19 (48): 5464–70. doi:10.1038 / sj.onc.1203930. PMID 11114723.
- ^ Hogg M, Aller P, Konigsberg W, Wallace SS, Doublié S (leden 2007). „Strukturální a biochemické zkoumání role v korekturách vlásenkové smyčky beta nalezené v doméně exonukleázy replikativní DNA polymerázy rodiny B“. The Journal of Biological Chemistry. 282 (2): 1432–44. doi:10,1074 / jbc.M605675200. PMID 17098747.
- ^ Lam YW, Trinkle-Mulcahy L (01.01.2015). „Nové poznatky o nukleolární struktuře a funkci“. F1000Prime Reports. 7: 48. doi:10.12703 / P7-48. PMC 4447046. PMID 26097721.
- ^ Mekhail K, Rivero-Lopez L, Al-Masri A, Brandon C, Khacho M, Lee S (říjen 2007). "Identifikace společného subnukleárního lokalizačního signálu". Molekulární biologie buňky. 18 (10): 3966–77. doi:10,1091 / mbc.E07-03-0295. PMC 1995723. PMID 17652456.
- ^ A b Audas TE, Jacob MD, Lee S (leden 2012). "Imobilizace proteinů v nukleolu pomocí ribozomální intergenní spacer nekódující RNA". Molekulární buňka. 45 (2): 147–57. doi:10.1016 / j.molcel.2011.12.012. PMID 22284675.
- ^ Netz DJ, Stith CM, Stümpfig M, Köpf G, Vogel D, Genau HM, Stodola JL, Lill R, Burgers PM, Pierik AJ (leden 2012). „Eukaryotické DNA polymerázy vyžadují pro tvorbu aktivních komplexů shluk železa a síry“. Přírodní chemická biologie. 8 (1): 125–32. doi:10.1038 / nchembio.721. PMC 3241888. PMID 22119860.
- ^ Paul VD, Lill R (červen 2015). „Biogeneze cytosolických a jaderných proteinů železo-síra a jejich role ve stabilitě genomu“. Biochimica et Biophysica Acta (BBA) - výzkum molekulárních buněk. 1853 (6): 1528–39. doi:10.1016 / j.bbamcr.2014.12.018. PMID 25583461.
- ^ Gari K, León Ortiz AM, Borel V, Flynn H, Skehel JM, Boulton SJ (červenec 2012). "MMS19 spojuje cytoplazmatické shluky železa a síry s metabolizmem DNA". Věda. 337 (6091): 243–5. Bibcode:2012Sci ... 337..243G. doi:10.1126 / science.1219664. PMID 22678361. S2CID 26605576.
- ^ Stehling O, Vashisht AA, Mascarenhas J, Jonsson ZO, Sharma T, Netz DJ, Pierik AJ, Wohlschlegel JA, Lill R (červenec 2012). „MMS19 sestavuje proteiny železo-síra potřebné pro metabolismus DNA a genomovou integritu“. Věda. 337 (6091): 195–9. Bibcode:2012Sci ... 337..195S. doi:10.1126 / science.1219723. PMC 3420340. PMID 22678362.
- ^ Hansen MF, Johansen J, Bjørnevoll I, Sylvander AE, Steinsbekk KS, Sætrom P, Sandvik AK, Drabløs F, Sjursen W (září 2015). „Nová mutace POLE spojená s rakovinou tlustého střeva, slinivky břišní, vaječníků a tenkého střeva“. Rodinná rakovina. 14 (3): 437–48. doi:10.1007 / s10689-015-9803-2. PMC 4559173. PMID 25860647.
- ^ Zuo Y, poslanec Deutscher (březen 2001). „Superrodiny exoribonukleázy: strukturní analýza a fylogenetická distribuce“. Výzkum nukleových kyselin. 29 (5): 1017–26. doi:10.1093 / nar / 29.5.1017. PMC 56904. PMID 11222749.
- ^ Simon M, Giot L, Faye G (srpen 1991). „Aktivita 3 'až 5' exonukleázy umístěná v podjednotce delta DNA polymerázy Saccharomyces cerevisiae je nutná pro přesnou replikaci.". Časopis EMBO. 10 (8): 2165–70. doi:10.1002 / j.1460-2075.1991.tb07751.x. PMC 452904. PMID 1648480.
- ^ Makarova KS, Krupovic M, Koonin EV (01.01.2014). "Evoluce replikativních DNA polymeráz v archaeách a jejich příspěvky k eukaryotické replikační mašinérii". Hranice v mikrobiologii. 5: 354. doi:10.3389 / fmicb.2014.00354. PMC 4104785. PMID 25101062.
- ^ Xie B, Mazloum N, Liu L, Rahmeh A, Li H, Lee MY (listopad 2002). "Rekonstituce a charakterizace čtyřpodjednotkového holoenzymu lidské DNA polymerázy delta". Biochemie. 41 (44): 13133–42. doi:10.1021 / bi0262707. PMID 12403614.
- ^ Laboratoř, Mike Tyers. "Databáze proteinových, chemických a genetických interakcí | BioGRID". thebiogrid.org. Citováno 2016-04-25.
- ^ „Protein POLD1 (Homo sapiens) - STRING síťové zobrazení“. string-db.org. Citováno 2016-04-25.
- ^ „Databáze metabolismu rakovinných buněk ~~ Bioinformatika a laboratoř systémové medicíny ~~“. bioinfo.mc.vanderbilt.edu. Archivovány od originál dne 2016-04-26. Citováno 2016-04-25.
- ^ „STRING: funkční asociační sítě proteinů“. string-db.org. Citováno 2016-04-25.
- ^ Ono, Keiichiro. „Cytoscape: Open Source Platform pro komplexní síťovou analýzu a vizualizaci“. www.cytoscape.org. Citováno 2016-04-25.
- ^ „Protein POLD1 (Homo sapiens) - STRING síťové zobrazení“. string-db.org. Citováno 2016-04-25.
- ^ A b C Weedon MN, Ellard S, Prindle MJ, Caswell R, Lango Allen H, Oram R, Godbole K, Yajnik CS, Sbraccia P, Novelli G, Turnpenny P, McCann E, Goh KJ, Wang Y, Fulford J, McCulloch LJ, Savage DB, O'Rahilly S, Kos K, Loeb LA, Semple RK, Hattersley AT (srpen 2013). „Delece v rámci v aktivním místě polymerázy POLD1 způsobuje multisystémovou poruchu s lipodystrofií“. Genetika přírody. 45 (8): 947–50. doi:10,1038 / ng.2670. PMC 3785143. PMID 23770608.
- ^ "List Genatlas". genatlas.medecine.univ-paris5.fr. Citováno 2016-04-25.
- ^ Takahashi Y, Moriwaki S, Sugiyama Y, Endo Y, Yamazaki K, Mori T, Takigawa M, Inoue S (únor 2005). „Snížená genová exprese odpovědná za post-ultrafialovou syntézu opravy DNA ve stárnutí: možný mechanismus snížení kapacity opravy DNA související s věkem“. The Journal of Investigative Dermatology. 124 (2): 435–42. doi:10.1111 / j.0022-202X.2004.23591.x. PMID 15675965.
- ^ A b Wang JL, Guo HL, Wang PC, Liu CG (prosinec 2012). "Věkově závislá down-regulace DNA polymerázy δ1 v lidských lymfocytech". Molekulární a buněčná biochemie. 371 (1–2): 157–63. doi:10.1007 / s11010-012-1432-6. PMID 22915169. S2CID 15443915.
- ^ Karkhanis V, Wang L, Tae S, Hu YJ, Imbalzano AN, Sif S (srpen 2012). "Protein arginine methyltransferase 7 regulates cellular response to DNA damage by methylating promoter histones H2A and H4 of the polymerase δ catalytic subunit gene, POLD1". The Journal of Biological Chemistry. 287 (35): 29801–14. doi:10.1074/jbc.M112.378281. PMC 3436169. PMID 22761421.
- ^ Cui M, You L, Ren X, Zhao W, Liao Q, Zhao Y (únor 2016). "Dlouhá nekódující RNA PVT1 a rakovina". Sdělení o biochemickém a biofyzikálním výzkumu. 471 (1): 10–4. doi:10.1016 / j.bbrc.2015.12.101. PMID 26850852.
- ^ Li H, Xie B, Zhou Y, Rahmeh A, Trusa S, Zhang S, Gao Y, Lee EY, Lee MY (May 2006). „Funkční role p12, čtvrté podjednotky delty lidské DNA polymerázy“. The Journal of Biological Chemistry. 281 (21): 14748–55. doi:10,1074 / jbc.M600322200. PMID 16510448.
- ^ Podust VN, Chang LS, Ott R, Dianov GL, Fanning E (February 2002). "Reconstitution of human DNA polymerase delta using recombinant baculoviruses: the p12 subunit potentiates DNA polymerizing activity of the four-subunit enzyme". The Journal of Biological Chemistry. 277 (6): 3894–901. doi:10.1074/jbc.M109684200. PMID 11711545.
- ^ Zhang S, Zhou Y, Trusa S, Meng X, Lee EY, Lee MY (May 2007). "A novel DNA damage response: rapid degradation of the p12 subunit of dna polymerase delta". The Journal of Biological Chemistry. 282 (21): 15330–40. doi:10.1074/jbc.M610356200. PMID 17317665.
- ^ Lee MY, Zhang S, Lin SH, Wang X, Darzynkiewicz Z, Zhang Z, Lee EY (2014-01-01). "The tail that wags the dog: p12, the smallest subunit of DNA polymerase δ, is degraded by ubiquitin ligases in response to DNA damage and during cell cycle progression". Buněčný cyklus. 13 (1): 23–31. doi:10.4161/cc.27407. PMC 3925730. PMID 24300032.
- ^ Zhang S, Zhao H, Darzynkiewicz Z, Zhou P, Zhang Z, Lee EY, Lee MY (October 2013). "A novel function of CRL4(Cdt2): regulation of the subunit structure of DNA polymerase δ in response to DNA damage and during the S phase". The Journal of Biological Chemistry. 288 (41): 29550–61. doi:10.1074/jbc.M113.490466. PMC 3795253. PMID 23913683.
- ^ Fan X, Zhang Q, You C, Qian Y, Gao J, Liu P, Chen H, Song H, Chen Y, Chen K, Zhou Y (2014-01-01). "Proteolysis of the human DNA polymerase delta smallest subunit p12 by μ-calpain in calcium-triggered apoptotic HeLa cells". PLOS ONE. 9 (4): e93642. Bibcode:2014PLoSO...993642F. doi:10.1371/journal.pone.0093642. PMC 3972206. PMID 24691096.
- ^ Zhang Q, Zhang Q, Chen H, Chen Y, Zhou Y (February 2016). "Multiple forms of human DNA polymerase delta sub-assembling in cellular DNA transactions". Současná věda o proteinech a peptidech. 17 (8): 746–755. doi:10.2174/1389203717666160226145006. PMID 26916162.
- ^ A b Szekely AM, Chen YH, Zhang C, Oshima J, Weissman SM (October 2000). "Werner protein recruits DNA polymerase delta to the nucleolus". Sborník Národní akademie věd Spojených států amerických. 97 (21): 11365–70. Bibcode:2000PNAS...9711365S. doi:10.1073/pnas.97.21.11365. PMC 17206. PMID 11027336.
- ^ A b Karmakar P, Bohr VA (November 2005). "Cellular dynamics and modulation of WRN protein is DNA damage specific". Mechanismy stárnutí a rozvoje. 126 (11): 1146–58. doi:10.1016/j.mad.2005.06.004. PMID 16087220. S2CID 6128022.
- ^ Lee SY, Lee H, Kim ES, Park S, Lee J, Ahn B (April 2015). "WRN translocation from nucleolus to nucleoplasm is regulated by SIRT1 and required for DNA repair and the development of chemoresistance". Mutační výzkum. 774: 40–8. doi:10.1016/j.mrfmmm.2015.03.001. PMID 25801465.
- ^ Wang X, Zhang S, Zheng R, Yue F, Lin SH, Rahmeh AA, Lee EY, Zhang Z, Lee MY (February 2016). "PDIP46 (DNA polymerase δ interacting protein 46) is an activating factor for human DNA polymerase δ". Cílový cíl. 7 (5): 6294–313. doi:10.18632/oncotarget.7034. PMC 4868757. PMID 26819372.
- ^ Boyer AS, Walter D, Sørensen CS (January 2016). "DNA replication and cancer: From dysfunctional replication origin activities to therapeutic opportunities". Semináře z biologie rakoviny. 37-38: 16–25. doi:10.1016/j.semcancer.2016.01.001. PMID 26805514.
- ^ Sincennes MC, Humbert M, Grondin B, Lisi V, Veiga DF, Haman A, Cazaux C, Mashtalir N, Affar el B, Verreault A, Hoang T (February 2016). "The LMO2 oncogene regulates DNA replication in hematopoietic cells". Sborník Národní akademie věd Spojených států amerických. 113 (5): 1393–8. Bibcode:2016PNAS..113.1393S. doi:10.1073/pnas.1515071113. PMC 4747768. PMID 26764384.
- ^ Nick McElhinny SA, Gordenin DA, Stith CM, Burgers PM, Kunkel TA (April 2008). "Division of labor at the eukaryotic replication fork". Molekulární buňka. 30 (2): 137–44. doi:10.1016/j.molcel.2008.02.022. PMC 2654179. PMID 18439893.
- ^ A b Johnson RE, Klassen R, Prakash L, Prakash S (červenec 2015). „Hlavní role DNA polymerázy δ v replikaci předních i zaostávajících pramenů DNA“. Molekulární buňka. 59 (2): 163–75. doi:10.1016 / j.molcel.2015.05.038. PMC 4517859. PMID 26145172.
- ^ Daigaku Y, Keszthelyi A, Müller CA, Miyabe I, Brooks T, Retkute R, Hubank M, Nieduszynski CA, Carr AM (March 2015). "A global profile of replicative polymerase usage". Přírodní strukturní a molekulární biologie. 22 (3): 192–8. doi:10.1038/nsmb.2962. PMC 4789492. PMID 25664722.
- ^ Pavlov YI, Shcherbakova PV (March 2010). "DNA polymerases at the eukaryotic fork-20 years later". Mutační výzkum. 685 (1–2): 45–53. doi:10.1016/j.mrfmmm.2009.08.002. PMC 2822129. PMID 19682465.
- ^ Stillman B (July 2015). "Reconsidering DNA Polymerases at the Replication Fork in Eukaryotes". Molekulární buňka. 59 (2): 139–41. doi:10.1016/j.molcel.2015.07.004. PMC 4636199. PMID 26186286.
- ^ Burgers PM, Gordenin D, Kunkel TA (February 2016). "Who Is Leading the Replication Fork, Pol ε or Pol δ?". Molekulární buňka. 61 (4): 492–3. doi:10.1016/j.molcel.2016.01.017. PMC 4838066. PMID 26895421.
- ^ Korona DA, Lecompte KG, Pursell ZF (March 2011). "The high fidelity and unique error signature of human DNA polymerase epsilon". Výzkum nukleových kyselin. 39 (5): 1763–73. doi:10.1093/nar/gkq1034. PMC 3061053. PMID 21036870.
- ^ Skoneczna A, Kaniak A, Skoneczny M (November 2015). "Genetic instability in budding and fission yeast-sources and mechanisms". Recenze mikrobiologie FEMS. 39 (6): 917–67. doi:10.1093/femsre/fuv028. PMC 4608483. PMID 26109598.
- ^ Hoopes JI, Cortez LM, Mertz TM, Malc EP, Mieczkowski PA, Roberts SA (February 2016). "APOBEC3A and APOBEC3B Preferentially Deaminate the Lagging Strand Template during DNA Replication". Zprávy buněk. 14 (6): 1273–82. doi:10.1016/j.celrep.2016.01.021. PMC 4758883. PMID 26832400.
- ^ Reijns MA, Kemp H, Ding J, de Procé SM, Jackson AP, Taylor MS (February 2015). "Lagging-strand replication shapes the mutational landscape of the genome". Příroda. 518 (7540): 502–6. Bibcode:2015Natur.518..502R. doi:10.1038/nature14183. PMC 4374164. PMID 25624100.
- ^ Flood CL, Rodriguez GP, Bao G, Shockley AH, Kow YW, Crouse GF (March 2015). "Replicative DNA polymerase δ but not ε proofreads errors in Cis and in Trans". Genetika PLOS. 11 (3): e1005049. doi:10.1371/journal.pgen.1005049. PMC 4351087. PMID 25742645.
- ^ Herr AJ, Kennedy SR, Knowels GM, Schultz EM, Preston BD (March 2014). "DNA replication error-induced extinction of diploid yeast". Genetika. 196 (3): 677–91. doi:10.1534/genetics.113.160960. PMC 3948800. PMID 24388879.
- ^ Morrison A, Johnson AL, Johnston LH, Sugino A (April 1993). "Pathway correcting DNA replication errors in Saccharomyces cerevisiae". Časopis EMBO. 12 (4): 1467–73. doi:10.1002/j.1460-2075.1993.tb05790.x. PMC 413358. PMID 8385605.
- ^ Li L, Murphy KM, Kanevets U, Reha-Krantz LJ (June 2005). "Sensitivity to phosphonoacetic acid: a new phenotype to probe DNA polymerase delta in Saccharomyces cerevisiae". Genetika. 170 (2): 569–80. doi:10.1534/genetics.104.040295. PMC 1450396. PMID 15802517.
- ^ Meng X, Zhou Y, Zhang S, Lee EY, Frick DN, Lee MY (February 2009). "DNA damage alters DNA polymerase delta to a form that exhibits increased discrimination against modified template bases and mismatched primers". Výzkum nukleových kyselin. 37 (2): 647–57. doi:10.1093/nar/gkn1000. PMC 2632934. PMID 19074196.
- ^ Baranovskiy AG, Lada AG, Siebler HM, Zhang Y, Pavlov YI, Tahirov TH (May 2012). "DNA polymerase δ and ζ switch by sharing accessory subunits of DNA polymerase δ". The Journal of Biological Chemistry. 287 (21): 17281–7. doi:10.1074/jbc.M112.351122. PMC 3366816. PMID 22465957.
- ^ Markkanen E, Castrec B, Villani G, Hübscher U (December 2012). "A switch between DNA polymerases δ and λ promotes error-free bypass of 8-oxo-G lesions". Sborník Národní akademie věd Spojených států amerických. 109 (50): 20401–6. Bibcode:2012PNAS..10920401M. doi:10.1073/pnas.1211532109. PMC 3528542. PMID 23175785.
- ^ Song J, Hong P, Liu C, Zhang Y, Wang J, Wang P (2015-01-01). "Human POLD1 modulates cell cycle progression and DNA damage repair". BMC biochemie. 16: 14. doi:10.1186/s12858-015-0044-7. PMC 4471906. PMID 26087769.
- ^ Hocke S, Guo Y, Job A, Orth M, Ziesch A, Lauber K, De Toni EN, Gress TM, Herbst A, Göke B, Gallmeier E (February 2016). "A synthetic lethal screen identifies ATR-inhibition as a novel therapeutic approach for POLD1-deficient cancers". Cílový cíl. 7 (6): 7080–95. doi:10.18632/oncotarget.6857. PMC 4872770. PMID 26755646.
- ^ Miyabe I, Mizuno K, Keszthelyi A, Daigaku Y, Skouteri M, Mohebi S, Kunkel TA, Murray JM, Carr AM (November 2015). "Polymerase δ replicates both strands after homologous recombination-dependent fork restart". Přírodní strukturní a molekulární biologie. 22 (11): 932–8. doi:10.1038/nsmb.3100. PMC 4655445. PMID 26436826.
- ^ Kamath-Loeb AS, Shen JC, Schmitt MW, Loeb LA (April 2012). „Exonukleáza s Wernerovým syndromem usnadňuje degradaci DNA a vysoce věrnou polymeraci DNA lidskou DNA polymerázou δ“. The Journal of Biological Chemistry. 287 (15): 12480–90. doi:10.1074 / jbc.M111.332577. PMC 3320997. PMID 22351772.
- ^ Jansen AM, van Wezel T, van den Akker BE, Ventayol Garcia M, Ruano D, Tops CM, Wagner A, Letteboer TG, Gómez-García EB, Devilee P, Wijnen JT, Hes FJ, Morreau H (December 2015). "Combined mismatch repair and POLE/POLD1 defects explain unresolved suspected Lynch syndrome cancers". European Journal of Human Genetics. 24 (7): 1089–1092. doi:10.1038/ejhg.2015.252. PMC 5070903. PMID 26648449.
- ^ A b C d E F Palles C, Cazier JB, Howarth KM, Domingo E, Jones AM, Broderick P, et al. (Únor 2013). "Germline mutations affecting the proofreading domains of POLE and POLD1 predispose to colorectal adenomas and carcinomas". Genetika přírody. 45 (2): 136–44. doi:10.1038/ng.2503. PMC 3785128. PMID 23263490.
- ^ A b C d Valle L, Hernández-Illán E, Bellido F, Aiza G, Castillejo A, Castillejo MI, et al. (Červenec 2014). "New insights into POLE and POLD1 germline mutations in familial colorectal cancer and polyposis". Lidská molekulární genetika. 23 (13): 3506–12. doi:10.1093/hmg/ddu058. PMID 24501277.
- ^ A b C d Bellido F, Pineda M, Aiza G, Valdés-Mas R, Navarro M, Puente DA, Pons T, González S, Iglesias S, Darder E, Piñol V, Soto JL, Valencia A, Blanco I, Urioste M, Brunet J, Lázaro C, Capellá G, Puente XS, Valle L (April 2016). "POLE and POLD1 mutations in 529 kindred with familial colorectal cancer and/or polyposis: review of reported cases and recommendations for genetic testing and surveillance". Genetika v medicíně. 18 (4): 325–32. doi:10.1038/gim.2015.75. PMC 4823640. PMID 26133394.
- ^ A b C d Briggs S, Tomlinson I (June 2013). "Germline and somatic polymerase ε and δ mutations define a new class of hypermutated colorectal and endometrial cancers". The Journal of Pathology. 230 (2): 148–53. doi:10.1002/path.4185. PMC 3709119. PMID 23447401.
- ^ A b C Church DN, Briggs SE, Palles C, Domingo E, Kearsey SJ, Grimes JM, Gorman M, Martin L, Howarth KM, Hodgson SV, Kaur K, Taylor J, Tomlinson IP (July 2013). "DNA polymerase ε and δ exonuclease domain mutations in endometrial cancer". Lidská molekulární genetika. 22 (14): 2820–8. doi:10.1093/hmg/ddt131. PMC 3690967. PMID 23528559.
- ^ A b Heitzer E, Tomlinson I (February 2014). "Replicative DNA polymerase mutations in cancer". Aktuální názor na genetiku a vývoj. 24: 107–13. doi:10.1016/j.gde.2013.12.005. PMC 4003352. PMID 24583393.
- ^ A b Shinbrot E, Henninger EE, Weinhold N, Covington KR, Göksenin AY, Schultz N, Chao H, Doddapaneni H, Muzny DM, Gibbs RA, Sander C, Pursell ZF, Wheeler DA (November 2014). "Exonuclease mutations in DNA polymerase epsilon reveal replication strand specific mutation patterns and human origins of replication". Výzkum genomu. 24 (11): 1740–50. doi:10.1101/gr.174789.114. PMC 4216916. PMID 25228659.
- ^ Arora S, Yan H, Cho I, Fan HY, Luo B, Gai X, Bodian DL, Vockley JG, Zhou Y, Handorf EA, Egleston BL, Andrake MD, Nicolas E, Serebriiskii IG, Yen TJ, Hall MJ, Golemis EA, Enders GH (December 2015). "Genetic Variants That Predispose to DNA Double-Strand Breaks in Lymphocytes From a Subset of Patients With Familial Colorectal Carcinomas". Gastroenterologie. 149 (7): 1872–1883.e9. doi:10.1053/j.gastro.2015.08.052. PMC 4663158. PMID 26344056.
- ^ Waterfall JJ, Meltzer PS (March 2015). "Avalanching mutations in biallelic mismatch repair deficiency syndrome". Genetika přírody. 47 (3): 194–6. doi:10.1038/ng.3227. PMID 25711864. S2CID 28165945.
- ^ A b Schlesner M, Eils R (2015-01-01). "Hypermutation takes the driver's seat". Genomová medicína. 7 (1): 31. doi:10.1186/s13073-015-0159-x. PMC 4376156. PMID 25821521.
- ^ Shlien A, Campbell BB, de Borja R, Alexandrov LB, Merico D, Wedge D, et al. (Březen 2015). "Combined hereditary and somatic mutations of replication error repair genes result in rapid onset of ultra-hypermutated cancers". Genetika přírody. 47 (3): 257–62. doi:10.1038/ng.3202. PMID 25642631. S2CID 5338516.
- ^ Bouffet E, Larouche V, Campbell BB, Merico D, de Borja R, Aronson M, et al. (Březen 2016). "Immune Checkpoint Inhibition for Hypermutant Glioblastoma Multiforme Resulting From Germline Biallelic Mismatch Repair Deficiency". Journal of Clinical Oncology. 34 (19): 2206–2211. doi:10.1200/JCO.2016.66.6552. PMID 27001570.
- ^ Howitt BE, Shukla SA, Sholl LM, Ritterhouse LL, Watkins JC, Rodig S, Stover E, Strickland KC, D'Andrea AD, Wu CJ, Matulonis UA, Konstantinopoulos PA (December 2015). "Association of Polymerase e-Mutated and Microsatellite-Instable Endometrial Cancers With Neoantigen Load, Number of Tumor-Infiltrating Lymphocytes, and Expression of PD-1 and PD-L1" (PDF). JAMA Onkologie. 1 (9): 1319–23. doi:10.1001/jamaoncol.2015.2151. PMID 26181000.
- ^ van Gool IC, Eggink FA, Freeman-Mills L, Stelloo E, Marchi E, de Bruyn M, Palles C, Nout RA, de Kroon CD, Osse EM, Klenerman P, Creutzberg CL, Tomlinson IP, Smit VT, Nijman HW, Bosse T, Church DN (July 2015). "POLE Proofreading Mutations Elicit an Antitumor Immune Response in Endometrial Cancer". Klinický výzkum rakoviny. 21 (14): 3347–55. doi:10.1158/1078-0432.CCR-15-0057. PMC 4627582. PMID 25878334.
- ^ Khanna A (June 2015). "DNA damage in cancer therapeutics: a boon or a curse?". Výzkum rakoviny. 75 (11): 2133–8. doi:10.1158/0008-5472.CAN-14-3247. PMID 25931285.
- ^ Roberts SA, Gordenin DA (December 2014). „Hypermutace v lidských genomech rakoviny: stopy a mechanismy“. Recenze přírody. Rakovina. 14 (12): 786–800. doi:10.1038 / nrc3816. PMC 4280484. PMID 25568919.
- ^ Roos WP, Thomas AD, Kaina B (January 2016). "DNA damage and the balance between survival and death in cancer biology" (PDF). Recenze přírody. Rakovina. 16 (1): 20–33. doi:10.1038/nrc.2015.2. PMID 26678314. S2CID 10159855.
- ^ da Costa LT, Liu B, el-Deiry W, Hamilton SR, Kinzler KW, Vogelstein B, Markowitz S, Willson JK, de la Chapelle A, Downey KM (January 1995). "Polymerase delta variants in RER colorectal tumours". Genetika přírody. 9 (1): 10–1. doi:10.1038/ng0195-10. PMID 7704014. S2CID 19545401.
- ^ Flohr T, Dai JC, Büttner J, Popanda O, Hagmüller E, Thielmann HW (March 1999). "Detection of mutations in the DNA polymerase delta gene of human sporadic colorectal cancers and colon cancer cell lines". International Journal of Cancer. 80 (6): 919–29. doi:10.1002/(sici)1097-0215(19990315)80:6<919::aid-ijc19>3.0.co;2-u. PMID 10074927.
- ^ Preston BD, Albertson TM, Herr AJ (October 2010). "DNA replication fidelity and cancer". Semináře z biologie rakoviny. 20 (5): 281–93. doi:10.1016/j.semcancer.2010.10.009. PMC 2993855. PMID 20951805.
- ^ Popanda O, Flohr T, Fox G, Thielmann HW (November 1999). "A mutation detected in DNA polymerase delta cDNA from Novikoff hepatoma cells correlates with abnormal catalytic properties of the enzyme". Journal of Cancer Research and Clinical Oncology. 125 (11): 598–608. doi:10.1007/s004320050322. PMID 10541966. S2CID 11582153.
- ^ Venkatesan RN, Treuting PM, Fuller ED, Goldsby RE, Norwood TH, Gooley TA, Ladiges WC, Preston BD, Loeb LA (November 2007). "Mutation at the polymerase active site of mouse DNA polymerase delta increases genomic instability and accelerates tumorigenesis". Molekulární a buněčná biologie. 27 (21): 7669–82. doi:10.1128/MCB.00002-07. PMC 2169052. PMID 17785453.
- ^ A b C Pelosini C, Martinelli S, Ceccarini G, Magno S, Barone I, Basolo A, Fierabracci P, Vitti P, Maffei M, Santini F (November 2014). "Identification of a novel mutation in the polymerase delta 1 (POLD1) gene in a lipodystrophic patient affected by mandibular hypoplasia, deafness, progeroid features (MDPL) syndrome". Metabolismus. 63 (11): 1385–9. doi:10.1016/j.metabol.2014.07.010. PMID 25131834.
- ^ Reinier F, Zoledziewska M, Hanna D, Smith JD, Valentini M, Zara I, Berutti R, Sanna S, Oppo M, Cusano R, Satta R, Montesu MA, Jones C, Cerimele D, Nickerson DA, Angius A, Cucca F, Cottoni F, Crisponi L (November 2015). "Mandibular hypoplasia, deafness, progeroid features and lipodystrophy (MDPL) syndrome in the context of inherited lipodystrophies". Metabolismus. 64 (11): 1530–40. doi:10.1016/j.metabol.2015.07.022. PMID 26350127.
- ^ Oshima J, Sidorova JM, Monnat RJ (March 2016). "Werner syndrome: Clinical features, pathogenesis and potential therapeutic interventions". Recenze výzkumu stárnutí. 33: 105–114. doi:10.1016/j.arr.2016.03.002. PMC 5025328. PMID 26993153.
- ^ Lessel D, Hisama FM, Szakszon K, Saha B, Sanjuanelo AB, Salbert BA, Steele PD, Baldwin J, Brown WT, Piussan C, Plauchu H, Szilvássy J, Horkay E, Högel J, Martin GM, Herr AJ, Oshima J, Kubisch C (November 2015). "POLD1 Germline Mutations in Patients Initially Diagnosed with Werner Syndrome". Lidská mutace. 36 (11): 1070–9. doi:10.1002/humu.22833. PMC 4684254. PMID 26172944.
- ^ Pachlopnik Schmid J, Lemoine R, Nehme N, Cormier-Daire V, Revy P, Debeurme F, Debré M, Nitschke P, Bole-Feysot C, Legeai-Mallet L, Lim A, de Villartay JP, Picard C, Durandy A, Fischer A, de Saint Basile G (December 2012). "Polymerase ε1 mutation in a human syndrome with facial dysmorphism, immunodeficiency, livedo, and short stature ("FILS syndrome")". The Journal of Experimental Medicine. 209 (13): 2323–30. doi:10.1084/jem.20121303. PMC 3526359. PMID 23230001.
- ^ Thiffault I, Saunders C, Jenkins J, Raje N, Canty K, Sharma M, Grote L, Welsh HI, Farrow E, Twist G, Miller N, Zwick D, Zellmer L, Kingsmore SF, Safina NP (2015-01-01). "A patient with polymerase E1 deficiency (POLE1): clinical features and overlap with DNA breakage/instability syndromes". Lékařská genetika BMC. 16: 31. doi:10.1186/s12881-015-0177-y. PMC 4630961. PMID 25948378.
- ^ Guénantin AC, Briand N, Bidault G, Afonso P, Béréziat V, Vatier C, Lascols O, Caron-Debarle M, Capeau J, Vigouroux C (May 2014). "Nuclear envelope-related lipodystrophies". Semináře z buněčné a vývojové biologie. 29: 148–57. doi:10.1016/j.semcdb.2013.12.015. PMID 24384368.
- ^ A b "GeneTests.org". GeneTests.org. Citováno 2016-04-25.
- ^ "MDP syndrome caused by a change in the POLD1 gene".





