Struktura bílkovin - Protein structure
tento článek potřebuje další citace pro ověření. (Květen 2018) (Zjistěte, jak a kdy odstranit tuto zprávu šablony) |

Struktura bílkovin je trojrozměrné uspořádání atomů v aminokyselina -řetěz molekula. Proteiny jsou polymery - konkrétně polypeptidy - vytvořeno ze sekvencí aminokyseliny, monomery polymeru. Jeden aminokyselinový monomer může být také nazýván a zbytek označující opakující se jednotku polymeru. Proteiny se tvoří podstupovanými aminokyselinami kondenzační reakce, ve kterém aminokyseliny jednu ztratí molekula vody za reakce za účelem vzájemného připojení pomocí a peptidová vazba. Podle konvence je řetězec s 30 aminokyselinami často identifikován jako a peptid spíše než protein.[1] Aby bylo možné vykonávat svou biologickou funkci, proteiny se skládají do jedné nebo více specifických prostorových konformací řízených řadou nekovalentní interakce jako vodíkové vazby, iontové interakce, Van der Waalsovy síly, a hydrofobní balení. Abychom porozuměli funkcím proteinů na molekulární úrovni, je často nutné určit jejich trojrozměrnou strukturu. Toto je téma vědecké oblasti strukturní biologie, který využívá techniky jako Rentgenová krystalografie, NMR spektroskopie, kryoelektronová mikroskopie (cryo-EM) a duální polarizační interferometrie k určení struktury proteinů.
Velikost proteinových struktur se pohybuje v rozmezí desítek až několika tisíc aminokyselin.[2] Podle fyzické velikosti jsou proteiny klasifikovány jako nanočástice mezi 1–100 nm. Mohou být vytvořeny velmi velké agregáty proteinové podjednotky. Například mnoho tisíc aktin molekuly se spojí do mikrofilament.
Protein obecně prochází reverzibilní strukturální změny při výkonu jeho biologické funkce. Alternativní struktury stejného proteinu jsou označovány jako odlišné konformační izomery nebo jednoduše se nazývají konformace a přechody mezi nimi konformační změny.
Úrovně struktury bílkovin
Existují čtyři odlišné úrovně proteinové struktury.
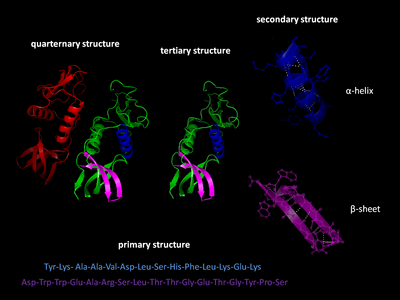
Primární struktura
The primární struktura proteinu označuje sekvenci aminokyseliny v polypeptidovém řetězci. Primární strukturu drží pohromadě peptidové vazby které se vyrábějí během procesu biosyntéza bílkovin. Oba konce polypeptidový řetězec jsou označovány jako karboxylový konec (C-konec) a amino konec (N-konec) na základě povahy volné skupiny na každé končetině. Počítání zbytků vždy začíná na N-terminálním konci (NH2-skupina), což je konec, kde aminoskupina není zahrnuta v peptidové vazbě. Primární struktura proteinu je určena gen odpovídající proteinu. Specifická posloupnost nukleotidy v DNA je přepsal do mRNA, který čte ribozom v procesu zvaném překlad. Sekvence aminokyselin v inzulínu byla objevena Frederick Sanger, kterým se stanoví, že proteiny mají definující aminokyselinové sekvence.[3][4] Sekvence proteinu je pro tento protein jedinečná a definuje strukturu a funkci proteinu. Sekvence proteinu může být stanovena způsoby, jako je Edmanova degradace nebo tandemová hmotnostní spektrometrie. Často se však čte přímo ze sekvence genu pomocí genetický kód. Při diskusi o proteinech se striktně doporučuje používat slova „aminokyselinové zbytky“, protože když se vytvoří peptidová vazba, a molekula vody se ztrácí, a proto jsou bílkoviny tvořeny aminokyselinovými zbytky. Posttranslační modifikace jako fosforylace a glykosylace jsou obvykle také považovány za součást primární struktury a nelze je číst z genu. Například, inzulín se skládá z 51 aminokyselin ve 2 řetězcích. Jeden řetězec má 31 aminokyselin a druhý 20 aminokyselin.
Sekundární struktura

Sekundární struktura "Polypeptidový řetězec" označuje velmi pravidelné lokální substruktury na skutečném řetězci páteřního polypeptidu. Dva hlavní typy sekundární struktury, α-šroubovice a β-vlákno nebo β-listy, navrhl v roce 1951 Linus Pauling et al.[5] Tyto sekundární struktury jsou definovány vzory Vodíkové vazby mezi peptidovými skupinami s hlavním řetězcem. Mají pravidelnou geometrii, přičemž jsou omezeny na konkrétní hodnoty vzepětí úhlů ψ a φ na Ramachandran spiknutí. Jak a-helix, tak β-list představují způsob nasycení všech donorů a akceptorů vodíkové vazby v peptidové kostře. Některé části proteinu jsou uspořádány, ale netvoří žádné pravidelné struktury. Neměli by být zaměňováni s náhodná cívka, rozložený polypeptidový řetězec postrádající jakoukoli pevnou trojrozměrnou strukturu. Několik po sobě následujících sekundárních struktur může tvořitsupersekundární jednotka ".[6]
Terciární struktura
Terciární struktura "Termín" označuje trojrozměrnou strukturu monomerních a multimerních proteinových molekul. Α-šroubovice a β-skládané listy jsou složeny do kompaktu kulová struktura. Skládání je poháněno nespecifické hydrofobní interakce pohřeb hydrofobní zbytky z voda, ale struktura je stabilní, pouze když části a proteinová doména jsou zajištěny na místě pomocí charakteristický terciární interakce, jako např solné mosty, vodíkové vazby a těsné balení postranních řetězců a disulfidové vazby. Disulfidové vazby jsou v cytosolických proteinech extrémně vzácné, protože cytosol (intracelulární tekutina) je obecně a snižování životní prostředí.
Kvartérní struktura
Kvartérní struktura je trojrozměrná struktura skládající se z agregace dvou nebo více jednotlivých polypeptidových řetězců (podjednotek), které fungují jako jedna funkční jednotka (multimer ). Výsledný multimer je stejný nekovalentní interakce a disulfidové vazby jako v terciární struktuře. Existuje mnoho možných organizací kvartérní struktury.[7] Jsou nazývány komplexy dvou nebo více polypeptidů (tj. Více podjednotek) multimery. Konkrétně by se tomu říkalo a dimer pokud obsahuje dvě podjednotky, a zastřihovač pokud obsahuje tři podjednotky, a tetramer pokud obsahuje čtyři podjednotky, a pentamer pokud obsahuje pět podjednotek. Podjednotky spolu často souvisí operace symetrie, například 2-násobná osa v dimeru. Multimery vytvořené ze stejných podjednotek se označují předponou „homo-“ a ty, které se skládají z různých podjednotek, se označují předponou „hetero-“, například heterotetramer, jako jsou dva alfa a dva beta řetězy hemoglobin.
Domény, motivy a záhyby proteinové struktury

Proteiny jsou často popisovány jako sestávající z několika strukturních jednotek. Tyto jednotky zahrnují domény, motivy a záhyby. Navzdory skutečnosti, že existuje přibližně 100 000 různých proteinů exprimovaných v eukaryotický systémů, existuje mnohem méně různých domén, strukturálních motivů a záhybů.
Strukturální doména
A strukturální doména je prvek celkové struktury proteinu, který se často samo stabilizuje záhyby nezávisle na zbytku proteinového řetězce. Mnoho domén není jedinečných pro proteinové produkty jedné gen nebo jeden genová rodina ale místo toho se objevují v různých proteinech. Domény jsou často pojmenovány a vyčleněny, protože zaujímají prominentní místo v biologické funkci proteinu, ke kterému patří; například „vápník -vázací doména klimodulin ". Protože jsou nezávisle stabilní, lze domény" vyměnit "za genetické inženýrství mezi jedním a druhým proteinem chiméra bílkoviny.
Strukturální a sekvenční motiv
The strukturální a sekvence motivy označují krátké segmenty trojrozměrné struktury proteinu nebo aminokyselinové sekvence, které byly nalezeny ve velkém počtu různých proteinů.
Naddruhá struktura
The supersekundární struktura se týká konkrétní kombinace sekundární struktura prvky, jako jsou jednotky β-α-β nebo a helix-turn-helix motiv. Některé z nich mohou být také označovány jako strukturální motivy.
Složení bílkovin
Proteinový záhyb označuje obecnou proteinovou architekturu, jako a svazek šroubovice, β-hlaveň, Rossmann fold nebo různé "záhyby" uvedené v Databáze strukturní klasifikace proteinů.[8] Související koncept je topologie proteinů to se týká uspořádání kontaktů v proteinu.
Superdoména
Superdoména se skládá ze dvou nebo více nominálně nesouvisejících strukturálních domén, které se dědí jako jedna jednotka a vyskytují se v různých proteinech.[9] Příklad poskytuje protein tyrosin fosfatáza doména a Doména C2 spárovat PTEN, několik tensin bílkoviny, auxilin a bílkoviny v rostlinách a houbách. Superdoména PTP-C2 zřejmě vznikla před divergencí hub, rostlin a živočichů, a proto bude pravděpodobně stará asi 1,5 miliardy let.[Citace je zapotřebí ]
Dynamika proteinů

Proteiny nejsou však přísně statické objekty, ale spíše naplňují soubory konformační stavy K přechodům mezi těmito stavy obvykle dochází nanoměřítky, a byly spojeny s funkčně relevantními jevy jako např alosterická signalizace[10] a enzymová katalýza.[11] Dynamika proteinů a konformační změny allowproteins fungovat jako nanoscale biologické stroje uvnitř buněk, často ve formě multi-proteinové komplexy.[12] Mezi příklady patří motorické proteiny, jako myosin, který je odpovědný za sval kontrakce, kinesin, který pohybuje nákladem uvnitř buněk směrem od jádro podél mikrotubuly, a dynein, který pohybuje nákladem uvnitř buněk směrem k jádru a produkuje axonemální výprask pohyblivé řasinky a bičíky. „[I] n efekt, [motile cilium] je nanomachin složený z asi více než 600 proteinů v molekulárních komplexech, z nichž mnohé také fungují nezávisle jako nanomachines ...Flexibilní linkery povolit mobilní proteinové domény jimi propojené, aby přijaly své závazné partnery a přiměly dálkový dosah allostery přes dynamika proteinové domény. "[13]
Skládání bílkovin
Tato sekce potřebuje expanzi. Můžete pomoci přidávat k tomu. (Dubna 2019) |
Jak je přeloženo, polypeptidy opouštějí ribozom jako náhodná cívka a složí se do jeho rodný stát.[14][15] Vzhledem k tomu, že záhyb je určen sítí interakcí mezi aminokyselinami v polypeptidu, je konečná struktura proteinového řetězce určena jeho aminokyselinovou sekvencí (Anfinsenovo dogma ).[16]
Stabilita bílkovin
Stabilita proteinu závisí na několika faktorech, například 1) nekovalentní elektrostatické interakce 2) hydrofobní interakce Tyto interakční energie jsou řádově 20–40 kJ / mol.[Citace je zapotřebí ] Bílkoviny jsou velmi citlivé na měnící se teploty a změna teploty může mít za následek vývoj nebo denaturaci. Denaturace bílkovin může mít za následek ztrátu funkce a ztrátu nativního stavu. Nebo to může být také primitivní stav.
Rentgenová krystalografie a kalorimetrie naznačuje, že neexistuje žádný obecný mechanismus, který by popisoval vliv změny teploty na funkce a strukturu proteinů. To je způsobeno skutečností, že proteiny z energetického hlediska nepředstavují jednotnou třídu chemických entit. Struktura a stabilita jednotlivého proteinu závisí na poměru jeho polárních a nepolárních zbytků. Přispívají ke konformačním a čistým entalpím lokálních a nelokálních interakcí.
Vezmeme-li v úvahu slabé intermolekulární interakce odpovědné za strukturální integritu, je těžké předpovědět účinky teploty, protože existuje příliš mnoho neznámých faktorů, které přispívají k hypotetické bilanci volné energie a její teplotní závislosti. Vnitřní solné vazby produkují tepelnou stabilitu a není známo, zda má studená teplota za následek destabilizaci těchto vazeb.
Volná energie pro stabilizaci rozpustných globulárních proteinů v zásadě nepřesahuje 50-100 kJ / mol.[Citace je zapotřebí ] Stabilizace je založena na ekvivalentu několika vodíkových vazeb, iontových párů nebo hydrofobních interakcí, přestože výsledkem stabilizace je mnoho intramolekulárních interakcí. Vezmeme-li v úvahu velký počet vodíkových vazeb, které probíhají ke stabilizaci sekundárních struktur a ke stabilizaci vnitřního jádra prostřednictvím hydrofobních interakcí, se volná energie stabilizace jeví jako malý rozdíl mezi velkým počtem. Struktura nativního proteinu proto není optimalizována pro maximální stabilitu.[17]
Stanovení proteinové struktury


Asi 90% proteinových struktur dostupných v Proteinová datová banka byly určeny Rentgenová krystalografie.[18] Tato metoda umožňuje měřit trojrozměrné (3-D) rozdělení hustoty elektrony v proteinu, v vykrystalizoval stát, a tím usoudit 3-D souřadnice všech atomy být určen k určitému rozlišení. Zhruba 9% známých proteinových struktur bylo získáno pomocí nukleární magnetická rezonance (NMR) techniky.[Citace je zapotřebí ] U větších proteinových komplexů kryo-elektronová mikroskopie může určit proteinové struktury. Rozlišení je obvykle nižší než rozlišení rentgenové krystalografie nebo NMR, ale maximální rozlišení se neustále zvyšuje. Tato technika je stále zvláště cenná pro velmi velké proteinové komplexy, jako je virové obalové proteiny a amyloid vlákna.
Obecné složení sekundární struktury lze určit pomocí kruhový dichroismus. Vibrační spektroskopie lze také použít k charakterizaci konformace peptidů, polypeptidů a proteinů.[19] Dvojrozměrná infračervená spektroskopie se stala cennou metodou pro zkoumání struktur flexibilních peptidů a proteinů, které nelze studovat jinými metodami.[20][21] Kvalitativnější obraz proteinové struktury často získá proteolýza, což je také užitečné pro screening více krystalizovatelných proteinových vzorků. Nové implementace tohoto přístupu, včetně rychlá paralelní proteolýza (FASTpp), může zkoumat strukturovanou frakci a její stabilitu bez nutnosti čištění.[22] Jakmile byla struktura proteinu experimentálně stanovena, lze provést další podrobné studie výpočtově pomocí molekulární dynamika simulace této struktury.[23]
Analýza proteinové sekvence: Soubory
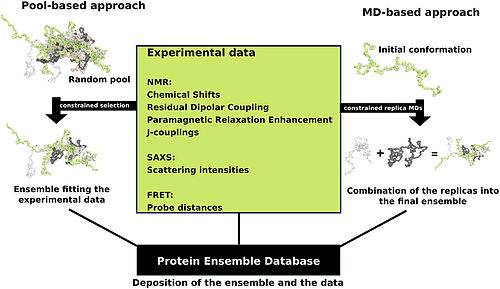
Proteiny jsou často považovány za relativně stabilní struktury, které mají soubor terciární struktura a zažít konformační změny v důsledku modifikace jinými proteiny nebo jako součást enzymatické aktivity. Proteiny však mají různé stupně stability a některé méně stabilní varianty ano vnitřně neuspořádané proteiny. Tyto proteiny existují a fungují v relativně „neuspořádaném“ stavu bez stabilního stavu terciární struktura. Ve výsledku je obtížné je popsat ve standardním modelu proteinové struktury, který byl navržen pro proteiny s fixní terciární struktura. Konformační soubory byly navrženy jako způsob, jak zajistit přesnější a „dynamičtější“ reprezentaci konformačního stavu vnitřně neuspořádané proteiny. Konformační soubory funkce pokusem reprezentovat různé konformace vnitřně neuspořádané proteiny v souboru souboru (typ nalezený na Databáze proteinových souborů ).
Protein soubor soubory jsou reprezentací proteinu, který lze považovat za flexibilní strukturu. Vytváření těchto souborů vyžaduje určení, které z různých teoreticky možných konformací proteinů skutečně existují. Jedním z přístupů je použití výpočetních algoritmů na data o proteinu, aby se pokusila určit nejpravděpodobnější sadu konformací pro soubor soubor.
Existuje několik metod pro přípravu dat pro Databáze proteinových souborů které spadají do dvou obecných metodologií - přístupy pool a molekulární dynamiky (MD) (znázorněné na obrázku). Přístup založený na poolu využívá aminokyselinovou sekvenci proteinu k vytvoření obrovské zásoby náhodných konformací. Tento fond je poté podroben více výpočetnímu zpracování, které vytváří sadu teoretických parametrů pro každou konformaci založenou na struktuře. Jsou vybrány konformační podskupiny z tohoto souboru, jejichž průměrné teoretické parametry se velmi shodují se známými experimentálními daty pro tento protein.[24]
Přístup molekulární dynamiky vyžaduje více náhodných konformací najednou a všechny podrobuje experimentálním datům. Zde experimentální data slouží jako omezení pro konformace (např. Známé vzdálenosti mezi atomy). Přijaty jsou pouze konformace, kterým se podaří zůstat v mezích stanovených experimentálními daty. Tento přístup často aplikuje velké množství experimentálních dat na konformace, což je výpočetně velmi náročný úkol.[24]
| Protein | Datový typ | Protokol | PED ID | Reference |
|---|---|---|---|---|
| Sic1 /Cdc4 | NMR a SAXS | Na základě bazénu | PED9AAA | [25] |
| p15 PAF | NMR a SAXS | Na základě bazénu | PED6AAA | [26] |
| MKK7 | NMR | Na základě bazénu | PED5AAB | [27] |
| Beta-synuklein | NMR | Na bázi MD | PED1AAD | [28] |
| P27 DÍTĚ | NMR | Na bázi MD | PED2AAA | [29] |
(převzato z obraz v „Výpočtových přístupech pro odvození funkcí vnitřně neuspořádaných proteinů“[24])
Databáze proteinové struktury
A databáze proteinové struktury je databáze, která je modelován kolem různých experimentálně stanoveno proteinové struktury. Cílem většiny databází proteinových struktur je uspořádat a anotovat proteinové struktury a poskytnout biologické komunitě přístup k experimentálním datům užitečným způsobem. Data obsažená v databázích proteinových struktur často obsahují 3D souřadnice i experimentální informace, jako jsou rozměry a úhly jednotkových buněk rentgenová krystalografie určené struktury. Ačkoli většina případů, v tomto případě buď proteiny, nebo stanovení specifické struktury proteinu, obsahují také informace o sekvenci a některé databáze dokonce poskytují prostředky pro provádění dotazů na sekvenci, primárním atributem struktury databáze jsou strukturální informace, zatímco sekvenční databáze zaměřit se na informace o sekvenci a neobsahovat žádné strukturální informace pro většinu záznamů. Databáze struktur proteinů jsou pro mnoho snah kritické výpočetní biologie jako návrh léku na základě struktury, a to jak při vývoji použitých výpočetních metod, tak při poskytování velkého experimentálního datového souboru používaného některými metodami k získání poznatků o funkci proteinu.[30]
Klasifikace struktury
Proteinové struktury lze seskupit na základě jejich strukturní podobnosti, topologická třída nebo obyčejný evoluční původ. The Databáze strukturní klasifikace proteinů[31] a KOCOUR databáze[32] poskytují dvě různé strukturní klasifikace proteinů. Když je strukturální podobnost velká, oba proteiny se pravděpodobně lišily od společného předka[33]a sdílená struktura mezi proteiny je považována za důkaz homologie. Podobnost struktury pak může být použita ke seskupení proteinů do proteinové superrodiny.[34]. Pokud je sdílená struktura významná, ale sdílený zlomek je malý, sdílený fragment může být důsledkem dramatičtější evoluční události, jako je horizontální přenos genů a připojení proteinů sdílejících tyto fragmenty do proteinových superrodin již není oprávněné.[33] Topologii proteinu lze také použít ke klasifikaci proteinů. Teorie uzlů a topologie obvodu jsou dva topologické rámce vyvinuté pro klasifikaci proteinových záhybů na základě křížení řetězců a intrachainových kontaktů.
Výpočetní predikce proteinové struktury
Generace a proteinová sekvence je mnohem jednodušší než stanovení proteinové struktury. Struktura proteinu však poskytuje mnohem více informací o funkci proteinu než o jeho sekvenci. Proto byla vyvinuta řada metod pro výpočetní predikci proteinové struktury z její sekvence.[35] Ab initio metody predikce používají pouze sekvenci proteinu. Závitování a homologické modelování metody mohou postavit 3-D model pro protein neznámé struktury z experimentálních struktur evolučně příbuzných proteinů, nazývaných a rodina bílkovin.
Viz také
- 3did
- Biomolekulární struktura
- Genová struktura
- Struktura nukleové kyseliny
- Z-matice převod z torzních úhlů na kartézské souřadnice
- Pás karet diagram 3D schematické znázornění proteinů
Reference
- ^ H. Stephen Stoker (1. ledna 2015). Organická a biologická chemie. Cengage Learning. p. 371. ISBN 978-1-305-68645-8.
- ^ Brocchieri L, Karlin S (10. června 2005). "Délka proteinu v eukaryotických a prokaryotických proteomech". Výzkum nukleových kyselin. 33 (10): 3390–3400. doi:10.1093 / nar / gki615. PMC 1150220. PMID 15951512.
- ^ Sanger, F .; Tuppy, H. (1. září 1951). „Aminokyselinová sekvence ve fenylalanylovém řetězci inzulínu. I. Identifikace nižších peptidů z parciálních hydrolyzátů“. The Biochemical Journal. 49 (4): 463–481. doi:10.1042 / bj0490463. ISSN 0264-6021. PMC 1197535. PMID 14886310.
- ^ Sanger, F. (15. května 1959). "Chemie inzulínu". Věda. 129 (3359): 1340–1344. Bibcode:1959Sci ... 129.1340G. doi:10.1126 / science.129.3359.1340. ISSN 0036-8075. PMID 13658959.
- ^ Pauling L, Corey RB, Branson HR (1951). "Struktura proteinů; dvě vodíkově vázané šroubovicové konfigurace polypeptidového řetězce". Proc Natl Acad Sci USA. 37 (4): 205–211. Bibcode:1951PNAS ... 37..205P. doi:10.1073 / pnas.37.4.205. PMC 1063337. PMID 14816373.
- ^ Chiang YS, Gelfand TI, Kister AE, Gelfand IM (2007). „Nová klasifikace supersekundárních struktur sendvičových proteinů odkrývá přísné vzory sdružování řetězců“. Proteiny. 68 (4): 915–921. doi:10,1002 / prot. 21473. PMID 17557333.
- ^ Moutevelis E, Woolfson DN (leden 2009). "Periodická tabulka proteinových struktur ve svitcích-cívkách". J. Mol. Biol. 385 (3): 726–32. doi:10.1016 / j.jmb.2008.11.028. ISSN 0022-2836. PMID 19059267.
- ^ Govindarajan S, Recabarren R, Goldstein RA (17. září 1999). „Odhad celkového počtu záhybů proteinů“. Proteiny. 35 (4): 408–414. doi:10.1002 / (SICI) 1097-0134 (19990601) 35: 4 <408 :: AID-PROT4> 3.0.CO; 2-A. hdl:2027.42/34969. PMID 10382668. Archivovány od originál dne 5. ledna 2013.
- ^ Haynie DT, Xue B (2015). „Superdoména v hierarchii proteinové struktury: případ PTP-C2“. Věda o bílkovinách. 24 (5): 874–82. doi:10.1002 / pro.2664. PMC 4420535. PMID 25694109.
- ^ Bu Z, Callaway DJ (2011). "Proteiny POHYB! Dynamika proteinů a dálková dálnice v buněčné signalizaci". Struktura bílkovin a nemoci. Pokroky v chemii proteinů a strukturní biologii. 83. str. 163–221. doi:10.1016 / B978-0-12-381262-9.00005-7. ISBN 9780123812629. PMID 21570668.
- ^ Fraser JS, Clarkson MW, Degnan SC, Erion R, Kern D, Alber T (prosinec 2009). „Skryté alternativní struktury prolin izomerázy nezbytné pro katalýzu“. Příroda. 462 (7273): 669–673. Bibcode:2009Natur.462..669F. doi:10.1038 / nature08615. PMC 2805857. PMID 19956261.
- ^ Donald, Voet (2011). Biochemie. Voet, Judith G. (4. vydání). Hoboken, NJ: John Wiley & Sons. ISBN 9780470570951. OCLC 690489261.
- ^ Satir, Peter; Søren T. Christensen (26. března 2008). "Struktura a funkce řas savců". Histochemie a buněčná biologie. 129 (6): 687–93. doi:10.1007 / s00418-008-0416-9. PMC 2386530. PMID 18365235. 1432-119X.
- ^ Zhang, Gong; Ignatova, Zoya (1. února 2011). "Skládání při zrodu rodícího se řetězce: koordinace překladu s překládacím skládáním". Aktuální názor na strukturní biologii. 21 (1): 25–31. doi:10.1016 / j.sbi.2010.10.008. ISSN 0959-440X. PMID 21111607.
- ^ Alberts, Bruce; Alexander Johnson; Julian Lewis; Martin Raff; Keith Roberts; Peter Walters (2002). "Tvar a struktura proteinů". Molekulární biologie buňky; Čtvrté vydání. New York a London: Garland Science. ISBN 978-0-8153-3218-3.
- ^ Anfinsen, C. (1972). „Tvorba a stabilizace proteinové struktury“. Biochem. J. 128 (4): 737–49. doi:10.1042 / bj1280737. PMC 1173893. PMID 4565129.
- ^ Jaenicke, R .; Heber, U .; Franks, F .; Chapman, D .; Griffin, Mary C. A .; Hvidt, A .; Cowan, D. A. (1990). "Proteinová struktura a funkce při nízkých teplotách [a diskuse]". Filozofické transakce Královské společnosti v Londýně. Série B, Biologické vědy. 326 (1237): 535–553. doi:10.1098 / rstb.1990.0030. JSTOR 2398703. PMID 1969647.
- ^ Kendrew, J.C .; Bodo, G .; Dintzis, H. M .; Parrish, R. G .; Wyckoff, H .; Phillips, D.C. (1958). „Trojrozměrný model molekuly myoglobinu získaný rentgenovou analýzou“. Příroda. 181 (4610): 662–666. Bibcode:1958Natur.181..662K. doi:10.1038 / 181662a0. PMID 13517261. S2CID 4162786.
- ^ Krimm S, Bandekar J (1986). "Vibrační spektroskopie a konformace peptidů, polypeptidů a proteinů". Pokroky v chemii proteinů, svazek 38. Adv. Protein Chem. Pokroky v chemii proteinů. 38. 181–364. doi:10.1016 / S0065-3233 (08) 60528-8. ISBN 9780120342389. PMID 3541539.
- ^ Lessing, J .; Roy, S .; Reppert, M .; Baer, M .; Marx, D .; Jansen, T.L.C .; Knoester, J .; Tokmakoff, A. (2012). „Identifikace zbytkové struktury v systémech s vnitřní poruchou: 2D IR spektroskopická studie peptidu GVGXPGVG“. J. Am. Chem. Soc. 134 (11): 5032–5035. doi:10.1021 / ja2114135. PMID 22356513.
- ^ Jansen, T.L.C .; Knoester, J. (2008). „Dvojrozměrná infračervená populační přenosová spektroskopie pro zlepšení strukturních markerů proteinů“. Biophys. J. 94 (5): 1818–1825. Bibcode:2008BpJ .... 94,1818J. doi:10.1529 / biophysj.107.118851. PMC 2242754. PMID 17981904.
- ^ Minde DP, Maurice MM, Rüdiger SG (2012). „Stanovení biofyzikální stability proteinu v lyzátech rychlou proteolýzou, FASTpp“. PLOS ONE. 7 (10): e46147. Bibcode:2012PLoSO ... 746147M. doi:10.1371 / journal.pone.0046147. PMC 3463568. PMID 23056252.
- ^ Kumari I, Sandhu P, Ahmed M, Akhter Y (srpen 2017). „Simulace, výzvy a příležitosti molekulární dynamiky: perspektiva biologa“. Curr. Proteinový peptid. Sci. 18 (11): 1163–1179. doi:10.2174/1389203718666170622074741. PMID 28637405.
- ^ A b C d Varadi, Mihaly; Vranken, Wim; Guharoy, Mainak; Tompa, Peter (1. ledna 2015). „Výpočtové přístupy pro odvození funkcí vnitřně neuspořádaných proteinů“. Frontiers in Molecular Biosciences. 2: 45. doi:10.3389 / fmolb.2015.00045. PMC 4525029. PMID 26301226.
- ^ Mittag, Tanja; Marsh, Joseph; Grishaev, Alexander; Orlický, Stephen; Lin, Hong; Sicheri, Frank; Tyers, Mike; Forman-Kay, Julie D. (14. března 2010). „Důsledky struktury / funkce v dynamickém komplexu vnitřně neuspořádaného Sic1 s podjednotkou Cdc4 ubiquitinové ligázy SCF“. Struktura. 18 (4): 494–506. doi:10.1016 / j.str.2010.01.020. ISSN 1878-4186. PMC 2924144. PMID 20399186.
- ^ De Biasio, Alfredo; Ibáñez de Opakua, Alain; Cordeiro, Tiago N .; Villate, Maider; Merino, Nekane; Sibille, Nathalie; Lelli, Moreno; Diercks, Tammo; Bernadó, Pau (18. února 2014). „p15PAF je protein s vnitřní poruchou s nerandomními strukturálními preferencemi v místech interakce s jinými proteiny“. Biofyzikální deník. 106 (4): 865–874. Bibcode:2014BpJ ... 106..865D. doi:10.1016 / j.bpj.2013.12.046. ISSN 1542-0086. PMC 3944474. PMID 24559989.
- ^ Kragelj, Jaka; Palencia, Andrés; Nanao, Max H .; Maurin, Damien; Bouvignies, Guillaume; Blackledge, Martin; Jensen, Malene Ringkjøbing (17. března 2015). "Struktura a dynamika signalizačního komplexu MKK7-JNK". Sborník Národní akademie věd Spojených států amerických. 112 (11): 3409–3414. Bibcode:2015PNAS..112,3409 tis. doi:10.1073 / pnas.1419528112. ISSN 1091-6490. PMC 4371970. PMID 25737554.
- ^ Allison, Jane R .; Rivers, Robert C .; Christodoulou, John C .; Vendruscolo, Michele; Dobson, Christopher M. (25. listopadu 2014). „Vztah mezi přechodnou strukturou v monomerním stavu a agregačními sklony α-synukleinu a β-synukleinu“. Biochemie. 53 (46): 7170–7183. doi:10.1021 / bi5009326. ISSN 1520-4995. PMC 4245978. PMID 25389903.
- ^ Sivakolundu, Sivashankar G .; Bashford, Donald; Kriwacki, Richard W. (11. listopadu 2005). "Neuspořádaný p27Kip1 vykazuje vnitřní strukturu připomínající konformaci vázanou na Cdk2 / cyklin A". Journal of Molecular Biology. 353 (5): 1118–1128. doi:10.1016 / j.jmb.2005.08.074. ISSN 0022-2836. PMID 16214166.
- ^ Laskowski, RA (2011). Msgstr "Databáze struktur proteinů". Mol Biotechnol. 48 (2): 183–98. doi:10.1007 / s12033-010-9372-4. PMID 21225378. S2CID 45184564.
- ^ Murzin, A. G .; Brenner, S.; Hubbard, T.; Chothia, C. (1995). „SCOP: Strukturální klasifikace databáze proteinů pro vyšetřování sekvencí a struktur“ (PDF). Journal of Molecular Biology. 247 (4): 536–540. doi:10.1016 / S0022-2836 (05) 80134-2. PMID 7723011. Archivovány od originál (PDF) dne 26. dubna 2012.
- ^ Orengo, C. A.; Michie, A. D .; Jones, S .; Jones, D. T .; Swindells, M. B .; Thornton, J. M. (1997). "CATH - hierarchická klasifikace struktur proteinových domén". Struktura. 5 (8): 1093–1108. doi:10.1016 / S0969-2126 (97) 00260-8. PMID 9309224.
- ^ A b Pascual-García, A .; Abia, D .; Ortiz, A.R .; Bastolla, U. (2009). „Křížení mezi diskrétním a kontinuálním prostorem proteinové struktury: pohledy na automatickou klasifikaci a sítě proteinových struktur“. PLOS výpočetní biologie. 5 (3): e1000331. Bibcode:2009PLSCB ... 5E0331P. doi:10.1371 / journal.pcbi.1000331. PMC 2654728. PMID 19325884.
- ^ Holm, L; Rosenström, P (červenec 2010). "Dali server: ochrana mapování ve 3D". Výzkum nukleových kyselin. 38 (Problém s webovým serverem): W545–9. doi:10.1093 / nar / gkq366. PMC 2896194. PMID 20457744.
- ^ Zhang Y (2008). „Pokrok a výzvy v predikci struktury proteinů“. Curr Opin Struct Biol. 18 (3): 342–348. doi:10.1016 / j.sbi.2008.02.004. PMC 2680823. PMID 18436442.
Další čtení
- Časová osa 50 let stanovení struktury bílkovin - verze HTML - Národní ústav všeobecných lékařských věd na NIH
externí odkazy
 Média související s Proteinové struktury na Wikimedia Commons
Média související s Proteinové struktury na Wikimedia Commons
