Přeneste RNA - Transfer RNA

| tRNA | |
|---|---|
| Identifikátory | |
| Symbol | t |
| Rfam | RF00005 |
| Další údaje | |
| RNA typ | gen, tRNA |
| PDB struktur | PDBe 3icq, 1asy, 1asz, 1il2, 2tra, 3tra, 486d, 1fir, 1yfg, 3eph, 3epj, 3epk, 3epl, 1efw, 1c0a, 2ake, 2azx, 2dr2, 1f7u, 1f7v, 3foz, 2hgp, 2j00, 2j02, 2ow 2v46, 2v48, 2wdg, 2wdh, 2wdk, 2wdm, 2wh1 |
A přenos RNA (zkráceně tRNA a dříve označované jako sRNA, pro rozpustná RNA[1]) je adaptér molekula složen z RNA, obvykle 76 až 90 nukleotidy v délce,[2] který slouží jako fyzické spojení mezi mRNA a aminokyselina sekvence proteinů. Přenos RNA to dělá tím, že nese aminokyselinu do proteinového syntetického aparátu buňky (ribozom ) podle pokynů komplementárního rozpoznávání 3-nukleotidové sekvence (kodon ) v messenger RNA (mRNA) 3-nukleotidovou sekvencí (antikodon) tRNA. Jako takové jsou tRNA nezbytnou součástí překlad, biologická syntéza nových bílkoviny v souladu s genetický kód.
Přehled
Zatímco specifická nukleotidová sekvence mRNA specifikuje které aminokyseliny jsou inkorporovány do proteinového produktu genu, ze kterého je mRNA transkribována, je úlohou tRNA určit, která sekvence z genetického kódu odpovídá které aminokyselině.[3] MRNA kóduje protein jako řadu souvislých kodonů, z nichž každý je rozpoznáván konkrétní tRNA. Jeden konec tRNA odpovídá genetický kód ve tří-nukleotidové sekvenci zvané antikodon. Antikodon tvoří tři komplementární základní páry s kodon v mRNA během biosyntézy proteinu. Na druhém konci tRNA je kovalentní připojení k aminokyselině, které odpovídá antikodonové sekvenci. Každý typ molekuly tRNA lze připojit pouze k jednomu typu aminokyseliny, takže každý organismus má mnoho typů tRNA. Protože genetický kód obsahuje více kodonů, které specifikují stejnou aminokyselinu, existuje několik molekul tRNA nesoucích různé antikodony, které nesou stejnou aminokyselinu. 3 'konec je katalyzován enzymy zvanými aminoacyl tRNA syntetázy. Během syntézy bílkovin jsou tRNA s připojenými aminokyselinami dodávány do ribozom proteiny zvané faktory prodloužení, které napomáhají ve spojení tRNA s ribozomem, syntéze nového polypeptidu a translokaci (pohybu) ribozomu podél mRNA. Pokud antikodon tRNA odpovídá mRNA, další tRNA již existuje vázán na ribozom přenáší rostoucí polypeptidový řetězec z jeho 3 'konce na aminokyselinu připojenou k 3' konci nově dodávané tRNA, reakci katalyzovanou ribozomem. Může být velké množství jednotlivých nukleotidů v molekule tRNA chemicky upraveno, často tím methylace nebo deamidace. Tyto neobvyklé báze někdy ovlivňují interakci tRNA s ribozomy a někdy se vyskytují v antikodon změnit vlastnosti párování bází.[4]
Struktura
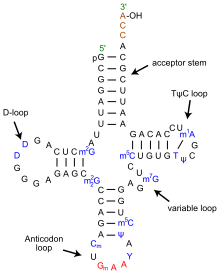

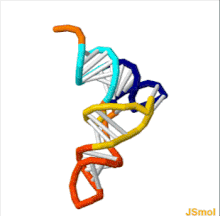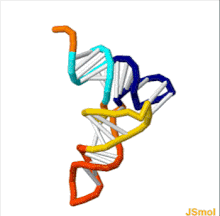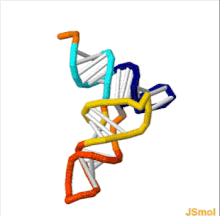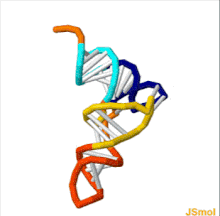
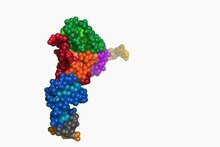
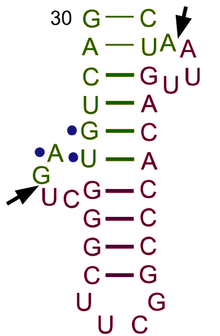
Struktura tRNA může být rozložena na její primární struktura, své sekundární struktura (obvykle vizualizováno jako struktura čtyřlístku), a jeho terciární struktura[6] (všechny tRNA mají podobnou 3D strukturu ve tvaru L, která jim umožňuje zapadnout do P a A stránky ribozom ). Struktura čtyřlístku se stává 3D strukturou ve tvaru L prostřednictvím koaxiálního stohování šroubovic, což je běžné Terciární struktura RNA Délka každého ramene, stejně jako „průměr“ smyčky, v molekule tRNA se u jednotlivých druhů liší.[6][7]Struktura tRNA se skládá z následujících:
- A 5'-terminál fosfát skupina.
- Akceptorový kmen je kmen 7- až 9-bázových párů (bp) vytvořený párováním bází 5'-koncového nukleotidu s 3'-koncovým nukleotidem (který obsahuje CCA 3'-koncovou skupinu použitou k připojení amino kyselina). Obecně se takové 3'-koncové struktury podobné tRNA označují jakogenomové značky '. Akceptorový kmen může obsahovat páry bází jiné než Watson-Crick.[6][8]
- CCA ocas je a cytosin -cytosin-adenin sekvence na 3 'konci molekuly tRNA. Aminokyselina zavedená do tRNA pomocí aminoacyl tRNA syntetázy, tvořit aminoacyl-tRNA, je kovalentně navázán na 3'-hydroxylovou skupinu na CCA konci.[9] Tato sekvence je důležitá pro rozpoznávání tRNA enzymy a kritická při translaci.[10][11] U prokaryot je sekvence CCA transkribována v některých sekvencích tRNA. Ve většině prokaryotických tRNA a eukaryotických tRNA je sekvence CCA přidána během zpracování, a proto se v genu tRNA neobjevuje.[12]
- The D rameno je 4 až 6 bp stopka končící ve smyčce, která často obsahuje dihydrouridin.[6]
- Antikodonové rameno je 5bp dřík, jehož smyčka obsahuje antikodon.[6] Primární struktura tRNA 5'-k-3 'obsahuje antikodon, ale v opačném pořadí, protože pro čtení mRNA od 5'-k-3' je nutná směrovost 3'-k-5 '.
- The T rameno je 4 až 5 bp kmen obsahující sekvenci TΨC, kde Ψ je pseudouridin, upravený uridin.[6]
- Báze, které byly upraveny, zejména uživatelem methylace (např. tRNA (guanin-N7 -) - methyltransferáza ), vyskytují se v několika polohách v celé tRNA. První antikodonová báze nebo kolísavá pozice je někdy upravena na inosin (odvozeno od adeninu), queuosin (odvozeno od guaninu), kyselina uridin-5-oxyoctová (odvozeno od uracilu), 5-methylaminomethyl-2-thiouridin (odvozeno od uracilu) nebo lysidin (odvozeno od cytosinu).[13]
Anticodon
An antikodon[14] je jednotka tří nukleotidy odpovídající třem základnám an mRNA kodon. Každá tRNA má odlišnou antikodonovou tripletovou sekvenci, která může tvořit 3 komplementární základní páry do jednoho nebo více kodonů pro aminokyselinu. Některé antikodony se spárují s více než jedním kodonem kvůli zakolísání párování základen. První nukleotid antikodonu se často nenachází na mRNA: inosin, které mohou vodíková vazba na více než jednu bázi v odpovídající poloze kodonu.[4]:29.3.9 v genetický kód, je běžné, že jedna aminokyselina může být specifikována všemi čtyřmi možnostmi třetí polohy, nebo alespoň oběma pyrimidiny a puriny; například aminokyselina glycin je kódován kodonovými sekvencemi GGU, GGC, GGA a GGG. Další modifikované nukleotidy se mohou také objevit v první antikodonové poloze - někdy známé jako „poloha viklání“ - což má za následek jemné změny genetického kódu, jako například v mitochondrie.[15]Na buňku je zapotřebí 61 typů tRNA, aby byla zajištěna vzájemná korespondence mezi molekulami tRNA a kodony, které specifikují aminokyseliny, protože existuje 61 sense kodonů standardního genetického kódu. Mnoho buněk však má méně než 61 typů tRNA, protože zakřivená báze je schopná vázat se na několik, i když ne nutně, všech kodonů, které specifikují konkrétní aminokyselinu. K překladu všech 61 sense kodonů je zapotřebí alespoň 31 tRNA.[3][16]
Aminoacylace
Aminoacylace je proces přidání aminoacylové skupiny ke sloučenině. Kovalentně propojuje aminokyselina na CCA 3 'konec molekuly tRNA. Každá tRNA je aminoacylována (nebo účtováno) se specifickou aminokyselinou pomocí aminoacyl tRNA syntetáza. Normálně pro každou aminokyselinu existuje jedna aminoacyl tRNA syntetáza, a to navzdory skutečnosti, že může existovat více než jedna tRNA a více než jeden antikodon pro aminokyselinu. Rozpoznání vhodné tRNA pomocí syntetáz není zprostředkováno pouze antikodonem a akceptorový kmen často hraje významnou roli.[17]Reakce:
U některých organismů může chybět jedna nebo více aminofosfát-tRNA syntetáz. To vede k nabíjení tRNA chemicky příbuznou aminokyselinou a použitím enzymu nebo enzymů je tRNA upravena tak, aby byla správně nabitá. Například, Helicobacter pylori chybí glutaminyl tRNA syntetáza. Glutamát tRNA syntetáza tedy nabíjí tRNA-glutamin (tRNA-Gln) glutamát. Amidotransferáza poté převádí kyselý postranní řetězec glutamátu na amid a vytváří správně nabitý gln-tRNA-Gln.
Vazba na ribozom
The ribozom má tři vazebná místa pro molekuly tRNA, která překlenují prostor mezi těmito dvěma ribozomální podjednotky: A (aminoacyl),[19] P (peptidyl), a E (výstupní) stránky. Kromě toho má ribozom dvě další místa pro vazbu tRNA, která se používají během mRNA dekódování nebo během zahájení proteosyntéza. Jedná se o web T (pojmenovaný součinitel prodloužení Tu ) a I web (zahájení).[20][21] Podle konvence jsou vazebná místa pro tRNA označena místem na malá ribozomální podjednotka uveden jako první a web na webu velká ribozomální podjednotka uvedené na druhém místě. Například web A je často psán A / A, web P, P / P a web E, E / E.[20] Vazebné proteiny jako L27, L2, L14, L15, L16 na A- a P- místech byly stanoveny afinitním značením A. P. Czernilofsky et al. (Proc. Natl. Acad. Sci, USA, str. 230–234, 1974).
Jakmile je iniciace translace dokončena, první aminoacyl tRNA je lokalizována v P / P místě, připraveném pro elongační cyklus popsaný níže. Během translační elongace se tRNA nejprve váže na ribozom jako součást komplexu s elongačním faktorem Tu (EF-Tu ) nebo jeho eukaryotický (eEF-1 ) nebo archaeal protějšek. Toto počáteční vazebné místo pro tRNA se nazývá A / T místo. V místě A / T je polovina místa A umístěna v malá ribozomální podjednotka kde se nachází dekódovací místo mRNA. Místo dekódování mRNA je místo, kde mRNA kodon se čte během překladu. Polovina T-místa sídlí hlavně na velká ribozomální podjednotka kde EF-Tu nebo eEF-1 interaguje s ribozomem. Po dokončení dekódování mRNA je aminoacyl-tRNA navázána na místo A / A a je připravena na další peptidová vazba aby se vytvořil na svou připojenou aminokyselinu. Peptidyl-tRNA, která přenáší rostoucí polypeptid na aminoacyl-tRNA vázanou v místě A / A, je vázána v místě P / P. Jakmile se vytvoří peptidová vazba, tRNA v místě P / P je deacylovaná nebo má a konec 3 'zdarma a tRNA v místě A / A nese rostoucí polypeptidový řetězec. Aby byl umožněn další elongační cyklus, tRNA se poté pohybují hybridními vazebnými místy A / P a P / E, než dokončí cyklus a pobývají v místech P / P a E / E. Jakmile se A / A a P / P tRNA přesunou na místa P / P a E / E, mRNA se také přesunula o jednu kodon a místo A / T je prázdné, připravené na další kolo dekódování mRNA. TRNA vázaná v E / E místě poté opouští ribozom.
Místo P / I je ve skutečnosti první, které se váže na aminoacyl tRNA, která je dodávána iniciačním faktorem zvaným IF2 v bakteriích.[21] Existence stránky P / I je však u eukaryotických nebo archaealních ribozomy dosud nebylo potvrzeno. Protein L27 v místě P byl stanoven afinitním značením E. Collatz a A. P. Czernilofsky (FEBS Lett., Sv. 63, s. 283–286, 1976).
geny tRNA
Organismy se liší v počtu tRNA geny v jejich genom. Například hlístice červ C. elegans, běžně používaný modelový organismus v České republice genetika studií, má 29 647 [22] geny v jeho jaderný genom, z toho 620 kóduje tRNA.[23][24] Nadějné droždí Saccharomyces cerevisiae má ve svém genomu 275 genů tRNA.
V lidském genomu, který má podle odhadů z ledna 2013 asi 20 848 genů kódujících proteiny [25] celkem existuje 497 jaderných genů kódujících cytoplazmatické molekuly tRNA a 324 odvozených od tRNA pseudogeny —TRNA geny považované za nefunkční[26] (i když bylo prokázáno, že pseudo tRNA jsou součástí odolnost proti antibiotikům v bakteriích).[27] Regiony v jaderné oblasti chromozomy Byly také identifikovány velmi podobné sekvence jako geny mitochondriální tRNA (podobná tRNA).[28] Tyto dvojice tRNA jsou také považovány za součást nukleární mitochondriální DNA (geny přenesené z mitochondrií do jádra).[28][29]
Stejně jako u všech eukaryot je jich 22 mitochondriální geny tRNA[30] u lidí. Mutace v některých z těchto genů byly spojeny s těžkými nemocemi, jako je MELAS syndrom.
Geny pro cytoplazmatickou tRNA lze rozdělit do 49 rodin podle jejich antikodonových rysů. Tyto geny se nacházejí na všech chromozomech, kromě chromozomu 22 a Y. Je pozorována vysoká shlukování na 6p (140 genů tRNA), stejně jako na 1 chromozomu.[26]
The HGNC, ve spolupráci s Genomic tRNA Database (GtRNAdb ) a odborníci v oboru schválili jedinečná jména pro lidské geny, které kódují tRNA.
Vývoj
Horní polovina tRNA (skládající se z T ramene a akceptorového kmene s 5'-koncovou fosfátovou skupinou a 3'-koncovou CCA skupinou) a spodní polovina (skládající se z D ramene a antikodonového ramene) jsou ve struktuře nezávislé jednotky stejně jako ve funkci. Horní polovina se mohla vyvinout jako první, včetně 3'-terminální genomové značky, která původně mohla značit molekuly podobné tRNA pro replikaci na počátku Svět RNA. Spodní polovina se mohla vyvinout později jako expanze, např. jak syntéza proteinů začala ve světě RNA a proměnila ji ve svět ribonukleoproteinů (Svět RNP ). Tento navrhovaný scénář se nazývá hypotéza genomové značky. Ve skutečnosti mají tRNA a agregáty podobné tRNA důležitý katalytický vliv (tj ribozymy ) na replikaci ještě dnes. Tyto role lze považovat zamolekulární (nebo chemické) fosilie „světa RNA.[31]
Obsah genomové tRNA je rozlišovacím znakem genomů mezi biologickými doménami života: Archaea představuje nejjednodušší situaci, pokud jde o obsah genomové tRNA, s jednotným počtem kopií genů, bakterie mají střední situaci a Eukarya představuje nejsložitější situaci.[32] Eukarya představuje nejen větší obsah genu pro tRNA než ostatní dvě království, ale také velké rozdíly v počtu kopií genů mezi různými izoacceptory a zdá se, že tato složitost je způsobena duplikací genů tRNA a změnami antikodonové specificity.[Citace je zapotřebí ].
Vývoj počtu kopií genu tRNA u různých druhů byl spojen s výskytem specifických enzymů pro modifikaci tRNA (uridinmethyltransferázy v bakteriích a adenosindeaminázy v Eukarya), které zvyšují dekódovací kapacitu dané tRNA.[32] Například tRNAAla kóduje čtyři různé izoakceptory tRNA (AGC, UGC, GGC a CGC). V Eukaryi jsou AGC isoacceptory extrémně obohaceny počtem kopií genů ve srovnání se zbytkem isoacceptorů, a to korelovalo s jeho A-to-I modifikací jeho vlnité báze. Stejný trend byl prokázán u většiny aminokyselin eukaryálních druhů. Účinek těchto dvou modifikací tRNA je také patrný v předpětí použití kodonů. Zdá se, že vysoce exprimované geny jsou obohaceny v kodonech, které používají výhradně kodony, které budou dekódovány těmito modifikovanými tRNA, což naznačuje možnou roli těchto kodonů - a následně těchto modifikací tRNA - v účinnosti translace.[32]
fragmenty odvozené od tRNA
Fragmenty odvozené od tRNA (nebo tRF) jsou krátké molekuly, které se objevují po štěpení zralých tRNA nebo prekurzorového transkriptu.[33][34][35][36] Cytoplazmatické i mitochondriální tRNA mohou produkovat fragmenty.[Citace je zapotřebí ] Existují nejméně čtyři strukturní typy tRF, o nichž se předpokládá, že pocházejí ze zralých tRNA, včetně relativně dlouhých polovin tRNA a krátkých 5’-tRF, 3’-tRF a i-tRF.[33][37] Prekurzorová tRNA může být štěpena za vzniku molekul z 5 'vedoucí nebo 3' stopové sekvence. Enzymy štěpení zahrnují Angiogenin, Dicer, RNase Z a RNase P.[33][34] Zejména v případě angiogeninu mají tRF na svém 3 'konci charakteristicky neobvyklý cyklický fosfát a na 5' konci hydroxylovou skupinu.[38] Zdá se, že tRF hrají roli v Interference RNA, konkrétně při potlačení retrovirů a retrotranspozonů, které používají tRNA jako primer pro replikaci. Poloviční tRNA štěpené angiogenin jsou také známé jako tiRNA. Biogeneze menších fragmentů, včetně těch, které fungují jako piRNA, jsou méně chápány.[39]
tRF mají více závislostí a rolí; jako například vykazování významných změn mezi pohlavími, mezi rasami a stavem nemoci.[Citace je zapotřebí ] Funkčně je lze načíst na Ago a působit cestou RNAi,[35][37][40] podílet se na tvorbě stresových granulí,[41] vytěsňují mRNA z proteinů vázajících RNA[42] nebo potlačit překlad.[43] Na úrovni systému nebo organismu mají čtyři typy tRF různorodé spektrum aktivit. Funkčně jsou tRF spojeny s virovou infekcí,[44] rakovina,[37] proliferace buněk [38] a také s epigenetickou transgenerační regulací metabolismu.[45]
tRF nejsou omezeny na člověka a bylo prokázáno, že existují v mnoha organismech.[37][46][47][48]
Pro ty, kteří se chtějí dozvědět více o tRF, jsou k dispozici dva online nástroje: rámec pro interaktivní průzkum mitochondrial a njasný tFragmenty RNA (MINTbase )[49] a relační databáze TRansfer RSouvisející s NA Fřádky (tRFdb ).[50] MINTbase také poskytuje schéma pojmenování pojmenovaných volaných tRF tRF poznávací značky (nebo MINTcodes), které jsou nezávislé na genomu; schéma komprimuje sekvenci RNA do kratšího řetězce.
Vytvořené tRNA
K začlenění se používají umělé supresorové elongátorové tRNA nepřirozené aminokyseliny v nesmyslných kodonech umístěných v kódující sekvenci genu. Upravené iniciátorové tRNA (tRNAfMet2 s antikodonem CUA kódovaným metY gen) byly použity k zahájení překlad v jantaru stop kodon UAG. Tento typ upravené tRNA se nazývá a potlačovač nesmyslů tRNA, protože potlačuje signál zastavení translace, který se normálně vyskytuje v kodonech UAG. Žlutý iniciátor tRNA vloží methionin[51] a glutamin[52] v kodonech UAG předchází silný Sekvence Shine-Dalgarno. Vyšetřování jantarového iniciátoru tRNA ukázalo, že je ortogonální vůči běžnému startovacímu kodonu AUG, který nevykazuje žádné detekovatelné iniciační události translace mimo cíl v genomicky překódovaném E-coli kmen.[51]
biogeneze tRNA
v eukaryotický buňky, tRNA jsou přepsal podle RNA polymeráza III jako pre-tRNA v jádře.[53]RNA polymeráza III rozpoznává dvě vysoce konzervované downstream promotorové sekvence: 5 'intragenní kontrolní oblast (5'-ICR, D-kontrolní oblast nebo A box) a 3'-ICR (T-kontrolní oblast nebo B box) uvnitř tRNA geny.[2][54][55]První promotor začíná na +8 zralých tRNA a druhý promotor je umístěn 30–60 nukleotidů za prvním promotorem. Přepis končí po úseku čtyř nebo více thymidiny.[2][55]
Pre-tRNA procházejí uvnitř jádra rozsáhlými modifikacemi. Některé pre-tRNA obsahují introny které jsou spojeny nebo rozřezány za vzniku funkční molekuly tRNA;[56] v bakteriíchspojit, zatímco u eukaryot a archaea jsou odstraněny sestřihem tRNA endonukleázy.[57] Eukaryotická pre-tRNA obsahuje motiv struktury bulge-helix-bulge (BHB), který je důležitý pro rozpoznání a přesné sestřih intronu tRNA endonukleázami.[58] Tato poloha a struktura motivu jsou evolučně zachovány. Některé organismy, jako jsou jednobuněčné řasy, však mají nekanonickou polohu motivu BHB, stejně jako 5'- a 3'-konce sestřižené intronové sekvence.[58]5 'sekvence je odstraněna pomocí RNáza P,[59] zatímco 3 'konec je odstraněn pomocí tRNase Z enzym.[60]Pozoruhodná výjimka je v archaeon Nanoarchaeum equitans, který nemá enzym RNáza P a má promotor umístěný tak, že transkripce začíná na 5 'konci zralé tRNA.[61]Nestratlovaný 3 'CCA ocas je přidán a nukleotidyltransferáza.[62]Předtím, než jsou tRNA exportováno do cytoplazma podle Los1 /Xpo-t,[63][64] tRNA jsou aminoacylovaný.[65]Pořadí událostí zpracování není zachováno. Například v droždí, sestřih se neprovádí v jádru, ale na cytoplazmatické straně mitochondriální membrány.[66]
Dějiny
O existenci tRNA se poprvé vyslovila hypotéza Francis Crick, na základě předpokladu, že musí existovat molekula adaptéru schopná zprostředkovat překlad RNA abecedy do proteinové abecedy. Paul C. Zámečník a Mahlon Hoagland objevená tRNA [67] Na počátku 60. let provedl významný výzkum struktury Alex Rich a Don Caspar, dva vědci v Bostonu, skupina Jacques Fresco v Univerzita Princeton a a Spojené království skupina v King's College London.[68] V roce 1965 Robert W. Holley z Cornell University uvedla primární strukturu a navrhla tři sekundární struktury.[69] tRNA byla nejprve krystalizována v Madisonu ve Wisconsinu Robertem Bockem.[70] Strukturu čtyřlístku zjistilo několik dalších studií v následujících letech[71] a byl nakonec potvrzen pomocí Rentgenová krystalografie studie v roce 1974. Dvě nezávislé skupiny, Kim Sung-Hou pracuje pod Alexander Rich a britská skupina v čele s Aaron Klug, publikoval stejné krystalografické nálezy do jednoho roku.[72][73]
Viz také
Reference
- ^ Plescia OJ, Palczuk NC, Cora-Figueroa E, Mukherjee A, Braun W (říjen 1965). "Produkce protilátek proti rozpustné RNA (sRNA)". Sborník Národní akademie věd Spojených států amerických. 54 (4): 1281–1285. Bibcode:1965PNAS ... 54.1281P. doi:10.1073 / pnas.54.4.1281. PMC 219862. PMID 5219832.
- ^ A b C Sharp SJ, Schaack J, Cooley L, Burke DJ, Söll D (1985). "Struktura a transkripce eukaryotických genů tRNA". CRC kritické recenze v biochemii. 19 (2): 107–144. doi:10.3109/10409238509082541. PMID 3905254.
- ^ A b Crick FH (prosinec 1968). "Původ genetického kódu". Journal of Molecular Biology. 38 (3): 367–379. doi:10.1016/0022-2836(68)90392-6. PMID 4887876.
- ^ A b Stryer L, Berg JM, Tymoczko JL (2002). Biochemie (5. vydání). San Francisco: W. H. Freeman. ISBN 978-0-7167-4955-4.
- ^ RNA% 28tRNA% 29 "Transfer RNA (tRNA)" Šek
| url =hodnota (Pomoc). Proteopedia.org. Citováno 7. listopadu 2018. - ^ A b C d E F Itoh Y, Sekine S, Suetsugu S, Yokoyama S (červenec 2013). "Terciární struktura bakteriální selenocysteinové tRNA". Výzkum nukleových kyselin. 41 (13): 6729–6738. doi:10.1093 / nar / gkt321. PMC 3711452. PMID 23649835.
- ^ Goodenbour JM, Pan T (29. října 2006). „Rozmanitost genů tRNA u eukaryotů“. Výzkum nukleových kyselin. 34 (21): 6137–6146. doi:10.1093 / nar / gkl725. PMC 1693877. PMID 17088292.
- ^ Jahn M, Rogers MJ, Söll D (červenec 1991). „Antikodonové a akceptorové kmenové nukleotidy v tRNA (Gln) jsou hlavními rozpoznávacími prvky pro glutaminyl-tRNA syntetázu z E. coli“. Příroda. 352 (6332): 258–260. Bibcode:1991Natur.352..258J. doi:10.1038 / 352258a0. PMID 1857423. S2CID 4263705.
- ^ Ibba M, Soll D (červen 2000). "Aminoacyl-tRNA syntéza". Roční přehled biochemie. 69 (1): 617–650. doi:10,1146 / annurev.biochem. 69.1.617. PMID 10966471.
- ^ Sprinzl M, Cramer F (1979). "Konec -C-C-A tRNA a jeho role v biosyntéze bílkovin". Pokrok ve výzkumu nukleových kyselin a molekulární biologie. 22: 1–69. doi:10.1016 / s0079-6603 (08) 60798-9. ISBN 978-0-12-540022-0. PMID 392600.
- ^ Green R, Noller HF (1997). "Ribozomy a překlad". Roční přehled biochemie. 66: 679–716. doi:10,1146 / annurev.biochem. 66.1.679. PMID 9242921.
- ^ Aebi M, Kirchner G, Chen JY, Vijayraghavan U, Jacobson A, Martin NC, Abelson J a kol. (Září 1990). „Izolace teplotně citlivého mutanta se změněnou tRNA nukleotidyltransferázou a klonování genu kódujícího tRNA nukleotidyltransferázu do kvasinek Saccharomyces cerevisiae“. The Journal of Biological Chemistry. 265 (27): 16216–16220. PMID 2204621.
- ^ McCloskey JA, Nishimura S (listopad 1977). "Modifikované nukleosidy v přenosové RNA". Účty chemického výzkumu. 10 (11): 403–410. doi:10.1021 / ar50119a004.
- ^ Felsenfeld G, Cantoni GL (květen 1964). „Využití studií tepelné denaturace ke zkoumání základní sekvence kvasinkové serinové sRNA“. Sborník Národní akademie věd Spojených států amerických. 51 (5): 818–826. Bibcode:1964PNAS ... 51..818F. doi:10.1073 / pnas.51.5.818. PMC 300168. PMID 14172997.
- ^ Suzuki T, Suzuki T (červen 2014). „Kompletní krajina posttranskripčních modifikací v savčích mitochondriálních tRNA“. Výzkum nukleových kyselin. 42 (11): 7346–7357. doi:10.1093 / nar / gku390. PMC 4066797. PMID 24831542.
- ^ Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). Molekulární biologie buňky. WH Freeman: New York. 5. vyd.[ISBN chybí ][stránka potřebná ]
- ^ Schimmel P, Giegé R, Moras D, Yokoyama S (říjen 1993). „Provozní kód RNA pro aminokyseliny a možný vztah ke genetickému kódu“. Sborník Národní akademie věd Spojených států amerických. 90 (19): 8763–8768. Bibcode:1993PNAS ... 90.8763S. doi:10.1073 / pnas.90.19.8763. PMC 47440. PMID 7692438.
- ^ Dunkle JA, Wang L, Feldman MB, Pulk A, Chen VB, Kapral GJ, Noeske J, Richardson JS, Blanchard SC, Cate JH (květen 2011). "Struktury bakteriálního ribozomu v klasickém a hybridním stavu vazby tRNA". Věda. 332 (6032): 981–984. Bibcode:2011Sci ... 332..981D. doi:10.1126 / science.1202692. PMC 3176341. PMID 21596992.
- ^ Konevega AL, Soboleva NG, Machno VI, Semenkov YP, Wintermeyer W, Rodnina MV, Katunin VI (leden 2004). „Purinové báze v poloze 37 tRNA stabilizují interakci kodon-antikodon v ribozomálním místě A stohováním a interakcemi závislými na Mg2 +“. RNA. 10 (1): 90–101. doi:10,1261 / rna.5142404. PMC 1370521. PMID 14681588.
- ^ A b Agirrezabala X, Frank J (srpen 2009). „Prodloužení v translaci jako dynamická interakce mezi ribozomy, tRNA a faktory prodloužení EF-G a EF-Tu“. Čtvrtletní recenze biofyziky. 42 (3): 159–200. doi:10.1017 / S0033583509990060. PMC 2832932. PMID 20025795.
- ^ A b Allen GS, Zavialov A, Gursky R, Ehrenberg M, Frank J (červen 2005). „Kryo-EM struktura komplexu iniciace translace z Escherichia coli“. Buňka. 121 (5): 703–712. doi:10.1016 / j.cell.2005.03.023. PMID 15935757. S2CID 16146867.
- ^ Web WormBase, http://www.wormbase.org, vydání WS187, datum 25. ledna 2008.
- ^ Spieth J, Lawson D (leden 2006). "Přehled genové struktury". WormBook: 1–10. doi:10.1895 / wormbook.1.65.1. PMC 4781370. PMID 18023127.
- ^ Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC. (2004). Genetika: Od genů k genomům 2. vyd. McGraw-Hill: New York. p. 264.
- ^ Vydání souboru 70 - leden 2013 http://www.ensembl.org/Homo_sapiens/Info/StatsTable?db=core Archivováno 2013-12-15 na Wayback Machine
- ^ A b Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J a kol. (Mezinárodní konsorcium pro sekvenování lidského genomu) (únor 2001). „Počáteční sekvenování a analýza lidského genomu“ (PDF). Příroda. 409 (6822): 860–921. Bibcode:2001 Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Rogers TE, Ataide SF, Dare K, Katz A, Seveau S, Roy H, Ibba M (2012). „Pseudo-tRNA moduluje rezistenci na Bacillus cereus vůči antibiotikům“. PLOS ONE. 7 (7): e41248. Bibcode:2012PLoSO ... 741248R. doi:10.1371 / journal.pone.0041248. PMC 3399842. PMID 22815980.
- ^ A b Telonis AG, Loher P, Kirino Y, Rigoutsos I (2014). „Jaderná a mitochondriální podoba tRNA v lidském genomu“. Frontiers in Genetics. 5: 344. doi:10.3389 / fgene.2014.00344. PMC 4189335. PMID 25339973.
- ^ Ramos A, Barbena E, Mateiu L, del Mar González M, Mairal Q, Lima M, Montiel R, Aluja MP, Santos C a kol. (Listopad 2011). „Nukleární inzerce mitochondriálního původu: aktualizace databáze a užitečnost ve studiích rakoviny“. Mitochondrie. 11 (6): 946–953. doi:10.1016 / j.mito.2011.08.009. PMID 21907832.
- ^ Tamtéž. p. 529.
- ^ Nancy Maizels a Alan M. Weiner: Hypotéza genomové značky - Co nám molekulární fosilie říkají o vývoji tRNA, in: The RNA World, Second Edition. 1999 Cold Spring Harbor Laboratory Press ISBN 978-0-87969-561-3/99, PDF
- ^ A b C Novoa EM, Pavon-Eternod M, Pan T, Ribas de Pouplana L (březen 2012). „Role pro modifikace tRNA ve struktuře genomu a použití kodonů“. Buňka. 149 (1): 202–213. doi:10.1016 / j.cell.2012.01.050. PMID 22464330. S2CID 16487609.
- ^ A b C Gebetsberger J, Polacek N (prosinec 2013). „Krájení tRNA pro zvýšení funkční rozmanitosti ncRNA“. RNA Biology. 10 (12): 1798–1806. doi:10,4161 / rna.27177. PMC 3917982. PMID 24351723.
- ^ A b Shigematsu M, Honda S, Kirino Y (2014). „Transfer RNA jako zdroj malé funkční RNA“. Journal of Molecular Biology and Molecular Imaging. 1 (2): 8. PMC 4572697. PMID 26389128.
- ^ A b Sobala A, Hutvagner G (2011). „Přenos fragmentů odvozených z RNA: počátky, zpracování a funkce“ (PDF). Wiley Interdisciplinary Reviews: RNA. 2 (6): 853–862. doi:10,1002 / wrna.96. hdl:10453/18187. PMID 21976287. S2CID 206554146.
- ^ Keam SP, Hutvagner G (listopad 2015). „Fragmenty odvozené od tRNA (tRF): vznikající nové role pro starou RNA v regulaci genové exprese“. Život. 5 (4): 1638–1651. doi:10,3390 / život5041638. PMC 4695841. PMID 26703738.
- ^ A b C d Kumar P, Anaya J, Mudunuri SB, Dutta A (říjen 2014). „Metaanalýza fragmentů RNA odvozených od tRNA ukazuje, že jsou evolučně konzervované a asociují se s proteiny AGO, aby rozpoznaly specifické cíle RNA“. Biologie BMC. 12: 78. doi:10.1186 / s12915-014-0078-0. PMC 4203973. PMID 25270025.
- ^ A b Honda S, Loher P, Shigematsu M, Palazzo JP, Suzuki R, Imoto I, Rigoutsos I, Kirino Y (červenec 2015). „Poloviny tRNA závislé na pohlavních hormonech zvyšují proliferaci buněk u rakoviny prsu a prostaty“. Sborník Národní akademie věd Spojených států amerických. 112 (29): E3816 – E3825. Bibcode:2015PNAS..112E3816H. doi:10.1073 / pnas.1510077112. PMC 4517238. PMID 26124144.
- ^ Schorn, AJ; Martienssen, R (říjen 2018). „Tie-Break: Host a Retrotransposons Play tRNA“. Trendy v buněčné biologii. 28 (10): 793–806. doi:10.1016 / j.tcb.2018.05.006. PMC 6520983. PMID 29934075.
- ^ Shigematsu M, Kirino Y (2015). „Krátká nekódující RNA odvozená od tRNA jako interakční partneři proteinů Argonaute“. Regulace genů a biologie systémů. 9: 27–33. doi:10,4137 / GRSB.S29411. PMC 4567038. PMID 26401098.
- ^ Emara MM, Ivanov P, Hickman T, Dawra N, Tisdale S, Kedersha N, Hu GF, Anderson P (duben 2010). „Angiogeninem indukovaná tRNA stresem indukovaná RNA podporuje stresem indukované sestavení stresových granulí“. The Journal of Biological Chemistry. 285 (14): 10959–10968. doi:10,1074 / jbc.M109.077560. PMC 2856301. PMID 20129916.
- ^ Goodarzi H, Liu X, Nguyen HC, Zhang S, Fish L, Tavazoie SF (květen 2015). „Endogenní fragmenty odvozené z tRNA potlačují progresi rakoviny prsu pomocí YBX1 výtlaku“. Buňka. 161 (4): 790–802. doi:10.1016 / j.cell.2015.02.053. PMC 4457382. PMID 25957686.
- ^ Ivanov P, Emara MM, Villen J, Gygi SP, Anderson P (srpen 2011). „Angiogeninem indukované fragmenty tRNA inhibují iniciaci translace“. Molekulární buňka. 43 (4): 613–623. doi:10.1016 / j.molcel.2011.06.022. PMC 3160621. PMID 21855800.
- ^ Selitsky SR, Baran-Gale J, Honda M, Yamane D, Masaki T, Fannin EE, Guerra B, Shirasaki T, Shimakami T, Kaneko S, Lanford RE, Lemon SM, Sethupathy P (leden 2015). „Malé RNA odvozené od tRNA jsou u chronické hepatitidy B a C zvýšené a hojnější než mikroRNA.“. Vědecké zprávy. 5: 7675. Bibcode:2015NatSR ... 5E7675S. doi:10.1038 / srep07675. PMC 4286764. PMID 25567797.
- ^ Sharma U, Conine CC, Shea JM, Boskovic A, Derr AG, Bing XY, Belleannee C, Kucukural A, Serra RW, Sun F, Song L, Carone BR, Ricci EP, Li XZ, Fauquier L, Moore MJ, Sullivan R , Mello CC, Garber M, Rando OJ (leden 2016). „Biogeneze a funkce fragmentů tRNA během zrání a oplodnění spermií u savců“. Věda. 351 (6271): 391–396. Bibcode:2016Sci ... 351..391S. doi:10.1126 / science.aad6780. PMC 4888079. PMID 26721685.
- ^ Casas E, Cai G, Neill JD (2015). "Charakterizace cirkulujících přenosových RNA fragmentů RNA u skotu". Frontiers in Genetics. 6: 271. doi:10.3389 / fgene.2015.00271. PMC 4547532. PMID 26379699.
- ^ Hirose Y, Ikeda KT, Noro E, Hiraoka K, Tomita M, Kanai A (červenec 2015). „Přesné mapování a dynamika fragmentů odvozených od tRNA (tRF) při vývoji Triops cancriformis (krevetka obecná)“. Genetika BMC. 16: 83. doi:10.1186 / s12863-015-0245-5. PMC 4501094. PMID 26168920.
- ^ Karaiskos S, Naqvi AS, Swanson KE, Grigoriev A (září 2015). „Věkem řízená modulace fragmentů odvozených od tRNA v Drosophile a jejich potenciálních cílech“. Biology Direct. 10: 51. doi:10.1186 / s13062-015-0081-6. PMC 4572633. PMID 26374501.
- ^ Pliatsika V, Loher P, Telonis AG, Rigoutsos I (srpen 2016). „MINTbase: rámec pro interaktivní průzkum mitochondriálních a jaderných fragmentů tRNA“. Bioinformatika. 32 (16): 2481–2489. doi:10.1093 / bioinformatika / btw194. PMC 4978933. PMID 27153631.
- ^ Kumar P, Mudunuri SB, Anaya J, Dutta A (leden 2015). „tRFdb: databáze pro přenos fragmentů RNA“. Výzkum nukleových kyselin. 43 (Problém s databází): D141-5. doi:10.1093 / nar / gku1138. PMC 4383946. PMID 25392422.
- ^ A b Vincent RM, Wright BW, Jaschke PR (duben 2019). „Měření jantarového iniciátoru tRNA ortogonality v genomicky překódovaném organismu“. ACS Syntetická biologie. 8 (4): 675–685. doi:10.1021 / acssynbio.9b00021. PMID 30856316.
- ^ Govindan A, Miryala S, Mondal S, Varshney U (listopad 2018). „Vývoj testovacích systémů pro dekódování jantarových kodonů v krocích iniciace a prodloužení u mykobakterií“. Journal of Bacteriology. 200 (22). doi:10.1128 / jb.00372-18. PMC 6199473. PMID 30181124.
- ^ White RJ (březen 1997). „Regulace RNA polymeráz I a III proteinem retinoblastomu: mechanismus pro kontrolu růstu?“. Trendy v biochemických vědách. 22 (3): 77–80. doi:10.1016 / S0968-0004 (96) 10067-0. PMID 9066256.
- ^ Sharp S, Dingermann T, Söll D (září 1982). „Minimální intragenní sekvence potřebné pro podporu transkripce genu eukaryotické tRNA“. Výzkum nukleových kyselin. 10 (18): 5393–5406. doi:10.1093 / nar / 10.18.5393. PMC 320884. PMID 6924209.
- ^ A b Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A (prosinec 2007). "Expandující transkriptom RNA RNA polymerázy III". Trendy v genetice. 23 (12): 614–622. doi:10.1016 / j.tig.2007.09.001. hdl:11381/1706964. PMID 17977614.
- ^ Tocchini-Valentini GD, Fruscoloni P, Tocchini-Valentini GP (prosinec 2009). "Zpracování pretRNA obsahující více intronů". Sborník Národní akademie věd Spojených států amerických. 106 (48): 20246–20251. Bibcode:2009PNAS..10620246T. doi:10.1073 / pnas.0911658106. PMC 2787110. PMID 19910528.
- ^ Abelson J, Trotta CR, Li H (květen 1998). "sestřih tRNA". The Journal of Biological Chemistry. 273 (21): 12685–12688. doi:10.1074 / jbc.273.21.12685. PMID 9582290.
- ^ A b Soma A (2014). „Cirkulárně permutované geny tRNA: jejich exprese a důsledky pro jejich fyziologický význam a vývoj“. Frontiers in Genetics. 5: 63. doi:10.3389 / fgene.2014.00063. PMC 3978253. PMID 24744771.
- ^ Frank DN, Pace NR (1998). „Ribonukleáza P: jednota a rozmanitost v ribozymu zpracovávajícím tRNA“. Roční přehled biochemie. 67 (1): 153–180. doi:10.1146 / annurev.biochem.67.1.153. PMID 9759486.
- ^ Ceballos M, Vioque A (2007). "tRNase Z". Proteinové a peptidové dopisy. 14 (2): 137–145. doi:10.2174/092986607779816050. PMID 17305600.
- ^ Randau L, Schröder I, Söll D (květen 2008). "Život bez RNázy P". Příroda. 453 (7191): 120–123. Bibcode:2008 Natur.453..120R. doi:10.1038 / nature06833. PMID 18451863. S2CID 3103527.
- ^ Weiner AM (říjen 2004). "maturace tRNA: polymerace RNA bez templátu nukleové kyseliny". Aktuální biologie. 14 (20): R883-5. doi:10.1016 / j.cub.2004.09.069. PMID 15498478.
- ^ Kutay U, Lipowsky G, Izaurralde E, Bischoff FR, Schwarzmaier P, Hartmann E, Görlich D (únor 1998). "Identifikace tRNA specifického jaderného exportního receptoru". Molekulární buňka. 1 (3): 359–369. doi:10.1016 / S1097-2765 (00) 80036-2. PMID 9660920.
- ^ Arts GJ, Fornerod M, Mattaj IW (březen 1998). "Identifikace nukleárního exportního receptoru pro tRNA". Aktuální biologie. 8 (6): 305–314. doi:10.1016 / S0960-9822 (98) 70130-7. PMID 9512417. S2CID 17803674.
- ^ Arts GJ, Kuersten S, Romby P, Ehresmann B, Mattaj IW (prosinec 1998). „Role exportinu-t při selektivním jaderném exportu zralých tRNA“. Časopis EMBO. 17 (24): 7430–7441. doi:10.1093 / emboj / 17.24.7430. PMC 1171087. PMID 9857198.
- ^ Yoshihisa T, Yunoki-Esaki K, Ohshima C, Tanaka N, Endo T (srpen 2003). „Možnost spojování cytoplazmatické pre-tRNA: kvasinková tRNA sestřihová endonukleáza se lokalizuje hlavně na mitochondriích“. Molekulární biologie buňky. 14 (8): 3266–3279. doi:10,1091 / mbc.E02-11-0757. PMC 181566. PMID 12925762.
- ^ http://www.jbc.org/content/280/40/e37
- ^ Clark BF (říjen 2006). „Krystalová struktura tRNA“ (PDF). Journal of Biosciences. 31 (4): 453–457. doi:10.1007 / BF02705184. PMID 17206065. S2CID 19558731.
- ^ Holley RW, Apgar J, Everett GA, Madison JT, Marquisee M, Merrill SH, Penswick JR, Zamir A (březen 1965). "Struktura ribonukleové kyseliny". Věda. 147 (3664): 1462–1465. Bibcode:1965Sci ... 147.1462H. doi:10.1126 / science.147.3664.1462. PMID 14263761. S2CID 40989800.
- ^ "Nekrolog". The New York Times. 4. července 1991.
- ^ „Nobelova cena za fyziologii nebo medicínu 1968“. Nobelova nadace. Citováno 2007-07-28.
- ^ Ladner JE, Jack A, Robertus JD, Brown RS, Rhodes D, Clark BF, Klug A (listopad 1975). "Struktura kvasinkové fenylalaninové přenosové RNA při rozlišení 2,5 A". Sborník Národní akademie věd Spojených států amerických. 72 (11): 4414–4418. Bibcode:1975PNAS ... 72,4414L. doi:10.1073 / pnas.72.11.4414. PMC 388732. PMID 1105583.
- ^ Kim SH, Quigley GJ, Suddath FL, McPherson A, Sneden D, Kim JJ, Weinzierl J, Rich A (leden 1973). "Trojrozměrná struktura kvasinkové fenylalaninové transferové RNA: skládání polynukleotidového řetězce". Věda. 179 (4070): 285–288. Bibcode:1973Sci ... 179..285K. doi:10.1126 / science.179.4070.285. PMID 4566654. S2CID 28916938.
externí odkazy
- tRNAdb (aktualizovaná a kompletně restrukturalizovaná verze kompilace Spritzls tRNA)
- Spojení tRNA se srdečními chorobami a mozkovou mrtvicí
- GtRNAdb: Sbírka tRNA identifikovaných z kompletních genomů
- HGNC: Genová nomenklatura lidských tRNA
- Molekula měsíce © RCSB Proteinová datová banka:
- Vstup Rfam pro tRNA