Stop kodonu - Stop codon

v molekulární biologie (konkrétně biosyntéza bílkovin ), a stop kodon (nebo terminační kodon) je kodon (nukleotid triplet uvnitř messenger RNA ), který signalizuje ukončení překlad proces proudu protein.[1] Většina kodonů v messengerové RNA odpovídá přidání an aminokyselina k rostoucímu polypeptid řetězec, který se nakonec může stát proteinem; stop kodony signalizují ukončení tohoto procesu vazbou uvolňovací faktory, které způsobují ribozomální podjednotky k disociaci, uvolnění aminokyselinového řetězce.
Zatímco spustit kodony potřebují blízké sekvence nebo iniciační faktory pro zahájení překladu je k zahájení ukončení dostatečný samotný stop kodon.
Vlastnosti
Standardní kodony
Ve standardním genetickém kódu existují tři různé terminační kodony:
| Codon | Standardní kód (Překladová tabulka 1) | název | ||
|---|---|---|---|---|
| DNA | RNA | |||
| ŠTÍTEK | UAG | STOP = Ter (*) | "jantar" | |
| TAA | UAA | STOP = Ter (*) | "okr" | |
| TGA | UGA | STOP = Ter (*) | „opál“ (nebo „umber“) | |
Alternativní stop kodony
Existují variace na standardní genetický kód, a alternativní stop kodony byly nalezeny v mitochondriální genomy z obratlovců,[2] Scenedesmus obliquus,[3] a Thraustochytrium.[4]
| Genetický kód | Překlad stůl | Codon | Překlad s tímto kódem | Standardní překlad | ||||
|---|---|---|---|---|---|---|---|---|
| DNA | RNA | |||||||
| Obratlovci mitochondriální | 2 | AGA | AGA | STOP = Ter (*) | Arg (R) | |||
| AGG | AGG | STOP = Ter (*) | Arg (R) | |||||
| Scenedesmus obliquus mitochondriální | 22 | TCA | UCA | STOP = Ter (*) | Ser (S) | |||
| Thraustochytrium mitochondriální | 23 | TTA | UUA | STOP = Ter (*) | Leu (L) | |||
| Aminokyselina biochemické vlastnosti | Nepolární | Polární | Základní | Kyselé | Ukončení: stop kodon |
Přepracované stop kodony
Nukleární genetický kód je flexibilní, jak dokládají alternativní genetické kódy, které přiřazují standardní stop kodony aminokyselinám.[5]
| Genetický kód | Překlad stůl | Codon | Podmiňovací způsob překlad | Standardní překlad | ||||
|---|---|---|---|---|---|---|---|---|
| DNA | RNA | |||||||
| Karyorelict nukleární | 27 | TGA | UGA | Ter (*) | nebo | Trp (Ž) | Ter (*) | |
| Condylostoma jaderný | 28 | TAA | UAA | Ter (*) | nebo | Gln (Q) | Ter (*) | |
| ŠTÍTEK | UAG | Ter (*) | nebo | Gln (Q) | Ter (*) | |||
| TGA | UGA | Ter (*) | nebo | Trp (Ž) | Ter (*) | |||
| Blastocrithidia jaderný | 31 | TAA | UAA | Ter (*) | nebo | Glu (E) | Ter (*) | |
| ŠTÍTEK | UAG | Ter (*) | nebo | Glu (E) | Ter (*) | |||
Překlad
V roce 2007 byl identifikován kodon UGA jako kodon kódující selenocystein (Sec) a nalezen v 25 selenoproteiny nachází se v aktivním místě proteinu. Překlad tohoto kodonu je umožněn blízkostí Prvek SECIS (SelenoCysteine Incorporation Sequence).[6]
Kodon UAG lze přeložit do pyrrolysin (Pyl) podobným způsobem.
Genomická distribuce
Distribuce stop kodonů v genomu organismu není náhodná a může s ní korelovat Obsah GC.[7][8] Například E-coli Genom K-12 obsahuje 2705 TAA (63%), 1257 TGA (29%) a 326 TAG (8%) stop kodonů (obsah GC 50,8%).[9] Také substráty faktoru uvolnění stop kodonů 1 nebo faktoru uvolnění 2 silně korelují s množstvím stop kodonů.[10] Rozsáhlá studie bakterií se širokou škálou obsahů GC ukazuje, že zatímco frekvence výskytu TAA negativně koreluje s obsahem GC a frekvence výskytu TGA pozitivně koreluje s obsahem GC, frekvence výskytu stop kodonu TAG, který je často minimálně použitým stop kodonem v genomu, není ovlivněn obsahem GC.[11]
Uznání
Rozpoznávání stop kodonů v bakteriích bylo spojeno s takzvaným „tripeptidovým antikodonem“,[12] vysoce konzervovaný aminokyselinový motiv v RF1 (PxT) a RF2 (SPF). I když to podporují strukturální studie, ukázalo se, že hypotéza tripeptidové antikodony je zjednodušením.[13]
Nomenklatura
Stop kodony dostaly historicky mnoho různých jmen, protože každá odpovídala odlišné třídě mutantů, kteří se všichni chovali podobným způsobem. Tito mutanti byli nejprve izolováni uvnitř bakteriofágy (T4 a lambda ), viry které infikují bakterie Escherichia coli. Mutace ve virových genech oslabily jejich infekční schopnost, někdy vytvářely viry, které byly schopné infikovat a růst pouze v určitých odrůdách E-coli.
jantar mutace (UAG)
Byli první sadou nesmyslné mutace být objeven, izolován Richardem H. Epsteinem a Charlesem Steinbergem a pojmenován podle jejich přítele a absolventa studenta Caltech Harrisa Bernsteina, jehož příjmení znamená "jantar " v němčině (srov. Bernstein ).[14][15]
Viry s jantarovými mutacemi se vyznačují schopností infikovat pouze určité kmeny bakterií, známé jako jantarové supresory. Tyto bakterie nesou svoji vlastní mutaci, která umožňuje obnovení funkce u mutantních virů. Například mutace v tRNA, která rozpoznává jantarový stop kodon, umožňuje translaci „přečíst“ kodon a produkovat protein plné délky, čímž se získá normální forma proteinu a „se potlačuje“ jantarová mutace.[16]Jantarové mutanty jsou tedy celou třídou virových mutantů, které mohou růst v bakteriích, které obsahují mutanty potlačující jantar. Podobné potlačující látky jsou známé také pro okrové a opálové stop kodony.
okr mutace (UAA)
Byla to objevená druhá mutace stop kodonu. Tento druhý stop kodon, který připomíná obvyklou žluto-oranžovo-hnědou barvu spojenou s jantarovou, dostal název "okr ", oranžovo-červenohnědý minerální pigment.[15]
Ochrové mutantní viry měly vlastnost podobnou jantarovým mutantům v tom, že obnovovaly infekční schopnost u určitých supresorových kmenů bakterií. Sada okrových supresorů byla odlišná od jantarových supresorů, takže byly okerové mutanty odvozeny tak, aby odpovídaly odlišnému tripletu nukleotidů. Prostřednictvím řady mutačních experimentů porovnávajících tyto mutanty navzájem a dalšími známými kodony aminokyselin, Sydney Brenner dospěli k závěru, že jantarová a okrová mutace odpovídají nukleotidovým tripletům „UAG“ a „UAA“.[17]
opál nebo umber mutace (UGA)
Třetí a poslední stop kodon ve standardním genetickém kódu byl objeven brzy poté a odpovídá nukleotidovému tripletu „UGA“.[18]
Abychom pokračovali ve shodě s tématem barevných minerálů, třetí nesmyslný kodon byl znám jako "opál ", což je druh oxidu křemičitého vykazující různé barvy.[15] Nesmyslné mutace, které vytvořily tento předčasný stop kodon, se později nazývaly opální mutace nebo umber mutace.
Mutace
Nesmysl
Nesmyslné mutace jsou změny v sekvenci DNA, které zavádějí předčasný stop kodon, což způsobuje abnormální zkrácení výsledného proteinu. To často způsobuje ztrátu funkce proteinu, protože kritické části řetězce aminokyselin již nejsou vytvářeny. Kvůli této terminologii byly stop kodony označovány také jako nesmyslné kodony.
Nepřetržitě
A nonstop mutace je bodová mutace který se vyskytuje v stop kodonu. Nonstop mutace způsobují pokračující překlad souboru mRNA řetězec do oblasti, která by měla být nepřekládanou oblastí. Většina polypeptidy vznikající z genu s nonstop mutací jsou nefunkční kvůli své extrémní délce.
Nonstop mutace se liší od nesmyslné mutace v tom, že nevytvářejí stop kodon, ale místo toho jeden odstraní. Nonstop mutace se také liší od missense mutace, což jsou bodové mutace, kdy je změněn jeden nukleotid a nahrazen jiným aminokyselina.
Nonstop mutace byly spojeny s několika vrozenými chorobami včetně vrozená hyperplázie nadledvin,[19] variabilní dysgeneze předního segmentu,[20] cystická fibróza[21] a mitochondriální neurogastrointestinální encefalomyopatie.[22]
Skryté zastávky
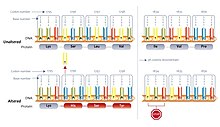
Skryté zastávky jsou non-stop kodony, které by se číst jako stop kodony, pokud by byly rámy posunuty +1 nebo -1. Tyto předčasně ukončují translaci, pokud dojde k odpovídajícímu posunu rámce (například v důsledku prokluzu ribozomální RNA) před skrytým zastavením. Předpokládá se, že to snižuje plýtvání zdroji nefunkčních proteinů a produkci potenciálu cytotoxiny. Výzkumní pracovníci v Louisianská státní univerzita navrhnout přepadová hypotéza, pro které jsou vybrány skryté zastávky. Kodony, které mohou vytvářet skryté zastávky, se v genomech používají častěji ve srovnání se synonymními kodony, které by jinak kódovaly stejnou aminokyselinu. Nestabilní rRNA v organismu koreluje s vyšší frekvencí skrytých zastávek.[23]Tuto hypotézu však nebylo možné ověřit u většího souboru dat.[24]
Stop-kodony a skryté dorazy společně se souhrnně označují jako stop-signály. Výzkumní pracovníci v University of Memphis zjistili, že poměry stop signálů na třech čtecích rámcích genomu (označovaných jako poměr translačních stop signálů nebo TSSR) geneticky příbuzných bakterií jsou navzdory jejich velkým rozdílům v obsahu genů velmi podobné. Tato téměř identická hodnota Genomic-TSSR u geneticky příbuzných bakterií může naznačovat, že expanze bakteriálního genomu je omezena jejich jedinečným zkreslením stop-signálů daného bakteriálního druhu.[25]
Překladové čtení
Zastavte potlačení kodonu nebo překladové čtení nastává, když je v překladu stop kodon interpretován jako sense kodon, to znamená, když je (standardní) aminokyselina „kódována“ stop kodonem. Mutováno tRNA může být příčinou čtení, ale také jisté nukleotid motivy blízko stop kodonu. Překladové čtení je velmi časté u virů a bakterií a bylo také nalezeno jako princip regulace genů u lidí, kvasinek, bakterií a drosophil.[26][27] Tento druh endogenního translačního čtení představuje variantu genetický kód, protože stop kodon kóduje aminokyselinu. V případě člověka malát dehydrogenáza se čte stop kodon s frekvencí asi 4%.[28] Aminokyselina vložená na stop kodon závisí na identitě samotného stop kodonu: Gln, Tyr a Lys byly nalezeny pro kodony UAA a UAG, zatímco Cys, Trp a Arg pro UGA kodon byly identifikovány hmotnostně spektrometrie.[29]
Použít jako vodoznak
V roce 2010, kdy Craig Venter představila první plně funkční reprodukující buňku ovládanou syntetická DNA popsal, jak jeho tým k tvorbě používal časté stop kodony vodoznaky v RNA a DNA, aby se potvrdilo, že výsledky jsou skutečně syntetické (a nejsou kontaminované ani jinak), přičemž se používají ke kódování jmen autorů a adres webových stránek.[30]
Viz také
Reference
- ^ Griffiths AJF, Miller JH, Suzuki DT, Lewontin RC, Gelbart WM (2000). „Kapitola 10 (Molekulární biologie genové funkce): Genetický kód: Stop kodony“. Úvod do genetické analýzy. W.H. Freeman a společnost.
- ^ Barrell, B. G .; Bankier, A. T .; Drouin, J. (11.11.1979). „Jiný genetický kód v lidských mitochondriích“. Příroda. 282 (5735): 189–194. doi:10.1038 / 282189a0. ISSN 0028-0836. PMID 226894. S2CID 4335828.
- ^ A. M. Nedelcu, R. W. Lee, G. Lemieux, M. W. Gray, G. Burger (červen 2000). "Kompletní mitochondriální sekvence DNA Scenedesmus obliquus odráží mezistupeň ve vývoji mitochondriálního genomu zelených řas ". Výzkum genomu. 10 (6): 819–831. doi:10,1101 / gr. 10.6.819. PMC 310893. PMID 10854413.CS1 maint: více jmen: seznam autorů (odkaz)
- ^ Wideman, Jeremy G .; Monier, Adam; Rodríguez-Martínez, Raquel; Leonard, Guy; Cook, Emily; Poirier, Camille; Maguire, Finlay; Milner, David S .; Irwin, Nicholas A. T .; Moore, Karen; Santoro, Alyson E. (2019-11-25). „Neočekávaná rozmanitost mitochondriálního genomu odhalená cílenou jednobuněčnou genomikou heterotrofních bičíkatých protistů“. Přírodní mikrobiologie. 5 (1): 154–165. doi:10.1038 / s41564-019-0605-4. hdl:10871/39819. ISSN 2058-5276. PMID 31768028. S2CID 208279678.
- ^ Swart, Estienne Carl; Serra, Valentina; Petroni, Giulio; Nowacki, Mariusz (2016). „Genetické kódy bez vyhrazeného stop kodonu: ukončení závislé na kontextu“. Buňka. 166 (3): 691–702. doi:10.1016 / j.cell.2016.06.020. PMC 4967479. PMID 27426948.
- ^ Papp, Laura Vanda; Lu, červen; Holmgren, Arne; Khanna, Kum Kum (2007). „Od selenu k selenoproteinům: syntéza, identita a jejich role v lidském zdraví“. Antioxidanty a redoxní signalizace. 9 (7): 775–806. doi:10.1089 / ars.2007.1528. PMID 17508906. S2CID 38176932.
- ^ Povolotskaya IS, Kondrashov FA, Ledda A, Vlasov PK (2012). „Stop kodony v bakteriích nejsou selektivně ekvivalentní“. Biology Direct. 7: 30. doi:10.1186/1745-6150-7-30. PMC 3549826. PMID 22974057.
- ^ Korkmaz, Gürkan; Holm, Mikael; Wiens, Tobias; Sanyal, Suparna (2014). „Komplexní analýza využití stop kodonu u bakterií a jejich korelace s množstvím faktoru uvolnění“. The Journal of Biological Chemistry. 289 (44): 775–806. doi:10,1074 / jbc.M114,606632. PMC 4215218. PMID 25217634.
- ^ "Escherichia coli str. Substrát K-12 MG1655, kompletní genom [přístupové číslo Genbank: U00096] ". GenBank. NCBI. Citováno 2013-01-27.
- ^ Korkmaz, Gürkan; Holm, Mikael; Wiens, Tobias; Sanyal, Suparna (2014). „Komplexní analýza využití stop kodonu u bakterií a jejich korelace s množstvím faktoru uvolnění“. The Journal of Biological Chemistry. 289 (44): 775–806. doi:10,1074 / jbc.M114,606632. PMC 4215218. PMID 25217634.
- ^ Wong, Tit-Yee; Fernandes, Sanjit; Sankhon, Naby; Leong, Patrick P; Kuo, Jimmy; Liu, Jong-Kang (2008). „Úloha předčasných stop kodonů v bakteriální evoluci“. Journal of Bacteriology. 190 (20): 6718–6725. doi:10.1128 / JB.00682-08. PMC 2566208. PMID 18708500.
- ^ Ito, Koichi; Uno, Makiko; Nakamura, Yoshikazu (1999). „Tripeptidové„ antikodonové “dešifry zastavují kodony v messengerové RNA“. Příroda. 403 (6770): 680–684. doi:10.1038/35001115. PMID 10688208. S2CID 4331695.
- ^ Korkmaz, Gürkan; Sanyal, Suparna (2017). „Mutace R213I v uvolňovacím faktoru 2 (RF2) je krokem vpřed pro konstrukci všemocného uvolňovacího faktoru v bakteriích Escherichia coli". Journal of Biological Chemistry. 292 (36): 15134–15142. doi:10,1074 / jbc.M117,785238. PMC 5592688. PMID 28743745.
- ^ Stahl FW (1995). „Jantarové mutanty fága T4“. Genetika. 141 (2): 439–442. PMC 1206745. PMID 8647382.
- ^ A b C Lewin, Benjamin; Krebs, Jocelyn E .; Goldstein, Elliott S .; Kilpatrick, Stephen T. (2011-04-18). Lewin's Essential GENES. Vydavatelé Jones & Bartlett. ISBN 978-1-4496-4380-5.
- ^ Robin Cook. "Shrnutí mutací jantaru, okrové a opálu". Svět genetiky. Vichřice.
- ^ Brenner, S .; Stretton, A. O. W .; Kaplan, S. (1965). „Genetický kód:„ nesmysly “triplety pro ukončení řetězce a jejich potlačení. Příroda. 206 (4988): 994–8. Bibcode:1965Natur.206..994B. doi:10.1038 / 206994a0. PMID 5320272. S2CID 28502898.
- ^ Brenner, S .; Barnett, L .; Katz, E. R .; Crick, F. H. C. (1967). „UGA: Třetí nesmyslný triplet v genetickém kódu“. Příroda. 213 (5075): 449–50. Bibcode:1967Natur.213..449B. doi:10.1038 / 213449a0. PMID 6032223. S2CID 4211867.
- ^ Pang S .; Wang W .; et al. (2002). „Nová nonstop mutace v stop kodonu a nová missense mutace v genu 3beta-hydroxysteroid dehydrogenázy (3beta-HSD) typu II způsobující neklasickou a klasickou vrozenou hyperplázii nadledvin s nedostatkem 3beta-HSD.“ J Clin Endocrinol Metab. 87 (6): 2556–63. doi:10.1210 / jc.87.6.2556. PMID 12050213.
- ^ Doucette, L .; et al. (2011). „Nová, nepřetržitá mutace v FOXE3 způsobuje autozomálně dominantní formu dysgeneze variabilního předního segmentu včetně Petersovy anomálie ". European Journal of Human Genetics. 19 (3): 293–299. doi:10.1038 / ejhg.2010.210. PMC 3062009. PMID 21150893.
- ^ Guimbellot, Jennifer; Sharma, Jyoti; Rowe, Steven M. (listopad 2017). „Směrem k inkluzivní terapii s modulátory CFTR: pokrok a výzvy“. Pediatrická pulmonologie. 52 (S48): S4 – S14. doi:10,1002 / ppul.23773. ISSN 1099-0496. PMC 6208153. PMID 28881097.
- ^ Torres-Torronteras, J .; Rodriguez-Palmero, A .; et al. (2011). „Nová nonstop mutace v TYMP neindukuje nonstop rozpad mRNA u pacientů s MNGIE s těžkou neuropatií“ (PDF). Hučení. Mutat. 32 (4): E2061 – E2068. doi:10,1002 / humu.21447. PMID 21412940.
- ^ Seligmann, Hervé; Pollock, David D. (2004). „Hypotéza přepadení: Skryté stop kodony zabraňují čtení genů mimo rámec“. DNA a buněčná biologie. 23 (10): 701–5. doi:10.1089/1044549042476910. PMID 15585128.
- ^ Cavalcanti, Andre; Chang, Charlotte H .; Morgens, David W. (2013). „Přepadení hypotézy přepadení: předpovídání a hodnocení kmitočtů kodonů mimo rámec v prokaryotických genomech“. BMC Genomics. 14 (418): 1–8. doi:10.1186/1471-2164-14-418. PMC 3700767. PMID 23799949.
- ^ Wong, Tit-Yee; Schwartzbach, Steve (2015). „Nesprávné ukončení bílkovin iniciuje genetická onemocnění, rakoviny a omezuje expanzi bakteriálního genomu.“ Journal of Environmental Science and Health, Part C. 33 (3): 255–85. doi:10.1080/10590501.2015.1053461. PMID 26087060. S2CID 20380447.
- ^ Namy O, Rousset JP, Napthine S, Brierley I (2004). "Přeprogramované genetické dekódování v expresi buněčných genů". Molekulární buňka. 13 (2): 157–68. doi:10.1016 / S1097-2765 (04) 00031-0. PMID 14759362.
- ^ Schueren F, Lingner T, George R, Hofhuis J, Gartner J, Thoms S (2014). „Peroxisomální laktátdehydrogenáza je generována translačním přečtením u savců“. eLife. 3: e03640. doi:10,7554 / eLife.03640. PMC 4359377. PMID 25247702.
- ^ Hofhuis J, Schueren F, Nötzel C, Lingner T, Gärtner J, Jahn O, Thoms S (2016). „Funkční rozšíření čtení malátdehydrogenázy odhaluje modifikaci genetického kódu“. Otevřená Biol. 6 (11): 160246. doi:10.1098 / rsob.160246. PMC 5133446. PMID 27881739.
- ^ Blanchet S, Cornu D, Argentini M, Namy O (2014). "Nové poznatky o začlenění přirozených supresorových tRNA na stop kodony v systému" Saccharomyces cerevisiae". Nucleic Acids Res. 42 (15): 10061–72. doi:10.1093 / nar / gku663. PMC 4150775. PMID 25056309.
- ^ „Sledujte, jak odhaluji“ syntetický život"".
