RNA polymeráza II - RNA polymerase II
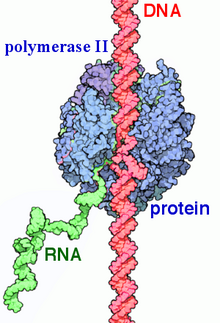
RNA polymeráza II (RNAP II a Pol II) je multiproteinový komplex že přepisuje DNA do předchůdců messenger RNA (mRNA) a většina malá jaderná RNA (snRNA) a mikroRNA.[1][2] Je to jeden ze tří RNAP enzymy nalezený v jádru eukaryotický buňky.[3] 550 kDa komplex 12 podjednotek, RNAP II je nejvíce studovaným typem RNA polymeráza. Široká škála transkripční faktory jsou potřebné k tomu, aby se mohl vázat na upstream gen promotéři a zahájit přepis.
Objev

První studie navrhly minimálně dva RNAP: jeden, který syntetizoval rRNA v jádro a ten, který syntetizoval další RNA v nukleoplazma, část jádra, ale mimo jádro.[5] V roce 1969 vědečtí experimentátoři Robert Roeder a William Rutter definitivně objevil další RNAP, který byl zodpovědný za transkripci nějakého druhu RNA v nukleoplazmě. Nález byl získán použitím iontoměničová chromatografie přes DEAE potažené Sephadex korálky. Technika odděluje enzymy v pořadí odpovídajících elucí, Ι, ΙΙ, ΙΙΙ, zvýšením koncentrace síranu amonného. Enzymy byly pojmenovány podle pořadí elucí, RNAP I, RNAP II, RNAP IΙI.[3] Tento objev prokázal, že v nukleoplazmě byl přítomen další enzym, který umožňoval diferenciaci mezi RNAP II a RNAP III.
Podjednotky

The eukaryotický jádrová polymeráza II byla nejprve purifikována pomocí transkripčních testů.[7] Vyčištěný enzym má obvykle 10–12 podjednotek (12 u lidí a kvasinek) a není schopen specifického rozpoznávání promotoru.[8] Je známo mnoho interakcí mezi podjednotkami a podjednotkami.[9]
- DNA-řízená RNA polymeráza II podjednotka RPB1 - an enzym že u lidí je kódován POLR2A gen a v kvasinkách je kódován RPO21. RPB1 je největší podjednotkou RNA polymerázy II. Obsahuje a karboxyterminální doména (CTD) složené až z 52 heptapeptidových repeticí (YSPTSPS), které jsou nezbytné pro aktivitu polymerázy.[10] CTD bylo poprvé objeveno v laboratoři C. J. Inglese na univerzitě v Torontu a JL Cordenem v Univerzita Johna Hopkinse. V kombinaci s několika dalšími podjednotkami polymerázy tvoří podjednotka RPB1 vazebnou doménu DNA polymerázy, drážku, ve které je DNA templát. přepsal do RNA.[11] Silně interaguje s RPB8.[9]
- RPB2 (POLR2B ) - druhá největší podjednotka, která v kombinaci s alespoň dvěma dalšími podjednotkami polymerázy tvoří strukturu uvnitř polymerázy, která udržuje kontakt v aktivním místě enzymu mezi templátem DNA a nově syntetizovanou RNA.[12]
- RPB3 (POLR2C ) - třetí největší podjednotka. Existuje jako heterodimer s jinou polymerázovou podjednotkou, POLR2J vytvoření podsestavy jádra. RPB3 silně interaguje s RPB1-5, 7, 10–12.[9]
- RNA polymeráza II podjednotka B4 (RPB4) - kódováno POLR2D gen[13] je čtvrtou největší podjednotkou a může mít roli chránící proti stresu.
- RPB5 - U lidí je kódován POLR2E gen. V každé RNA polymeráze II jsou přítomny dvě molekuly této podjednotky.[14] RPB5 silně interaguje s RPB1, RPB3 a RPB6.[9]
- RPB6 (POLR2F ) - tvoří strukturu s alespoň dvěma dalšími podjednotkami, která stabilizuje transkripční polymerázu na templátu DNA.[15]
- RPB7 - kódováno POLR2G a může hrát roli v regulaci funkce polymerázy.[16] RPB7 silně interaguje s RPB1 a RPB5.[9]
- RPB8 (POLR2H ) - interaguje s podjednotkami RPB1-3, 5 a 7.[9]
- RPB9 - Drážka, ve které je šablona DNA přepsána do RNA, se skládá z RPB9 (POLR2I ) a RPB1.
- RPB10 - produkt gen POLR2L. Interaguje s RPB1-3 a 5 a silně s RPB3.[9]
- RPB11 - podjednotka RPB11 se sama skládá ze tří podjednotek u lidí: POLR2J (RPB11-a), POLR2J2 (RPB11-b) a POLR2J3[17] (RPB11-c).
- RPB12 - Interakce s RPB3 je také RPB12 (POLR2K ).[9]
Shromáždění
RPB3 se podílí na sestavení RNA polymerázy II.[18] Podkomplex RPB2 a RPB3 se objeví brzy po syntéze podjednotky.[18] Tento komplex následně interaguje s RPB1.[18] RPB3, RPB5 a RPB7 interagují se sebou a vytvářejí homodimery a RPB3 a RPB5 společně mohou kontaktovat všechny ostatní podjednotky RPB, s výjimkou RPB9.[9] Pouze RPB1 se silně váže na RPB5.[9] Podjednotka RPB1 také kontaktuje RPB7, RPB10 a slaběji, ale nejúčinněji s RPB8.[9] Jakmile RPB1 vstoupí do komplexu, mohou vstoupit další podjednotky jako RPB5 a RPB7, kde se RPB5 váže na RPB6 a RPB8 a RPB3 přináší RPB10, RPB 11 a RPB12.[9] RPB4 a RPB9 mohou vstoupit, jakmile je sestavena většina komplexu. RPB4 tvoří komplex s RPB7.[9]
Kinetika
Enzymy může katalyzovat až několik milionů reakcí za sekundu. Rychlost enzymu závisí na podmínkách roztoku a koncentraci substrátu. Stejně jako ostatní enzymy má POLR2 saturační křivku a maximální rychlost (PROTImax). Má to K.m (koncentrace substrátu je nutná pro polovinu PROTImax) a a kkočka (počet molekul substrátu zpracovaných jedním aktivním místem za sekundu). Konstanta specificity je dána vztahem kkočka/K.m. Teoretické maximum pro konstantu specificity je difúzní limit asi 108 do 109 (M−1s−1), kde každá srážka enzymu s jeho substrátem vede k katalýze. U kvasinek může mutace v doméně Trigger-Loop největší podjednotky změnit kinetiku enzymu.[19]
Bakteriální RNA polymeráza, příbuzný RNA polymerázy II, přepíná mezi inaktivovaným a aktivovaným stavem translokací tam a zpět podél DNA.[20] Koncentrace [NTP]ekv = 10 μM GTP, 10 μM UTP, 5 μM ATP a 2,5 μM CTP, produkují průměrnou rychlost prodloužení, číslo obratu ~ 1 bp (NTP)−1 pro bakteriální RNAP, příbuzný RNA polymerázy II.[20]

RNA polymeráza II prochází během prodlužování transkripce rozsáhlou pa-transkripcí.[21][22] Tato pauza je zvláště výrazná u nukleosomů a vzniká částečně polymerázou, která vstupuje do transkripčně nekompetentního stavu zpětného odběru.[21] Doba trvání těchto pauz se pohybuje od sekund do minut nebo déle a ukončení z dlouhodobých pauz může být podporováno faktory prodloužení, jako je TFIIS.[23] Rychlost transkripce zase ovlivňuje, zda jsou histony transkribovaných nukleosomů vypuzeny z chromatinu, nebo jsou znovu vloženy za transkripční polymerázu.[24]
Alfa-Amanitin
RNA polymeráza II je inhibována α-Amanitin[25] a další amatoxiny. α-Amanitin je vysoce jedovatá látka nacházející se v mnoha houbách.[5] Houbový jed má různé účinky na každou z RNA polymeráz: I, II, III. RNAP I na látku zcela nereaguje a bude fungovat normálně, zatímco RNAP III má střední citlivost. RNAP II je však toxinem zcela inhibován. Alfa-Amanitin inhibuje RNAP II silnými interakcemi v „trychtýři“, „rozštěpu“ a klíčovém „můstku“ enzymu α-šroubovice "regiony podjednotky RPB-1.[26]
Holoenzym
Holoenzym RNA polymerázy II je forma eukaryotický RNA polymeráza II, která se získává do promotorů protein -kódující geny v živých buňkách.[8] Skládá se z RNA polymerázy II, podskupiny obecně transkripční faktory, a regulační proteiny známé jako SRB proteiny.
Část sestavy holoenzymu se označuje jako preiniciační komplex, protože jeho montáž probíhá na gen promotér před zahájením transkripce. The komplex mediátorů působí jako most mezi RNA polymerázou II a transkripčními faktory.
Kontrola strukturou chromatinu
Toto je nástin příkladu mechanismu kvasinkových buněk, kterým chromatin struktura a histon posttranslační modifikace pomáhají regulovat a zaznamenávat transkripce z geny RNA polymerázou II.
Tato cesta poskytuje příklady regulace v těchto bodech transkripce:
- Preinicializace (propagace Bre1, modifikace histonu)
- Zahájení (propagace TFIIH, modifikace Pol II A propagace KOMPASEM, modifikace histonu)
- Prodloužení (propagace Set2, úprava histonu)
Vezměte prosím na vědomí, že se jedná o různé fáze procesu jako regulační kroky. Nebylo prokázáno, že se používají k regulaci, ale je velmi pravděpodobné, že jsou.
Promotory pro prodloužení RNA Pol II lze shrnout do 3 tříd.
- Faktory ovlivněné zástavou závislou na léku / sekvenci (různé interferující proteiny)
- Faktory orientované na strukturu chromatinu (posttranskripční modifikátory histonu, např. Histonmethyltransferázy)
- Faktory zlepšující katalýzu RNA Pol II (různé interferující proteiny a kofaktory Pol II; viz RNA polymeráza II).
Transkripční mechanismy
- Faktory orientované na strukturu chromatinu:
(HMT (Histone MethylTransferázy)):
KOMPAS § † - (KOMplex Pbílkoviny TAK JAKOspojen s Set1) - Methyláty lysin 4 histonu H3: Je odpovědný za represi / umlčení transkripce. Normální součást buněčného růstu a regulace transkripce v rámci RNAP II.[27] - Set2 - Methyláty lysin 36 histonu H3: Set2 se podílí na prodloužení regulační transkripce prostřednictvím svého přímého kontaktu s CTD.[28]
(zajímavý irelevantní příklad: Dot1 * ‡ - methyláty lysin 79 histonu H3.) - Bre1 - ubikvináty (dodává ubikvitin až) lysin 123 histonu H2B. Spojeno s preiniciací a umožněním navázání RNA Pol II.
CTD RNA polymerázy
C-konec RPB1 je připojen k vytvoření C-terminální domény (CTD). Karboxyterminální doména RNA polymerázy II se obvykle skládá až z 52 opakování sekvence Tyr-Ser-Pro-Thr-Ser-Pro-Ser.[29] Doména se táhne od jádra enzymu RNAPII k výstupnímu kanálu, toto umístění je účinné díky své indukci „reakcí zpracování RNA, prostřednictvím přímých nebo nepřímých interakcí se složkami zařízení pro zpracování RNA“.[30] Doména CTD neexistuje v RNA polymeráza I nebo RNA polymeráza III.[3] RNA polymeráza CTD byla objevena nejprve v laboratoři C.J.Inglese na University of Toronto a také v laboratoři J. Cordena na Johns Hopkins University během procesů sekvenování DNA kódující podjednotku RPB1 RNA polymerázy z kvasinek a myší. Jiné proteiny se často vážou na C-koncovou doménu RNA polymerázy, aby aktivovaly aktivitu polymerázy. Je to proteinová doména, která se podílí na zahájení transkripce omezení z Přepis RNA a připojení k spliceosome pro Sestřih RNA.[10]
Fosforylace domény CTD
RNA polymeráza II existuje ve dvou formách, nefosforylovaných a fosforylovaných, IIA a IIO.[5][3] Přechod mezi těmito dvěma formami usnadňuje různé funkce transkripce. The fosforylace CTD je katalyzován jedním ze šesti obecné transkripční faktory, TFIIH. TFIIH slouží dvěma účelům: jedním je odvíjení DNA v počátečním místě transkripce a druhým je fosforylace. Forma polymerázy IIA se připojuje k preiniciačnímu komplexu, což se navrhuje, protože IIA se váže s vyšší afinitou na TBP (Vazebný protein TATA-box ), podjednotka obecného transkripčního faktoru TFIID, než forma polymerázy IIO. Forma polymerázy IIO usnadňuje prodloužení řetězce RNA.[5] Způsob iniciace prodloužení se provádí fosforylací Serine v poloze 5 (Ser5), přes TFIIH. Nově fosforylovaný Ser5 rekrutuje enzymy k uzavření 5 'konce nově syntetizované RNA a faktorů zpracování „3“ k poly (A) stránky ".[30] Jakmile je druhý Serin fosforylován, Ser2, je aktivováno prodloužení. K ukončení prodloužení musí dojít k defosforylaci. Jakmile je doména zcela defosforylována, je enzym RNAP II „recyklován“ a katalyzuje stejný proces s jiným iniciačním místem.[30]
Rekombinační oprava spojená s transkripcí
Oxidační poškození DNA může blokovat RNA polymerázu II transkripce a způsobit přetržení vlákna. Byl popsán rekombinační proces spojený s transkripcí spojený s RNA, který může chránit před poškozením DNA.[31] Během fází G1 / G0 buněčný cyklus, buňky vykazují shromáždění homologních rekombinačních faktorů při dvouvláknových zlomech v aktivně transkribovaných oblastech. Zdá se, že transkripce je spojena s opravou dvouřetězcových zlomů DNA homologní rekombinací templátovanou RNA. Tento proces opravy efektivně a přesně spojuje dvouvláknové vloupání geny je aktivně transkribován RNA polymerázou II.
Viz také
- Eukaryotická transkripce
- Post-transkripční modifikace
- RNA polymeráza I
- Holoenzym RNA polymerázy II
- RNA polymeráza III
- Přepis (genetika)
Reference
- ^ Kornberg RD (prosinec 1999). "Eukaryotická kontrola transkripce". Trendy v buněčné biologii. 9 (12): M46–9. doi:10.1016 / S0962-8924 (99) 01679-7. PMID 10611681.
- ^ Sims RJ, Mandal SS, Reinberg D (červen 2004). "Nejnovější poznatky o transkripci zprostředkované RNA-polymerázou-II". Aktuální názor na buněčnou biologii. 16 (3): 263–71. doi:10.1016 / j.ceb.2004.04.004. PMID 15145350.
- ^ A b C d Young, Richard A. (2003-11-28). „RNA polymeráza II“. Roční přehled biochemie. 60 (1): 689–715. doi:10.1146 / annurev.bi.60.070191.003353. PMID 1883205.
- ^ Meyer PA, Ye P, Zhang M, Suh MH, Fu J (červen 2006). „Fázování RNA polymerázy II pomocí vnitřně vázaných atomů Zn: aktualizovaný strukturální model“. Struktura. 14 (6): 973–82. doi:10.1016 / j.str.2006.04.003. PMID 16765890.
- ^ A b C d 1942-, Weaver, Robert Franklin (01.01.2012). Molekulární biologie. McGraw-Hill. ISBN 9780073525327. OCLC 789601172.CS1 maint: číselné názvy: seznam autorů (odkaz)
- ^ Armache, Karim-Jean; Mitterweger, Simone; Meinhart, Anton; Cramer, Patrick (2019). „Struktury kompletní RNA polymerázy II a jejího subkomplexu, Rpb4 / 7“ (PDF). Journal of Biological Chemistry. 280 (8): 7131–1734. doi:10,2210 / pdb1wcm / pdb. PMID 15591044.
- ^ Sawadogo M, Sentenac A (1990). „RNA polymeráza B (II) a obecné transkripční faktory“. Roční přehled biochemie. 59: 711–54. doi:10.1146 / annurev.bi.59.070190.003431. PMID 2197989.
- ^ A b Myer VE, Young RA (říjen 1998). "RNA polymeráza II holoenzymy a subkomplexy". The Journal of Biological Chemistry. 273 (43): 27757–60. doi:10.1074 / jbc.273.43.27757. PMID 9774381.
- ^ A b C d E F G h i j k l m Acker J, de Graaff M, Cheynel I, Khazak V, Kedinger C, Vigneron M (červenec 1997). „Interakce mezi podjednotkami lidské RNA polymerázy II“. The Journal of Biological Chemistry. 272 (27): 16815–21. doi:10.1074 / jbc.272.27.16815. PMID 9201987.
- ^ A b Brickey WJ, Greenleaf AL (červen 1995). „Funkční studie karboxy-terminální repetiční domény Drosophila RNA polymerázy II in vivo“. Genetika. 140 (2): 599–613. PMC 1206638. PMID 7498740.
- ^ „Entrez Gene: POLR2A polymeráza (RNA) II (řízená DNA) polypeptid A, 220 kDa“.
- ^ „Entrez Gene: POLR2B polymeráza (RNA) II (DNA řízený) polypeptid B, 140kDa“.
- ^ Khazak V, Estojak J, Cho H, Majors J, Sonoda G, Testa JR, Golemis EA (duben 1998). „Analýza interakce nové podjednotky hsRPB4 RNA polymerázy II (pol II) s partnerem hsRPB7 a s pol II“. Molekulární a buněčná biologie. 18 (4): 1935–45. doi:10.1128 / mcb.18.4.1935. PMC 121423. PMID 9528765.
- ^ „Entrez Gene: POLR2E polymeráza (RNA) II (řízená DNA) polypeptid E, 25 kDa“.
- ^ „Entrez Gene: POLR2F polymeráza (RNA) II (DNA řízený) polypeptid F“.
- ^ „Entrez Gene: POLR2G polymeráza (RNA) II (DNA řízený) polypeptid G“.
- ^ „POLR2J3 polymeráza (RNA) II (DNA řízený) polypeptid J3“.
- ^ A b C Kolodziej PA, Young RA (září 1991). „Mutace ve třech největších podjednotkách kvasinkové RNA polymerázy II, které ovlivňují sestavu enzymů“. Molekulární a buněčná biologie. 11 (9): 4669–78. doi:10,1128 / mcb.11.9.4669. PMC 361357. PMID 1715023.
- ^ Kaplan CD, Jin H, Zhang IL, Belyanin A (12. dubna 2012). „Disekce funkce spouštěcí smyčky Pol II a řízení aktivity místa Pol závislé na aktivaci výběru počátečního místa in vivo“. Genetika PLOS. 8 (4): e1002627. doi:10.1371 / journal.pgen.1002627. PMC 3325174. PMID 22511879.
- ^ A b Abbondanzieri EA, Greenleaf WJ, Shaevitz JW, Landick R, Block SM (listopad 2005). „Přímé pozorování krokování párů bází pomocí RNA polymerázy“. Příroda. 438 (7067): 460–5. Bibcode:2005 Natur.438..460A. doi:10.1038 / příroda04268. PMC 1356566. PMID 16284617.
- ^ A b Hodges, Courtney; Bintu, Lacramioara; Lubkowska, Lucyna; Kashlev, Michail; Bustamante, Carlos (2009-07-31). „Nukleosomální fluktuace určují dynamiku transkripce RNA polymerázy II“. Věda. 325 (5940): 626–628. Bibcode:2009Sci ... 325..626H. doi:10.1126 / science.1172926. ISSN 1095-9203. PMC 2775800. PMID 19644123.
- ^ Churchman, L. Stirling; Weissman, Jonathan S. (2011-01-20). „Sekvenční transkripční sekvenování vizualizuje transkripci v rozlišení nukleotidů“. Příroda. 469 (7330): 368–373. Bibcode:2011 Natur.469..368C. doi:10.1038 / nature09652. ISSN 1476-4687. PMC 3880149. PMID 21248844.
- ^ Galburt, Eric A .; Grill, Stephan W .; Wiedmann, Anna; Lubkowska, Lucyna; Choy, Jasone; Nogales, Eva; Kashlev, Michail; Bustamante, Carlos (12.04.2007). "Zpětné sledování určuje silovou citlivost RNAP II faktorově závislým způsobem". Příroda. 446 (7137): 820–823. Bibcode:2007Natur.446..820G. doi:10.1038 / nature05701. ISSN 1476-4687. PMID 17361130. S2CID 4310108.
- ^ Bintu, Lacramioara; Kopaczynska, Marta; Hodges, Courtney; Lubkowska, Lucyna; Kashlev, Michail; Bustamante, Carlos (2011-11-13). „Rychlost prodloužení RNA polymerázy určuje osud transkribovaných nukleosomů“. Přírodní strukturní a molekulární biologie. 18 (12): 1394–1399. doi:10.1038 / nsmb.2164. ISSN 1545-9985. PMC 3279329. PMID 22081017.
- ^ Kaplan CD, Larsson KM, Kornberg RD (červen 2008). „Spouštěcí smyčka RNA polymerázy II funguje při výběru substrátu a je přímo zaměřena na alfa-amanitin.“. Molekulární buňka. 30 (5): 547–56. doi:10.1016 / j.molcel.2008.04.023. PMC 2475549. PMID 18538653.
- ^ Gong, Xue Q .; Nedialkov, Yuri A .; Burton, Zachary F. (2004-06-25). „α-Amanitin blokuje translokaci lidskou RNA polymerázou II“. Journal of Biological Chemistry. 279 (26): 27422–27427. doi:10,1074 / jbc.M402163200. ISSN 0021-9258. PMID 15096519.
- ^ Briggs, Scott D .; Bryk, Mary; Strahl, Brian D .; Cheung, Wang L .; Davie, Judith K .; Dent, Sharon Y. R .; Winston, Fred; Allis, C. David (2001-12-15). „Methylace histonu H3 lysinu 4 je zprostředkována Set1 a je nutná pro růst buněk a umlčení rDNA u Saccharomyces cerevisiae“. Geny a vývoj. 15 (24): 3286–3295. doi:10,1101 / gad. 940201. ISSN 0890-9369. PMC 312847. PMID 11751634.
- ^ Li, Bing; Howe, LeAnn; Anderson, Scott; Yates, John R .; Workman, Jerry L. (2003-03-14). „Funkce Set2 histonmethyltransferázy prostřednictvím fosforylované karboxylové koncové domény RNA polymerázy II“. Journal of Biological Chemistry. 278 (11): 8897–8903. doi:10,1074 / jbc.M212134200. ISSN 0021-9258. PMID 12511561.
- ^ Meinhart A, Cramer P (červenec 2004). "Rozpoznání karboxyterminální domény RNA polymerázy II pomocí faktorů zpracování 3'-RNA". Příroda. 430 (6996): 223–6. Bibcode:2004 Natur.430..223M. doi:10.1038 / nature02679. PMID 15241417. S2CID 4418258.
- ^ A b C Egloff, Sylvain; Murphy, Shona (2008). Msgstr "Praskání kódu CTD RNA polymerázy II". Trendy v genetice. 24 (6): 280–288. doi:10.1016 / j.tig.2008.03.008. PMID 18457900.
- ^ Wei L, Levine AS, Lan L (2016). "Homologní rekombinace spojená s transkripcí po oxidačním poškození". Oprava DNA (Amst.). 44: 76–80. doi:10.1016 / j.dnarep.2016.05.009. PMID 27233112.
externí odkazy
(Wayback Machine kopírovat)
- RNA + polymeráza + II v americké národní lékařské knihovně Lékařské předměty (Pletivo)
