Genetické studie o Chorvatech - Genetic studies on Croats
Populační genetika je vědecká disciplína, která přispívá ke zkoumání lidské evoluční a historické migrace. Obzvláště užitečné informace poskytuje výzkum dvou uniparentálních markerů v našem genomu, Y-chromozom (Y-DNA) a mitochondriální DNA (mtDNA), stejně jako autozomální DNA. Údaje z Y-DNA a autosomální DNA naznačují, že Chorvati většinou jsou to potomci Slovanů ze středověku migrační období, podle mtDNA mají genetickou rozmanitost, která zapadá do širší evropské mateřské genetické krajiny, a celkově mají jednotnost s ostatními jižními Slovany z území bývalých Jugoslávie.
Existuje mnoho Paleolitické dobové lokality nacházející se na území Chorvatska, většinou připisované Mousterian fáze v Střední paleolit doba. V období neolitu v Jihovýchodní Evropa byly založeny hlavní kultury jako Vinča, Varna, Starčevo. V Doba bronzová došlo k symbióze mezi Protoindoevropané z Kurganská kultura a autochtonní populace, což vede mimo jiné k tvorběIlyrové. Postupně se mísili a byli asimilováni Římané, Keltové, Avars, a nakonec Slované od 6. století. Další významná migrace proběhla z Bosna a Hercegovina, expanze, která byla ovlivněna Osmanská říše Dobytí od 15. století, stejně jako chorvatská imigrace před a po Světové války I. a II a Chorvatská válka za nezávislost.
DNA chromozomu Y
Prehistorická Y-DNA
Ve studii z roku 2014 ze tří úspěšně vygenerovaných profilů SNP neolitu Starčevo kultura vzorky z Vinkovci, dvě patřily k haploskupině Y-DNA G2a-P15 a jedna k I2a1-P37.2, což by mohlo označovat G2a jako potenciální zástupce šíření zemědělství z Blízkého východu do Evropy, zatímco I2a jako mezolitický substrát v Evropě.[1]
Ve studii z roku 2018 mělo 10 ze 17 vzorků z Chorvatska úspěšné sekvenování Y-DNA; dva vzorky chorvatského neolitu (6005-5786 př. n. l.) ze jeskyně Zemunica patřily raně neolitickým C1a2 a E1b1b1a1b1 Starčevo (5837–5659 př. N. L.) Z Beli Manastir-Popova zemlja do C, raně neolitické Chorvatsko Impressa (5641–5560 př. N. L.) Z Kargaduru do G2a2a1, dva vzorky ze středního neolitu (Sopoty) (5207–4546 př. Pozdně neolitický Sopot (4790–4558 př. N. L.) Z Beli Manastir-Popova zemlja do I, dva Vučedol (2884-2582 př. N. L.) Vzorky z Beli Manastir-Popova zemlja a Vucedol Tell pro R1b1a1a2a2 a G2a2a1a2a a vzorek raně střední doby bronzové (1631-1521 př. N. L.) Od Velikiho Vanika patřil k J2b2a.[2]
Současná Y-DNA
Geneticky, z otcovy strany Y chromozom linie a studie publikované v letech 2003 až 2008 patří většina (> 85%) mužských Chorvatů z Chorvatska k jedné ze tří hlavních evropských Y-DNA haploskupiny - Já (38%[3][4][5]-44%[6]), R1a (27%[6]-34%[3][4][5]) a R1b (12.4%[6]-15%[3][4][5]), zatímco menšina (> 15%) většinou patří do haploskupiny E (9%[6]) a další do haplskupin J (4.4%[6]), N (2%[6]), a G (1%[6]). Podle nejnovějších a rozsáhlých studií zveřejněných v letech 2012 až 2016 patří většina (75%) chorvatských mužů do haploskupin I (38% -43%), R1a (22% -25%) a R1b (8% - 9%), zatímco menšina (25%) patří většinou k haploskupinám E (10%), J (7% -10%), G (2% -4%), H (0,3 - 1,8%) a N (<1%).[7][8]


Haploskupina I mezi Chorvati z Chorvatska je kolem 43,5%,[7] zatímco v Šokci, podskupina Chorvatů z východního Chorvatska, je mezi 44% a 50% stejné haploskupiny.[9][10] Je rozdělena do dvou hlavních pododdělení - subclade I2 (37,7%), typické pro populace Balkánu a východní Evropy, a I1 (5,8%), typické pro populace Skandinávie.[6] Ze subkladu I2 je nejvíce převládající I2a1a (I-P37.2), tj. Jeho subclade I-M423 (18 400-14 000 YBP)> I-Y3104 (14 000-11 400 YBP)> I-L621 (11 400-6 500 YBP)> I-CTS10936 (6 500–5 600 YBP)> I-S19848 (5 600 YBP)> I-CTS4002 (5 600–5 100 YBP)> I-CTS10228 (5 100–3 400 YBP)> I-Y3120 (3 400–2 100 YBP)> I- S17250 (2100 - 1850 YBP)> I-PH908 (1 850 - 1 700 YBP),[11] což je typické pro jihoslovanské populace Jihovýchodní Evropa, nejvyšší v Bosna a Hercegovina (>50%).[5] V Chorvatsku je nejvyšší frekvence pozorována v Dalmácie, vrcholící ve městech Dubrovník (53%) a Zadar (60%),[8] stejně jako jižní ostrovy Vis (44.6%),[8] Brač a Korčula (~ 55%) a Hvar (65%).[6] Četnost je nižší ve městě Osijek (27,6%) na břehu řeky Drava, v západní hornatině Žumberak region (18,2%),[8] a na severních ostrovech Cres (3%) a Krk (27%).[6][3] Nejvyšší frekvence haploskupiny se nachází v Chorvati z Hercegovina (73%).[6] Subclade I1 nebyl nalezen v Osijek a bosenských Chorvatů,[6] ale v Dubrovníku dosáhl vrcholu 8,9%.[8] Velmi vysoká frekvence subkladu I-P37.2 na západním Balkáně klesá ve všech směrech.[12][8]
Populace s haploskupinou I migrovala do Evropy ze Středního východu přibližně před 25 000–13 000 lety. Představuje paleolit a mezolit populace lovců a sběračů v Evropě. Ve srovnání se staršími výzkumy, které tvrdily o prehistorickém autochtonním původu haploskupiny I2 v Chorvatsku a na Balkáně,[poznámka 1] jak již Battaglia a kol. (2009) pozorovali nejvyšší rozptyl haploskupiny v Ukrajina Zupan a kol. (2013) poznamenal, že naznačuje, že dorazil Slovanská migrace z vlasti, která byla na dnešní Ukrajině.[17] Nejnovější výzkum O.M. Utevska (2017) dospěl k závěru, že haploskupina STR haplotypy mají nejvyšší rozmanitost na Ukrajině s výsledkem STR předků "DYS448 = 20" zahrnující "Dněpr -Karpaty „shluk, zatímco mladší odvozený výsledek“ DYS448 = 19 “zahrnující„ balkánský shluk “, který převládá mezi jižními Slovany.[18] Tento „balkánský klastr“ má také nejvyšší rozptyl na Ukrajině, což naznačuje, že velmi vysoká frekvence na západním Balkáně je způsobena zakladatelský efekt.[18] Utevska vypočítal, že divergence shluku STR a její sekundární expanze ze středního toku řeky Dněpr nebo z Východní Karpaty směrem na Balkánský poloostrov se stalo přibližně před 2 860 ± 730 lety, což souvisí s dobami před Slovany, ale hodně po úpadku Kultura tripolyů.[18] Přesněji řečeno, klastr je reprezentován jediným SNP, I-PH908, známým jako I2a1a2b1a1a1c v ISOGG fylogenetický strom (2019) a podle YFull YTree se vytvořil a měl TMRCA přibližně 1 850 - 1 700 YBP.[11] Ačkoli se má za to, že I-L621 mohl být přítomen v kultuře Cucuteni – Trypillia,[19] až dosud byl nalezen G2a,[20] a další subclade I2a1a1-CTS595 byl přítomen v Badenská kultura kalcholitické karpatské pánve.[19][21] Ačkoli je dominantní mezi moderními slovanskými národy na území bývalých balkánských provincií římská říše, dosud nebyl nalezen mezi vzorky z doby římské a téměř chybí v současné populaci Itálie.[22] Bylo nalezeno v kosterních pozůstatcích s artefakty označujícími vůdce z Maďarští dobyvatelé z Karpatská pánev od 9. století, součást západní euroasijsko-slovanské složky Maďarů.[22][19] Podle Fóthi et al. (2020), distribuce předků jako I-CTS10228 mezi současnými dopravci naznačuje rychlý rozvoj od Jihovýchodní Polsko, souvisí hlavně se Slovany a „největší demografická exploze nastala na Balkáně“.[22] Nejstarší archeogenetický vzorek doposud je Sungir 6 (~ 900 YBP) blízko Vladimir, Rusko který patřil k subkladu I-S17250> I-Y5596> I-Z16971> I-Y5595> I-A16681.[23][24]

R1a1a1-M17 (22,1% - 25,6%) je druhou nejvíce převládající haploskupinou.[7] Haploskupina R-M17 v Chorvatsku je většinou rozdělena do dvou subclades, R-M558, která převládá (19,2%), a R-M458 (4,9%), zatímco R-Z282 je vzácný (1,2%).[8] Má nejvyšší frekvenci v severní části Chorvatska (29,1%) a téměř stejnou nejnižší frekvenci ve východní části (18,6%), jižní (19,1%) a západní (20%) oblasti Chorvatska.[7] Nejvyšší místní frekvence R1a1a1-M17 byla zjištěna u Chorvatů z Osijeku (39%),[6] Žumberak (34,1),[8] a na severních ostrovech Krk (37%) a Cres (56,5%),[3][8] jsou podobné hodnotám ostatních Slovanů, jako jsou Slovinci, Češi a Slováci. Četnost je nižší v Šokci z východního Chorvatska (16%),[9][10] ve městech Zadar (4%) a Dubrovník (13,4%) v Dalmácii, stejně jako na jižních ostrovech Hvar (8%), Vis (17%) a střední v Korčule (20%) a Brač (25%) ).[3][8] U bosenských Chorvatů je frekvence podobná jako u ostatních jižních Slovanů (12%).[6] Subclade R-M558 je častější mezi Východní Slované ve východní Evropě a Volga-Ural regionu, zatímco R-M458 mezi Západní Slované ve střední a východní Evropě. Oba jsou přítomny v „informativní frekvenci u balkánských populací se známým slovanským dědictvím“.[25] R-M558 subclade CTS1211 byl také nalezen mezi maďarskými dobyvateli, což naznačuje míchání a asimilaci Slovanů mezi Maďary.[19]
Nejvyšší frekvence haploskupiny Haploskupina R1b (7,9% -9,1%), který je v Chorvatsku rozdělen na několik subclades (hlavně R-L23 a jeho subclade R-U152 ), má v severní (10,9%) a střední (11,8%) oblasti Chorvatska,[7] zatímco místně v Chorvatsku z ostrova Krk (16,2%) a Dugi Otok (25%),[3][8] a Žumberak (11,3%),[8] zatímco na jižních ostrovech, ve městě Dubrovník (3,9%) a v bosenských Chorvatích téměř chybí (1-6%),[3][6][8] nebo jako v Osijeku nebyl nalezen.[6] K těmto dvěma haploskupinám (R1a a R1b) jsou připojeny Protoindoevropané migrace z euroasijské oblasti asi před 5 000 lety,[12] s R1a zejména k migraci slovanského obyvatelstva.[5][8] Jejich frekvence ukazuje gradaci sever-jih a opačnou distribuci frekvence než haploskupina I-P37.2,[12] a nejvyšší frekvence je pozorována v severním, západním a východním Chorvatsku.[3][6]
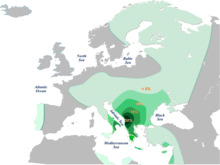
Z haploskupiny E (9,8% -10,6%) u Chorvatů je nejčastější subclade E1b1b1a1b-V13 (6,7%), zatímco E1b1b1a3-M149 a E1b1b1c-M123 byly také nalezeny v malém počtu (1,1%).[6] E-V13 je typický pro populace jihovýchodní Evropy, s nejvyššími vrcholy u kosovských Albánců (44%), a vysoký je také u Makedonců, Řeků, Rumunů, Bulharů a Srbů.[5] Nejvyšší frekvence v chorvatské pevnině byla nalezena v Žumberaku (18,2%)[8] a Osijek (10,3%),[6] na středních ostrovech Dugi Otok (15,9%) a Ugljan (13,2%) a také na jižních ostrovech Vis (23,4%) a Mljet (15.4%).[8] Na severních ostrovech Cres (3%)[8] a Krk (6,8%) byl podobný jako na ostatních jižních ostrovech (3,7–4,3%).[3] U bosenských Chorvatů byla frekvence stejná jako u Chorvatů z Chorvatska (8,9%).[6] Subclady J1 jsou v Chorvatsku vzácné, zatímco J2 jsou vyšší u Chorvatů z Chorvatska, vrcholí u Chorvatů z Osijeku (10,2%) a středních ostrovů Ugljan (10,2%) a Pašman (16,6%), stejně jako severní ostrov Krk (10,8%) ) a Cres (14,1%),[3][6][8] než u bosenských Chorvatů (oba 1,1%).[6] Subclade G2a-P15 u chorvatských i bosenských Chorvatů se vyskytuje v malém počtu (1,1%),[6] ale vrcholí lokálně v severovýchodním městě Osijek (13,8%),[6] a jižní ostrovy Mljet (15,4%), Korčula (10,4%), Brač (6%) a také severní ostrov Cres (7%).[3][8] Haploskupina E a J souvisí s neolitickou migrací populace z Anatolie po LGM, která s sebou přinesla domestikaci divokých zvířat a rostlin. Konkrétně subclade haploskupiny E pravděpodobně vznikl lokálně na Balkáně ne dříve než před 8 000–10 000 lety. Tyto haploskupiny ukazují gradaci jih-sever.[12] Haploskupina G mohla být v Evropě přítomna během LGM nebo populace s některými jejími subclades dorazila s časnými zemědělci.[12]
Haploskupina N subklady jsou v Chorvatsku vzácné (0,6% - 2,2%).[6][8] To je velmi časté v Dálný východ, jako Sibiř a Čína, zatímco v Evropě jsou to Finové (60%) a pobaltské země (45%). Neobvykle pro evropské populace další středoasijsko-sibiřská haploskupina P (tj. Q ) byl nalezen na neobvykle vysokých frekvencích kvůli efektu zakladatele na ostrovech Hvar (14%) a Korčula (6%).[3]
Abstrakt a data
Region současného Chorvatska byl součástí širšího balkánského regionu, který mohl sloužit jako jeden z několika refugia během LGM, zdrojové oblasti pro rekolonizaci Evropy během post-glaciálního období a holocénu (před 10 000 lety).[6][12] Východní pobřeží Jaderského moře bylo mnohem dále na jih.[4] Severní a západní část moře byly stepi a pláně, zatímco moderní chorvatské ostrovy (bohaté na paleolitická archeologická naleziště) byly kopce a hory.[4][6] Tento region měl zvláštní roli při strukturování evropského, zejména mezi slovanským, otcovským genetickým dědictvím, charakterizovaným převahou R1a a I a nedostatkem linií E.[5] Současná genetická rozmanitost ostrovních populací se vyznačuje silnou izolací a endogamie.[26]
V následující tabulce je uvedena dosud nejrozsáhlejší studie (2012) o populaci v Chorvatsku. Jedná se o národní odkaz DNA databáze ze 17 loci systém, který získal Y-STR haplotypy byly předpovídány v odhadované (s pravděpodobností nad 90%) Y-SNP haploskupiny.[7] Subpopulace byly rozděleny do pěti regionů, přičemž subpopulace vykazovala silnou podobnost a homogenitu otcovského genetického přínosu, s výjimkou subpopulace z jižního Chorvatska, která vykazovala mírný rozdíl. Kromě vysokého stupně celkové homogenity existují gradientní podobnosti se středoevropským klastrem (germánský, slovanský, maďarský) a jihoevropským klastrem (bosenský, srbský, bulharský, makedonský, rumunský, albánský, jihoitalský) od severu k jihu (a na východ a na západ).[7]
| Populace | Vzorky | Zdroj | I2a (xI2a1) | R1a | E1b1b1-M35 | R1b | I1 | J2b | G2a | H | J2a1h | J1 | J2a1b | E1b1a1-M2 | G2c | I2a1 | I2b1 | I2b (xI2b1) | J2a1-bh | L | N | Q | T |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Celkově Chorvatsko | 1,100 | Mršić a kol. (2012) | 37.7% (415/1100) | 22.1% (243/1100) | 10.6% (117/1100) | 7.9% (87/1100) | 5.8% (64/1100) | 3.7% (41/1100) | 2.7% (30/1100) | 1.8% (20/1100) | 1.2% (13/1100) | 1.1% (12/1100) | 1% (11/1100) | <1 | <1 | <1 | <1 | <1 | <1 | <1 | <1 | <1 | <1 |
| Střední Chorvatsko | 220 | Mršić a kol. (2012) | 31.8% (70/220) | 23.6% (52/220) | 11.8% (26/220) | 10.4% (23/220) | 5% (11/220) | 5% (11/220) | 3.6% (8/220) | 1.3% (3/220) | 0.4% (1/220) | 2.2% (5/220) | 0.9% | 0 | 0 | 0.4% | 0.9% | 0.9% | 0 | 0.4% | 0.9% | 0 | 0 |
| Severní Chorvatsko | 220 | Mršić a kol. (2012) | 25.4% (56/220) | 29.1% (64/220) | 10.9% (24/220) | 10.4% (23/220) | 4.1% (9/220) | 5% (11/220) | 3.1% (7/220) | 5% (11/220) | 0.4% (1/220) | 0 | 0.4% (1/220) | 0.4% | 0 | 0 | 2.2% | 0.9% | 0 | 0.4% | 0.4% | 0 | 1.3% |
| Východní Chorvatsko | 220 | Mršić a kol. (2012) | 40% (88/220) | 18.6% (41/220) | 11.3% (25/220) | 8.2% (18/220) | 5.9% (13/220) | 2.7% (6/220) | 1.8% (4/220) | 0.9% (2/220) | 2.2% (5/220) | 2.7% (6/220) | 1.3% | 0 | 0 | 0 | 0.4% | 0 | 0.4% | 0 | 0.9% | 1.8% | 0.4% |
| Západní Chorvatsko | 220 | Mršić a kol. (2012) | 36.8% (81/220) | 20% (44/220) | 12.7% (28/220) | 5.9% (13/220) | 8.6% (19/220) | 3.2% (7/220) | 3.2% (7/220) | 1.8% (4/220) | 1.8% (4/220) | 0.4% (1/220) | 1.8% | 0 | 0.4% | 0 | 0.4% | 0 | 0.4% | 0 | 0.9% | 0.4% | 0.9% |
| Jižní Chorvatsko | 220 | Mršić a kol. (2012) | 54.5% (120/220) | 19.1% (42/220) | 6.3% (14/220) | 4.5% (10/220) | 5.4% (12/220) | 2.7% (6/220) | 1.8% (4/220) | 0.4% (1/220) | 0.9% (2/220) | 0 | 0.4% | 0 | 0 | 0 | 0.4% | 0 | 0.9% | 0.4% | 0 | 1.3% | 0.4% |
| Záhřeb a Chorvatsko | 239 | Purps a kol. (2014)[27] | 36.1% | 23.8% | 6.4% | 13.9% | 3.9% | 2.1% | n / a | n / a | 1.9% | 0.9% | n / a | n / a | n / a | n / a | n / a | n / a | n / a | n / a | n / a | n / a | n / a |
| Chorvatsko | 720 | Šarac a kol. (2016) | 32.5% | 25.6% | 9.8% | 9.1% | 4.1% | 5.0% | 4.4% | 0.3% | 2.7% | 0.5% | 1.0% | 0.4% | 0 | 0.3% | 0.8% | 0.5% | 1.0% | 0 | 0.6% | 0.9% | 1.2% |
Mitochondriální DNA
Prehistorická mtDNA
Ve studii Y-DNA a mtDNA z roku 2014 byl jeden mezolitický vzorek datován 6080-6020 př. Vela Spila u Vela Luka na Islandu Korčula patřil k haploskupině mtDNA U5b2a5 běžné v komunitách lovců a sběračů, zatímco dalších jedenáct neolitických Starčevo kultura vzorky datované kolem 6000–5400 př Vinkovci byly přiřazeny haploskupiny J1c, K1a, T2b, HV0, K, V, V6, které odhalují podobnou rozmanitost mtDNA a sdílený původ v počátečních farmářských populacích z Panonská pánev a populace střední Evropy LBK, doprovázené redukcí mezolitického mtDNA substrátu.[1]
Předběžné výsledky studie mtDNA z roku 2016, která bude zahrnovat přibližně 30 vzorků z neolitu a 5 vzorků ze starší až pozdní doby bronzové, na 5 staro chorvatských drobné kosti (3 neolitické Cardial Impresso z jeskyně Zemunica poblíž Bisko, 1 střední neolit Danilo kultura z jeskyně Zidana poblíž Staničići Žumberački, 1 střední / pozdní doba bronzová z jeskyně Jazinka poblíž Nečven ) označil haploskupiny mtDNA K2 a K1b1a, H1e / H41, H1b pro neolitické vzorky podobné Raní evropští zemědělci (EEF) a moderní Sardinians a Jižní Evropané Zatímco haploskupina HV nebo H4 pro vzorek z doby bronzové je obdobou moderní chorvatské a balkánské populace, ale bez jasných důkazů o souvislosti s indoevropskou migrací.[28]
Studie z roku 2018, která zahrnovala 17 vzorků z Chorvatska; Mesolithic (7308-7027 BCE) z Vela Spila do U5b2b, tři vzorky chorvatského kardinálního neolitu (6005-5751 BCE) z jeskyně Zemunica do H1, K1b1a a N1a1, raně neolitický Starčevo (5837-5659 BCE) z Beli Manastir-Popova zemlja do U8b1b1, dva raně neolitické chorvatské Impressa (5670-5560 př. n. l.) vzorky z Kargaduru do H5a a H7c, dva středně neolitické sopoty (5207-4546 př. n. l.) z Osijeku do U5a1a2 a H10, dva pozdně neolitické sopoty (4790- 4536 př. N. L.) Vzorky z Beli Manastir-Popova zemlja do U5b2b a N1a1, eneolit (3710–3360 př. N. L.) Z Radovanců do J1c2, tři Vučedol (3000-2582 př. N. L.) Vzorky z Beli Manastir-Popova zemlja a Vucedol Tell pro T2e, T2c2 a U4a, starší doby střední doby bronzové (1631-1521 př. N. L.) Od Veliki Vanik po I1a1 a pozdní doby bronzové (805-761 př. ) vzorek z jeskyně Jazinka patřil HV0e.[2]
Středověká mtDNA
Studie mtDNA z roku 2011 na 27 raně středověkých kosterních pozůstatcích v Naklicích poblíž Omiši v jižní Dalmácii ukázalo, že 67% patřilo haploskupině H, 18% J, 11% U5 a 4% HV.[29] Studie mtDNA z roku 2015 o středověkých kosterních pozůstatcích v roce 2015 Šopot (14.-15. Století) a Ostrovica (9. století) v Severní Dalmácii potvrdilo, že profily zděděné mateřskou linií se nelišily ani mezi lokalitami Ostrovica a Šopot, ani mezi středověkou a moderní populací, přičemž vykazovaly stejnou prevalenci haploskupiny (H, J, U) u středověkých i současných populací.[30] Studie mužské kostry z roku 2014 nalezená v Rozdělit z pozdní doby římské (datováno 1760 ± 80 YBP) ukázalo, že patří do haploskupiny H.[31]
Současná mtDNA

Geneticky na mateřské X chromozom většina (> 65%) chorvatských Chorvatek (pevniny a pobřeží) patří do tří z jedenácti hlavních evropských haploskupin mtDNA - H (25.5-45%), U (18.85-22%), J (7,5-11%), zatímco velká menšina (> 35%) patří k mnoha dalším menším haploskupinám.[32][33]
Ve všech studiích je haploskupina H nejčastější (45% v roce 2014) mateřskou haploskupinou na chorvatské pevnině (kontinentální) a na pobřeží, ale v poslední studii z roku 2020 je nižší frekvence 25,5% kvůli rozdílům v nomenklatuře primárně R / R0 (7,5%) linie.[33] Nejvyšší frekvence v Chorvatsku pozorovaná u populace ostrova Korčula (60,2%) a Mljet (73.5%),[32] zatímco nejnižší frekvence na ostrovech Cres (27.7%),[34] a Hvar (27.8%).[35] Jedná se o dominantní evropskou haploskupinu.[32] Zvýšená frekvence subhaploskupiny H1b v Mljet (30,9%), která je v jiných studiích vzácná, je typickým příkladem efektu zakladatele - migrace z nejbližší pobřežní oblasti a mikroevoluční expanze na ostrově.[32]
Haploskupina U (18,85% v roce 2014,[36] 22% v roce 2020[33]) je většinou reprezentován jeho subclade U5 což je druhá nejčastější haploskupina s 11,6% na pevnině a 10,4% na pobřeží s podobnou frekvencí na ostrovech Brač (8.6%), Krk (11,3%) a Hvar (14,8%), zatímco nejnižší na Korčule (2%).[35] Celkově je haploskupina U, včetně jejích subclades jako U5, nejčastější ve městě Dubrovník (19,9%) a ostrovy Lastovo (23,5%) a Cres (29,4%).[34][32] Je to nejstarší evropská haploskupina (koalescenční věk kolem 50 000 YBP) a její subclade U5 tvoří většinu rozmanitosti haploskupin v Evropě. Vysoká frekvence U4 v Lastově (11,8%) označuje efekt zakladatele.[32]
Haploskupina J je třetí nejčastější haploskupinou (7,5% v roce 2020[33]), s 11,9% na pevnině, ale pouze 3,1% na pobřeží, avšak ostrovy měly vyšší frekvenci než populace na pobřeží Korčula (6,1%), Brač (8,6%), Krk (9%), Hvar (9,3%),[35] vrchol v Žumberak (12.34%)[36] a Lastovo (19,6%),[32] zatímco v Cres téměř úplně chybí.[34]
Haploskupina T je třetí nebo čtvrtá nejčastější haploskupina (10% v roce 2020[33]). Jeho subclade T2 má podobnou frekvenci 3,1-5,8% jak u pobřežní, tak u pevninské i ostrovní populace, s výjimečným vrcholem na ostrově Hvar (12%),[35][34] nicméně celková haploskupina T má nižší frekvenci v Mljet (1,5%), Lastovo (3,9%) a Dubrovník (2,3).[32]
Haploskupina K. (7,5% v roce 2020[33]) má průměrnou frekvenci 3,6% na pevnině a 6,3% na pobřeží, chybí v Lastovu[32] a má nejnižší frekvenci na ostrovech Cres (3,36%)[34] a Hvar (3,7%), zatímco nejvyšší na ostrově Brač (9,5%).[35]
Haploskupina PROTI (pod HV > HV0) je mladší sesterský subtyp haploskupiny H,[32] a má téměř stejnou minimální a maximální frekvenci (4 až 5,7%) v kontinentální i ostrovní populaci, s výjimkou Korčuly (8,2%),[35] stejně tak nižší frekvence v Mljet (1,5%), Lastovo (2%) a Dubrovník (3,3%).[32]
Haploskupina Ž frekvence na pevnině a na pobřeží je mezi 2,2–4,2%, zatímco mezi ostrovními populacemi 1,9–3,1%, s výjimkou Krku (7,5%),[35] a Cres (12,6%).[34] Na ostrovech Mljet a Lastovo je mezi 4,4-5,9%, zatímco v Dubrovníku téměř chybí (0,6%).[32]
Další haploskupina mtDNA s významnými místními vrcholy jsou: subklady VN s nízkou frekvencí na pevnině a na pobřeží (0,4–2,1%), ale průměrné (4,1–4,6) na ostrovech a vysoké v Dubrovníku (7,7%)[32] a Brač (10,5%).[35] Haploskupina N1a v Cresu (9,24%) je dosud nejsevernějším nálezem tohoto odvětví v Evropě a haplotypy naznačují relativně nedávný zakladatelský efekt.[34] Je to charakteristická haploskupina raných farmářů.[1] Haploskupina F což téměř chybí (pouze 0,2–0,4% na pevnině), ale vrcholí na 8,3% na Hvaru.[35][36] Haploskupina Já v Krku (11,3%), které se subhaploskupiny oddělily kolem LGM.[37]
Abstrakt a data
Po celá desetiletí byly chorvatské ostrovní populace studovány kvůli jejich izolaci, která dokáže sledovat mikroevoluční procesy a porozumět evolučním silám, jako je genetický drift (specifický genetický výraz), efekt zakladatele a úzká místa populace (zmenšení velikosti populace), které formovaly současnou populaci . Dosavadní výsledky naznačují, že genetický tok a příliv žen na ostrovy byly omezené.[32] Mírný genetický izolát lze také zvážit pro kontinentální populaci horské oblasti Žumberak protože měli volnou afinitu k Uskokové navrhovaná oblast původu (Bosňané, Hercegovinci, jižní Chorvaté) nebo k jejich současným nejbližším sousedům (severní Chorvaté a Slovinci).[36] Na příkladu populace ostrova Krk ukázala analýza mtDNA s vysokým rozlišením důkaz, že osady Omišalj, Vrbnik, a Dobrinj jsou ve společném klastru raných slovanských sídel, zatímco regiony Poljica a Dubašnica jsou samostatným klastrem založeným slovanskými a Vlachs nebo Morlachs migranti z vnitrozemského vnitrozemí, kteří přijeli v 15. století.[37] Na příkladu populace ostrova Mljet lze vnímat demografické a historické události, jako je využití ostrova pro karanténní stanici, zatímco v rámci Vis a Lastovo praxe příbuznosti a příbuzenské plemenitby kvůli nedostatku genetické rozmanitosti jsou vhodné pro geneticko-epidemiologický výzkum.[32]
V analýze mtDNA z roku 2004 byl jeden shluk tvořen populacemi z ostrovů Hvar, Krk a Brač a druhý shluk zahrnoval chorvatskou pevninu a chorvatské pobřeží, zatímco ostrov Korčula se vyznačoval mimořádně vysokou frekvencí haploskupiny H.[35] V interpopulační analýze mtDNA v roce 2009 PCA analýza subhaploskupin, ostrovních populací z Krku, Ugljan, Korčula, Brač, Hvar byly seskupeny, což naznačuje blízké mateřské linie, s Visem v jejich blízkosti, ale Cres (prevalence U2, W, N1a) a Rab (U4, H6, J1c) měly oddělené odlehlé polohy od klastru i od sebe navzájem.[34] V analýze PCA z roku 2014 mtDNA se populace z východního a jižního Chorvatska shlukovaly společně s Bosnou a Hercegovinou, zatímco západní a severní Chorvatsko se Slovinskem. Jelikož slovinská populace netvoří klastr jihovýchodní Evropy, považuje se to za možný vstup z různých migračních vln Slovanů v EU Středověk.[36]
| Populace | Vzorky | Zdroj | H | HV | J | T | K. | U * | U1 | U2 | U3 | U4 | U5 | U6 | U7 | U8 | R | N | Já | Ž | X | jiný |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Chorvatsko | 488 | Šarac a kol. (2014) | 45.29 | 4.07 | 9.83 | 5.98 | 4.30 | 0 | 1.23 | 2.66 | 1.43 | 2.66 | 10.06 | 0.20 | 0.41 | 0.20 | 0 | 0.82 | 2.61 | 1.84 | 1.84 | 4.29 |
| Východní Chorvatsko | 61 | Šarac a kol. (2014) | 49.18 | 11.48 | 9.84 | 3.28 | 4.92 | 0 | 0 | 0 | 1.64 | 1.64 | 9.84 | 0 | 0 | 0 | 0 | 1.64 | 0 | 4.92 | 1.64 | 0 |
| Severní Chorvatsko | 155 | Šarac a kol. (2014) | 41.77 | 5.06 | 14.56 | 10.76 | 3.16 | 0 | 0.63 | 1.90 | 0.63 | 3.16 | 11.39 | 0.63 | 0 | 0.63 | 0 | 0 | 1.27 | 2.53 | 0.63 | 0.63 |
| Západní Chorvatsko | 209 | Šarac a kol. (2014) | 46.41 | 11.48 | 6.70 | 2.39 | 5.74 | 0 | 2.39 | 4.31 | 1.91 | 2.39 | 6.22 | 0 | 0.96 | 0 | 0 | 0.96 | 4.78 | 0 | 2.39 | 0 |
| Jižní Chorvatsko | 63 | Šarac a kol. (2014) | 49.21 | 3.17 | 7.94 | 9.52 | 3.17 | 0 | 0 | 1.59 | 1.59 | 3.17 | 12.70 | 0 | 0 | 0 | 0 | 1.59 | 1.59 | 1.59 | 3.17 | 0 |
| Chorvatsko | 200 | Barbarić a kol. (2020) | 25.5 | 11.5 | 7.5 | 10 | 7.5 | 0 | 2 | 4 | 2.5 | 2.5 | 10 | 0 | 1 | 0 | 7.5 | 2 | 3 | 1 | 2 | 0.5 |
Autozomální DNA
Podle roku 2013 autosomální IBD průzkum „nedávných genealogických předků za posledních 3 000 let v kontinentálním měřítku“, mluvčí srbochorvatského jazyka sdílejí velmi vysoký počet společných předků datovaných do migrační období přibližně před 1 500 lety s Polskem a Rumunskem a Bulharskem mimo jiné ve východní Evropě. Dochází k závěru, že je způsoben Hunnic a slovanská expanze, což byla „relativně malá populace, která se rozšířila na velkou geografickou oblast“, zejména „expanze slovanských populací do oblastí s nízkou hustotou obyvatelstva začínající v šestém století“ a že je „velmi shodná s moderní distribuce slovanských jazyků ".[38] Podle Kushniarevich et al., Hellenthal et al. Analýza IBD z roku 2014 také zjistila „vícesměrné příměsové události mezi východoevropany (slovanskými i neslovanskými), které se pohybovaly kolem 1 000–1 600 YBP“, což se shoduje s „navrhovaným časovým rámcem slovanské expanze“.[39] Analýza IBD z roku 2015 zjistila, že jižní Slované mají menší blízkost k Řekům než k východním a západním Slovanům a že existují „rovnoměrné vzorce sdílení IBD mezi obyvateli východ-západ -„ slovanskými “populacemi (Maďaři, Rumuni a Gagauz ) –A jižní Slované, tj. Napříč oblastí předpokládaných historických pohybů lidí, včetně Slovanů. “Mírný vrchol sdílených segmentů IBD mezi Slovany na jihu a na východě a západě naznačuje společný„ slovanský původ “.[39] Porovnání IBD analýz populací západního Balkánu a Středního východu z roku 2014 také zjistilo zanedbatelný tok genů mezi 16. a 19. stoletím během Islamizace Balkánu.[40]
Podle autosomální analýzy západního Balkánu z roku 2014 vykazuje chorvatská populace genetickou uniformitu s ostatními jihoslovanskými populacemi. Chorvaté a Bosňané měli blíže k východoevropským populacím a do značné míry se překrývali s Maďary ze střední Evropy.[40] V analýze z roku 2015 vytvořili západní jihoslovanský klastr s Bosňany a Slovinci ve srovnání s východním klastrem tvořeným Makedonci a Bulhary se Srby uprostřed. Západní skupina má sklon k Maďarům, Čechům a Slovákům, zatímco východní skupina k Rumunům a do jisté míry Řekům.[39] V analýze roku 2018 slovinský obyvatelstva se chorvatské obyvatelstvo opět seskupilo se Slovinci, Maďary a bylo blízko češtiny. Populace Chorvatska většinou sdílí společné předky s východními, západními a jižními Evropany a nemá téměř žádný vztah k izolovaným populacím, jako je Sardinians a Baskové.[40][41]
Podle 2016 celý sekvenování exome 176 osob z ostrov Vis to bylo potvrzeno izolovat stav populace ostrova, a odhalilo to „vzor mutací ztráty funkce, který se podobá stopám adaptivního vývoje“.[42]

Analýza příměsí autosomálních SNP v globálním kontextu na úrovni rozlišení 7 předpokládaných populací předků podle Kovačeviće a kol. (2014)

Analýza hlavních komponent (PC) variací autozomálních SNP u populací západního Balkánu v euroasijském kontextu podle Kovačeviće a kol. (2014)
Analýza příměsí na úrovni rozlišení 6 předpokládaných populací předků podle Kushniarevich et al. (2015)
Genetická struktura v evropském kontextu podle tří genetických systémů atDNA (A), YDNA (B) a mtDNA (C) podle Kushniarevich et al. (2015)

Graf PC1vsPC2 založený na datech SNP celého genomu podle Kushniarevich et al. (2015)
Viz také
- Bílí Chorvaté
- Hypotézy o původu Chorvatů
- Genetické studie o Srbech
- Genetické studie o Bosňácích
- Genetické studie o Bulharech
- Y-DNA haploskupiny podle etnické skupiny
- Haploskupiny Y-DNA v populacích Evropy
Poznámky
- ^ A b C Szécsényi-Nagy a kol. 2015.
- ^ A b Mathieson a kol. 2018.
- ^ A b C d E F G h i j k l m Barać a kol. 2003.
- ^ A b C d E Rootsi a kol. 2004.
- ^ A b C d E F G Peričić a kol. 2005.
- ^ A b C d E F G h i j k l m n Ó str q r s t u proti w X y z aa Battaglia a kol. 2008.
- ^ A b C d E F G Mršić a kol. 2012.
- ^ A b C d E F G h i j k l m n Ó str q r s t Šarac a kol. 2016.
- ^ A b Jurić, Ivan (2006). "Genetičko podrijetlo šokačkih rodova na području Vinkovaca" Genetický původ rodů Šokci v oblasti Vinkovci. Godišnjak Ogranka Matice Hrvatske Vinkovci (v chorvatštině). 24: 143–160. Citováno 13. srpna 2020.
- ^ A b Jurić, Ivan (2008). "Genetičko podrijetlo šokačkih rodova" [Genetický původ rodů Šokci]. Godišnjak Za Kulturu, Umjetnost I Društvena Pitanja (v chorvatštině). 25: 193–208. Citováno 13. srpna 2020.
- ^ A b C „I-PH908 YTree v8.06.01“. YFull.com. 27. června 2020. Citováno 17. července 2020.
- ^ A b C d E F Primorac a kol. 2011.
- ^ „I-P37 YTree v6.07.08“. YFull.com. 10. listopadu 2018. Citováno 11. listopadu 2018.
- ^ „I2a Y-Haplogroup - Výsledky: I2a2a-Dinaric“. DNA rodokmenu. Citováno 11. listopadu 2018.
Ken Nordtvedt rozdělil I2a2-M423-Dinaric na Din-N a Din-S. Din-N je starší než Din-S. N = severně od Dunaje a S = jižně od Dunaje ... 8. května 2007: Dinaric I1b1 a DYS 448. DYS448 19 pro S a 20 pro N.
- ^ Bernie Cullen (22. srpna 2016). „Odkaz na strom I-L621 zobrazující hlavní klastry STR (aktualizováno)“. i2aproject.blogspot.com. Blogger. Citováno 3. dubna 2019.
- ^ „Y-DNA Haploskupina I a její subclady - 2018“. ISOGG. 1. listopadu 2018. Citováno 11. listopadu 2018.
- ^ Zupan a kol. 2013.
- ^ A b C Utevska 2017, str. 219–226, 302.
- ^ A b C d Neparáczki, Endre; et al. (2019). „Y-chromozomové haploskupiny z Hunu, Avaru a dobyvatelé maďarských nomádských lidí z Karpatské kotliny“. Vědecké zprávy. Výzkum přírody. 9 (16569): 16569. doi:10.1038 / s41598-019-53105-5. PMC 6851379. PMID 31719606.
- ^ Mathieson, Iain (21. února 2018). „Genomická historie jihovýchodní Evropy“. Příroda. 555 (7695): 197–203. doi:10.1038 / příroda25778. PMC 6091220. PMID 29466330.
- ^ Lipson, Mark (2017). „Paralelní starověké genomové transekty odhalují složitou populační historii raných evropských farmářů“. Příroda. 551 (7680): 368–372. doi:10.1038 / příroda24476. PMC 5973800. PMID 29144465.
- ^ A b C Fóthi, E .; Gonzalez, A .; Fehér, T .; et al. (2020), „Genetická analýza mužských maďarských dobyvatelů: evropské a asijské otcovské linie dobytých maďarských kmenů“, Archeologické a antropologické vědy, 12 (1), doi:10.1007 / s12520-019-00996-0
- ^ Sikora, Martin; Seguin-Orlando, Andaine; Sousa, Vitor C; Albrechtsen, Anders; Korneliussen, Thorfinn; Ko, Amy; Rasmussen, Simon; Dupanloup, Isabelle; Nigst, Philip R; Bosch, Marjolein D; Renaud, Gabriel; Allentoft, Morten E; Margaryan, Ashot; Vasiljev, Sergej V; Veslovskaja, Elizaveta V; Borutskaya, Svetlana B; Deviese, Thibaut; Comeskey, Dan; Higham, Tom; Manica, Andrea; Foley, Robert; Meltzer, David J; Nielsen, Rasmus; Excoffier, Laurent; Mirazon Lahr, Marta; Orlando, Ludovic; Willerslev, Eske (2017). „Starověké genomy ukazují sociální a reprodukční chování raných mladopaleolitických sekaček“. Věda. 358 (6363): 659–662. doi:10.1126 / science.aao1807. PMID 28982795.
- ^ „I-A16681 YTree v8.06.01“. YFull.com. 27. června 2020. Citováno 17. července 2020.
- ^ Underhill, Peter A. (2015), „Fylogenetická a geografická struktura haploskupiny Ya chromozomu R1a“, European Journal of Human Genetics, 23 (1): 124–131, doi:10.1038 / ejhg.2014.50, PMC 4266736, PMID 24667786
- ^ Vitart a kol. 2006.
- ^ Šehović a kol. 2018.
- ^ Fernandes a kol. 2016.
- ^ Ljubković a kol. 2011.
- ^ Bašić a kol. 2015.
- ^ Sutlović a kol. 2014.
- ^ A b C d E F G h i j k l m n Ó Šarac a kol. 2012.
- ^ A b C d E F Barbarić a kol. 2020.
- ^ A b C d E F G h Jeran a kol. 2009.
- ^ A b C d E F G h i j Cvjetan a kol. 2004.
- ^ A b C d E Šarac a kol. 2014.
- ^ A b Havaš Auguštin a kol. 2012.
- ^ Ralph a kol. 2013.
- ^ A b C Kushniarevich a kol. 2015.
- ^ A b C Kovačević a kol. 2014.
- ^ Delser a kol. 2018.
- ^ Jeroncic a kol. 2016.
- ^ Samotný SNP I-P37 vytvořil přibližně 20 tisíc YBP a měl TMRCA 18 tisíc YBP podle YFull,[13] protože byl příliš starý a rozšířený jako SNP na argumentaci autochtonie, starý výzkum používal zastaralou nomenklaturu. Podle projektu „I-P37 (I2a)“ na DNA rodokmenu, divergence na STR markeru DYS448 20> 19 je hlášena od roku 2007,[14] zatímco SNP, který definuje klastr STR Dinaric-South, I-PH908, je hlášen od roku 2014.[15] SNP I-PH908 v ISOGG fylogenetický strom je pojmenován jako I2a1a2b1a1a1c,[16] zatímco tvořil a měl TMRCA přibližně 1 800 YBP podle YFull.[11]
Reference
- Y-DNA
- L. Barać; et al. (2003). „Y chromozomální dědictví chorvatské populace a jejích ostrovních izolátů“ (PDF). European Journal of Human Genetics. 11 (7): 535–42. doi:10.1038 / sj.ejhg.5200992. PMID 12825075. S2CID 15822710.
- S. Rootsi; et al. (2004). „Fylogeografie Y-chromozomové haploskupiny I odhaluje odlišné domény prehistorického genového toku v Evropě“ (PDF). American Journal of Human Genetics. 75 (1): 128–137. doi:10.1086/422196. PMC 1181996. PMID 15162323.
- M. Peričić; et al. (2005). „Fylogenetická analýza jihovýchodní Evropy s vysokým rozlišením sleduje hlavní epizody otcovského toku genů mezi slovanskými populacemi“. Molekulární biologie a evoluce. 22 (10): 1964–75. doi:10.1093 / molbev / msi185. PMID 15944443.
- V. Vitart; et al. (2006). „3000 let samoty: extrémní diferenciace na ostrovních izolátech v Dalmácii v Chorvatsku“. European Journal of Human Genetics. 14 (4): 478–487. doi:10.1038 / sj.ejhg.5201589. PMID 16493443.
- V. Battaglia; et al. (2008). „Y-chromozomální důkazy o kulturní difúzi zemědělství v jihovýchodní Evropě“. European Journal of Human Genetics. 17 (6): 820–830. doi:10.1038 / ejhg.2008.249. PMC 2947100. PMID 19107149.
- N. Haliti; et al. (2009). „Hodnocení populační variace na 17 autosomálních STR a 16 Y-STR haplotypových lokusech u Chorvatů“. Forensic Science International: Genetics. 3 (4): 137–138. doi:10.1016 / j.fsigen.2008.11.004. PMID 19647700.
- D. Primorac; et al. (2011). „Chorvatské genetické dědictví: příběh chromozomu Y“. Chorvatský lékařský deník. 52 (3): 225–234. doi:10,3325 / cmj.2011.52.225. PMC 3118711. PMID 21674820.
- G. Mršić; et al. (2012). „Chorvatská národní referenční databáze haplotypů Y-STR“. Zprávy o molekulární biologii. 39 (7): 7727–41. doi:10.1007 / s11033-012-1610-3. PMID 22391654. S2CID 18011987.
- A. Zupan; et al. (2013). „Otcovská perspektiva slovinského obyvatelstva a jeho vztah k ostatním populacím“. Annals of Human Biology. 40 (6): 515–526. doi:10.3109/03014460.2013.813584. PMID 23879710. S2CID 34621779.
- A. Szécsényi-Nagy; et al. (2015). „Sledování genetického původu prvních evropských zemědělců odhaluje pohled na jejich sociální organizaci“. Sborník Královské společnosti B. 282 (1805): 20150339. doi:10.1098 / rspb.2015.0339. PMC 4389623. PMID 25808890.
- J. Šarac; et al. (2016). „Genetické dědictví Chorvatů v genofondu jihovýchodní Evropy - analýza chromozomu Y chorvatské kontinentální a ostrovní populace“. AJHB. 28 (6): 837–845. doi:10.1002 / ajhb.22876. PMID 27279290. S2CID 25873634.
- O.M. Utevska (2017). Генофонд українців за різними системами генетичних маркерів: походження і місце на європейському [Genofond Ukrajinců odhalený různými systémy genetických markerů: původ a prohlášení v Evropě] (PhD) (v ukrajinštině). Národní výzkumné středisko pro radiační medicínu Národní akademie věd Ukrajiny.
- I. Mathieson; et al. (2018). „Genomická historie jihovýchodní Evropy“. Příroda. 555 (7695): 197–203. doi:10.1038 / příroda25778. PMC 6091220. PMID 29466330.
- Šehović, Emir; et al. (2018). „Pohled na genetické vztahy v balkánských populacích využívajících síťovou analýzu založenou na haploskupinách Y-DNA přiřazených in silico“. Antropologický přehled. 81 (3): 252–268. doi:10.2478 / anre-2018-0021.
- mtDNA
- S. Cvjetan; et al. (2004). „Frekvence haploskupin mtDNA v jihovýchodní Evropě - Chorvaté, Bosňané a Hercegovinci, Srbové, Makedonci a makedonští Romové“. Collegium Antropologicum. 28 (1).
- M. Peričić; et al. (2005). "Přehled chorvatského genetického dědictví odhaleného mitochondriální DNA a chromozomálními liniemi Y". Chorvatský lékařský deník. 46 (4): 502–513. PMID 16100752.
- N. Jeran; et al. (2011). „Dědictví mitochondriální DNA Cres Islanders - příklad chorvatských genetických odlehlých hodnot“. Collegium Antropologicum. PMID 20102088.
- J. Ljubković; et al. (2011). „Charakteristiky raně středověkých obyvatel Naklice (jižní Chorvatsko) odhalené analýzou mitochondriální DNA“ (PDF). Kniha sborníku ze 7. konference ISABS ve forenzní, antropologické a lékařské genetice a přednášky na klinice Mayo v translační medicíně: 157.
- J. Šarac; et al. (2012). „Vliv evolučních sil a demografických procesů na genetickou strukturu tří chorvatských populací: mateřská perspektiva“. Annals of Human Biology. 39 (2): 143–155. doi:10.3109/03014460.2012.660194. PMID 22324841. S2CID 4833862.
- D. Havaš Auguštin; et al. (2012). „Mateřské genetické dědictví východního jadranského ostrova Krk - souhra evolučních sil a historických událostí ostrova při utváření genetické struktury současné ostrovní populace“. Collegium Antropologicum. 36 (2).
- J. Šarac; et al. (2014). „Mateřské genetické dědictví jihovýchodní Evropy odhaluje nový chorvatský izolát a nové místní podrozvětvení v haploskupině X2“. Annals of Human Genetics. 78 (3): 178–194. doi:10.1111 / ahg.12056. PMID 24621318. S2CID 206980447.
- D. Sutlović; et al. (2014). „Pozůstatky koster z pozdní doby římské: Staré jako Diokleciánův palác“. Collegium Antropologicum. 38 (2): 749–754. PMID 25145017.
- Ž. Základní; et al. (2015). „Kulturní rozdíly mezi populacemi neodrážejí biologické vzdálenosti: příklad interdisciplinární analýzy populací z východního pobřeží Jaderského moře“. Chorvatský lékařský deník. 56 (3): 230–238. doi:10,3325 / cmj.2015.56.230. PMC 4500963. PMID 26088847.
- D. Fernandes; et al. (2016). „Předběžné výsledky prehistorické lidské časové řady starých DNA z pobřežního a vnitrozemského Chorvatska“. IUAES. doi:10,13140 / RG.2.1.2555.9920. Citovat deník vyžaduje
| deník =(Pomoc) - G. Mršić; et al. (2018). „Rozšířená chorvatská databáze 12 lokusů X-STR s přehledem anomálních profilů“. Forensic Science International: Genetics. 34: 249–256. doi:10.1016 / j.fsigen.2018.03.004. PMID 29573605.
- L. Barbarić; et al. (2020). „Mateřská perspektiva chorvatské genetické rozmanitosti“. Forensic Science International: Genetics. 44: 102190. doi:10.1016 / j.fsigen.2019.102190. PMID 31756631.
- atDNA
- P. Ralph; et al. (2013). „Geografie nedávného genetického původu v celé Evropě“. PLOS Biology. 11 (5): e105090. doi:10.1371 / journal.pbio.1001555. PMC 3646727. PMID 23667324.
- L. Kovačević; et al. (2014). „Stojíme u brány do Evropy - genetická struktura populací západního Balkánu na základě autozomálních a haploidních markerů“. PLOS One. 9 (8): e105090. doi:10.1371 / journal.pone.0105090. PMC 4141785. PMID 25148043.
- A. Kushniarevich; et al. (2015). „Genetické dědictví balto-slovanských mluvících populací: syntéza autozomálních, mitochondriálních a Y-chromozomálních dat“. PLOS One. 10 (9): e0135820. doi:10.1371 / journal.pone.0135820. PMC 4558026. PMID 26332464.
- A. Jeroncic; et al. (2016). „Sekvenování celého exomu u izolované populace z dalmatského ostrova Vis“. European Journal of Human Genetics. 24 (10): 1479–1487. doi:10.1038 / ejhg.2016.23. PMC 4950961. PMID 27049301.
- P. M. Delser; et al. (2018). „Genetická krajina Slovinců: minulá směs a přirozený výběr“. Frontiers in Genetics. 9: 551. doi:10.3389 / fgene.2018.00551. PMC 6252347. PMID 30510563.
externí odkazy
- The Chorvatský projekt DNA na DNA rodokmenu
- The 10.001 Dalmatinů - chorvatská biobanka na University of Split Lékařská fakulta






