Varianty PCR - Variants of PCR
Všestrannost polymerázová řetězová reakce (PCR) vedla k velkému počtu varianty PCR.
Základní úpravy
K dosažení požadovaného cíle je často nutné provést pouze malou úpravu standardního protokolu PCR:
Multiplex-PCR používá několik párů primerů nasedajících na různé cílové sekvence. To umožňuje simultánní analýzu více cílů v jednom vzorku. Například při testování genetických mutací lze kombinovat šest nebo více amplifikací. Ve standardním protokolu pro Otisk DNA, testované cíle jsou často amplifikovány ve skupinách po 3 nebo 4. Zesílení sondy závislé na multiplexní ligaci (nebo MLPA ) umožňuje amplifikaci více cílů pouze s použitím jediného páru primerů, čímž se zabrání omezení rozlišení u multiplexní PCR. Multiplexní PCR byla také použita pro analýzu mikrosatelity a SNP.[1]

Variabilní počet tandemových opakování (VNTR) PCR cílí na oblasti genomu, které se projevují variace délky. Analýza genotypů vzorku obvykle zahrnuje dimenzování produktů amplifikace podle Gelová elektroforéza. Analýza menších segmentů VNTR známých jako krátké tandemové opakování (nebo STR) je základem pro DNA otisky prstů databáze jako CODIS.
Asymetrická PCR přednostně amplifikuje jedno vlákno cílové DNA. V některých se používá sekvenování metody a hybridizace sondování, ke generování jednoho řetězce DNA jako produktu. Termocyklování se provádí jako v PCR, ale s omezeným množstvím nebo vynecháním jednoho z primerů. Když se omezující primer vyčerpá, replikace se zvyšuje aritmeticky prodloužením přebytečného primeru.[2] Modifikace tohoto procesu, pojmenovaná Lv uchu-After-Ton-Exponential-PCR (nebo LATE-PCR), používá omezující primer s vyšším Teplota tání (T.m) než přebytek primeru k udržení účinnosti reakce, protože mezní koncentrace primeru snižuje střední reakci.[3] (Viz také PCR s prodloužením překrytí ).
K provedení jsou nutné některé úpravy dlouhá PCR. Originál Klenowský Proces PCR negeneroval produkty, které byly větší než asi 400 bp. Taq polymeráza může však zesílit cíle dlouhé až několik tisíc bp.[4] Od té doby umožnily modifikované protokoly s Taq enzymem amplifikovat cíle větší než 50 kb.[5]
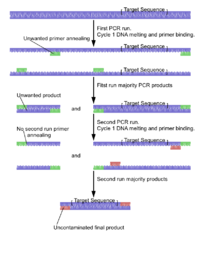
Vnořená PCR se používá ke zvýšení specificity amplifikace DNA. Dvě sady primerů se používají ve dvou po sobě jdoucích reakcích. V první PCR se jeden pár primerů používá ke generování produktů DNA, které mohou obsahovat produkty amplifikované z necílových oblastí. Produkty z první PCR se poté použijí jako templát ve druhé PCR, s použitím jednoho („polohnízdění“) nebo dvou různých primerů, jejichž vazebná místa jsou umístěna (vnořena) do první sady, čímž se zvyšuje specificita. Vnořená PCR je často úspěšnější ve specifické amplifikaci produktů dlouhé DNA než konvenční PCR, vyžaduje však podrobnější znalost sekvence cíle.
Kvantitativní PCR se používá k měření specifického množství cílové DNA (nebo RNA) ve vzorku. Měřením zesílení pouze ve fázi skutečného exponenciálního nárůstu množství měřeného produktu přesněji odráží počáteční množství cíle. Používají se speciální tepelné cyklovače, které sledují množství produktu během zesílení. Kvantitativní real-time PCR (QRT-PCR) metody používají fluorescenční barviva, jako je Sybr Green, nebo fluorofor -obsahující sondy DNA, jako např TaqMan, k měření množství zesíleného produktu, jak zesílení postupuje.
Hot-start PCR je technika prováděná ručně zahříváním reakčních složek na teplotu tání DNA (např. 95 ° C) před přidáním polymerázy. Tímto způsobem je zabráněno nespecifickému zesílení při nižších teplotách.[6] Alternativně mohou speciální činidla inhibovat aktivitu polymerázy při teplotě okolí, a to buď vazbou an protilátka, nebo přítomností kovalentně vázaných inhibitorů, které disociují pouze po kroku aktivace při vysoké teplotě. „Hot-start / cold-finish PCR“ se dosahuje pomocí nových hybridních polymeráz, které jsou neaktivní při teplotě okolí a jsou aktivovány pouze při zvýšených teplotách.
v touchdown PCR, teplota žíhání se v pozdějších cyklech postupně snižuje. Teplota žíhání v počátečních cyklech je obvykle 3–5 ° C nad standardní Tm použitých primerů, zatímco v pozdějších cyklech je to podobné množství pod Tm. Počáteční vyšší teplota žíhání vede k větší specificitě pro vazbu primeru, zatímco nižší teploty umožňují účinnější amplifikaci na konci reakce.[7]
Sestavení PCR (také známý jako Sestava pro cyklickou polymerázu nebo PCA) je syntéza dlouhých struktur DNA prováděním PCR na souboru dlouhých oligonukleotidů s krátkými překrývajícími se segmenty za účelem sestavení dvou nebo více kusů DNA do jednoho kusu. Zahrnuje počáteční PCR s primery, které se překrývají, a druhou PCR s použitím produktů jako templátu, který generuje konečný produkt plné délky. Tato technika může nahradit ligace - na základě montáže.[8]
v kolonie PCR, bakteriální kolonie jsou skrínovány přímo pomocí PCR, například skrínink na správnou DNA vektor konstrukty. Koloniím se odebírají vzorky pomocí sterilní špičky pipety a malé množství buněk se přenese do směsi PCR. K uvolnění DNA z buněk se PCR zahájí buď prodlouženou dobou při 95 ° C (pokud se použije standardní polymeráza), nebo zkráceným denaturačním krokem při 100 ° C a speciální chimérickou DNA polymerázou.[9]
The digitální polymerázová řetězová reakce současně zesiluje tisíce vzorků, každý zvlášť kapička v emulzi.
Sebevražedná PCR se obvykle používá v paleogenetika nebo jiné studie, kde je nejvyšší prioritou vyhnout se falešným pozitivům a zajistit specificitu amplifikovaného fragmentu. Původně to bylo popsáno ve studii, která měla ověřit přítomnost mikrobu Yersinia pestis ve vzorcích zubů získaných z hrobů lidí ze 14. století údajně zabitých morem během středověku Černá smrt epidemický.[10] Metoda předepisuje použití jakékoli kombinace primerů pouze jednou v PCR (odtud termín „sebevražda“), která by nikdy neměla být použita v žádné pozitivní kontrolní reakci PCR, a primery by měly vždy cílit na genomovou oblast, která v laboratoř používající tuto nebo jinou sadu primerů. Tím je zajištěno, že v laboratoři není přítomna žádná kontaminující DNA z předchozích PCR reakcí, které by jinak mohly generovat falešně pozitivní výsledky.
STUDENÉ PCR (co- zesílení na lower denaturation temperature-PCR) je upravený protokol, který obohacuje variantní alely ze směsi divokého typu a DNA obsahující mutaci.
Předběžné úpravy a rozšíření
Základní proces PCR může někdy předcházet nebo následovat jinou techniku.
RT-PCR (nebo Rodvrátit Tpřepis PCR) se používá k reverznímu přepisu a zesílení RNA na cDNA. PCR předchází reakce s použitím reverzní transkriptáza, enzym, který převádí RNA na cDNA. Tyto dvě reakce lze kombinovat ve zkumavce, přičemž k inaktivaci transkriptázy se použije počáteční zahřívací krok PCR.[4] T-polymeráza (popsaná níže) má RT aktivitu a může provádět celou reakci. RT-PCR je široce používán v profilování výrazů, který detekuje expresi genu. Může být také použit k získání sekvence transkriptu RNA, což může pomoci při stanovení začátek transkripce a terminační stránky (podle RACE-PCR ) a usnadnit mapování polohy exony a introny v genové sekvenci.
Oboustranná PCR používá jediný primer, který se váže na cíl microRNA s 3 'i 5' konci, známý jako hemiprobes.[11] Aby došlo k vazbě, musí být oba konce komplementární. 3'-konec je poté prodloužen reverzní transkriptázou za vzniku dlouhé cDNA. CDNA je poté amplifikována pomocí dvou cílových specifických primerů PCR. Kombinace dvou hemi sond, které obě cílí na krátký cíl mikroRNA, činí test se dvěma ocasy mimořádně citlivým a specifickým.
Ligací zprostředkovaná PCR používá malé oligonukleotidové „linkery“ (nebo adaptéry) DNA, které jsou nejprve ligovány k fragmentům cílové DNA. K amplifikaci cílových fragmentů se poté použijí PCR primery, které hybridizují k linkerovým sekvencím. Tato metoda je nasazena pro sekvenování DNA, chůzi genomu a Stopy DNA.[12] Příbuzná technika je zesílený polymorfismus délky fragmentu, který generuje diagnostické fragmenty genomu.
Methylačně specifická PCR (MSP) se používá k identifikaci vzorců Methylace DNA na ostrovy cytosin-guanin (CpG) v genomové DNA.[13] Cílová DNA je nejprve ošetřena hydrogensiřičitan sodný, který převádí nemetylovaný cytosin základny uracil, který je doplňkem k adenosin v primerech PCR. Poté se na DNA upravené hydrogensiřičitanem provedou dvě amplifikace: Jedna sada primerů hybridizuje s DNA s cytosiny (odpovídá methylovanému cytosinu) a druhá sada se hybridizuje s DNA s uracilem (odpovídá nemetylovanému cytosinu). MSP používané v kvantitativní PCR poskytuje kvantitativní informace o stavu methylace daného ostrova CpG.[14]
Další úpravy
Pro optimální výkon se běžně používají úpravy komponent v PCR.
Divalentní hořčík ion (Mg++) je vyžadován pro aktivitu polymerázy PCR. Nižší koncentrace Mg++ zvýší věrnost replikace, zatímco vyšší koncentrace zavedou více mutací.[15]
Denaturanty(jako DMSO ) může zvýšit specificitu amplifikace destabilizací nespecifické vazby primeru. Jiné chemikálie, jako např glycerol, jsou stabilizátory pro aktivitu polymerázy během amplifikace. Čistící prostředky (jako Triton X-100 ) může zabránit ulpívání polymerázy na sobě nebo na stěnách reakční zkumavky.
DNA polymerázy příležitostně začleňují do prodlužujícího se řetězce báze nesouladu. Vysoce věrná PCR zaměstnává enzymy s 3'-5 ' exonukleáza aktivita, která snižuje tuto míru chybného začlenění. Mezi příklady enzymů s korekturní aktivitou patří Pfu; úpravy Mg++ a koncentrace dNTP mohou pomoci maximalizovat počet produktů, které přesně odpovídají původní cílové DNA.[Citace je zapotřebí ]
Úpravy primeru
Úpravy syntetických oligonukleotidů použitých jako primery v PCR jsou bohatým zdrojem modifikace:
Normálně jsou PCR primery vybrány z neměnné části genomu a mohou být použity k amplifikaci polymorfní oblasti mezi nimi. v alela-specifická PCR opak je hotový. Alespoň jeden z primerů je vybrán z polymorfní oblasti s mutacemi umístěnými na (nebo blízko) jeho 3'-konce. Za přísných podmínek neodpovídající primer nebude inicializovat replikaci, zatímco odpovídající primer ano. Vzhled produktu amplifikace tedy naznačuje genotyp. (Další informace viz Genotypizace SNP.)
InterSekvenčně specifická PCR (nebo ISSR-PCR) je metoda pro DNA otisky prstů který používá primery vybrané ze segmentů opakovaných v celém genomu k vytvoření jedinečného otisku prstu se zesílenou délkou produktu.[16] Použití primerů z a běžně se opakující segment je nazýván Alu-PCR, a může pomoci amplifikovat sekvence sousedící (nebo mezi) s těmito repeticemi.
Primery mohou být také navrženy tak, aby byly „zdegenerované“ - schopné inicializovat replikaci z velkého počtu cílových míst. Zesílení celého genomu (nebo WGA) je skupina postupů, které umožňují amplifikaci na mnoha místech neznámého genomu a které mohou být k dispozici pouze v malém množství. Používají se jiné techniky zdegenerované primery které jsou syntetizovány pomocí více nukleotidů v určitých polohách (polymeráza „vybírá“ správně přiřazené primery). Primery lze také syntetizovat s nukleosidový analog inosin, který hybridizuje se třemi ze čtyř normálních bází. Podobná technika může vynutit provedení PCR Místně zaměřená mutageneze. (viz také Překryvná extenze polymerázové řetězové reakce )
Normálně jsou primery použité v PCR navrženy tak, aby byly plně komplementární k cíli. Polymeráza je však tolerantní k nesprávnému shoda od 3 'konce. Ocasní primery zahrnují nekomplementární sekvence na svých 5 'koncích. Běžným postupem je použití linkerové primery, které nakonec umístí restrikční místa na konce produktů PCR, což usnadní jejich pozdější inzerci do klonovacích vektorů.
Rozšířením metody „kolonie-PCR“ (výše) je použití vektorové primery. Cílové fragmenty DNA (nebo cDNA) se nejprve vloží do klonování vektor, a jedna sada primerů je navržena pro oblasti vektoru ohraničující místo inzerce. K amplifikaci dochází u jakékoli vložené DNA.[4]
PCR lze snadno upravit za vzniku a označený produkt pro následné použití jako a hybridizace sonda. Jeden nebo oba primery mohou být použity v PCR s již připojenou radioaktivní nebo fluorescenční značkou, nebo mohou být značky přidány po amplifikaci. Tyto metody značení lze kombinovat s „asymetrickou PCR“ (viz výše) za vzniku účinných hybridizačních sond.
PCR závislá na RNáze H (rhPCR) může snížit tvorbu primeru-dimeru a zvýšit počet testů v multiplexní PCR. Metoda využívá primery se štěpitelným blokem na 3 'konci, který je odstraněn působením termostabilního enzymu RNázy HII.[17]
DNA polymerázy
Existuje několik DNA polymeráz, které se používají při PCR.
The Klenowův fragment, odvozeno od originálu DNA polymeráza I z E-coli, byl prvním enzymem použitým v PCR. Kvůli nedostatečné stabilitě při vysoké teplotě je nutné jej doplňovat během každého cyklu, a proto se v PCR běžně nepoužívá.
Bakteriofág T4 DNA polymeráza (rodina A) byl také původně použit v PCR. Má vyšší věrnost replikace než Klenowův fragment, ale je také zničen teplem. T7 DNA polymeráza (rodina B) má podobné vlastnosti a účely. Bylo aplikováno na místně zaměřená mutageneze[18] a Sangerovo sekvenování.[19]
Taq polymeráza, DNA polymeráza I z Thermus aquaticus, byla první termostabilní polymerázou používanou v PCR a stále je nejběžněji používanou. Enzym lze izolovat z nativního zdroje nebo z jeho klonovaného genu exprimovaného v E-coli.[4] 61kDa zkrácená z důvodu chybějící aktivity 5'-3 'exonukleázy je známá jako Stoffelův fragment, a je vyjádřena v E-coli.[20] Nedostatek exonukleázové aktivity mu může umožnit amplifikovat delší cíle než nativní enzym. Bylo komercializováno jako AmpliTaq a Klentaq.[21] Byla také vyrobena varianta určená pro PCR s horkým startem, která se nazývá „Faststart polymerase“. Vyžaduje silnou tepelnou aktivaci, čímž se zabrání nespecifické amplifikaci v důsledku aktivity polymerázy při nízké teplotě. Mnoho dalších variant byly vytvořeny.[22]
jiný Thermus polymerázy, jako např Tth polymeráza I (P52028) z Thermus thermophilus, viděl nějaké použití. Tth má aktivitu reverzní transkriptázy v přítomnosti Mn2+ ionty, což umožňuje PCR amplifikaci z RNA cílů.[23]
The archean rod Pyrococcus se osvědčil jako bohatý zdroj termostabilních polymeráz s korekturou. Pfu DNA polymeráza, izolovaný od P. furiosus ukazuje pětinásobné snížení chybovosti replikace ve srovnání s Taq.[24] Vzhledem k tomu, že se s postupem PCR zvyšuje chyba, je Pfu preferovanou polymerázou, pokud mají být produkty jednotlivě klonovány pro sekvenování nebo expresi. Mezi další méně používané polymerázy tohoto rodu patří Pwo (P61876) z Pyrococcus woesei, Pfx od nejmenovaného druhu polymeráza „Deep Vent“ (Q51334) z kmene GB-D.[25]
Odvzdušněte nebo Tli polymeráza je extrémně termostabilní DNA polymeráza izolovaná z Thermococcus litoralis. Polymeráza z Thermococcus fumicolans (Tfu) byl také uveden na trh.[25]
Úpravy mechanismu
Někdy lze upravit i základní mechanismus PCR.

Na rozdíl od normální PCR Inverzní PCR umožňuje amplifikaci a sekvenování DNA, která obklopuje známou sekvenci. Zahrnuje to nejprve podrobení cílové DNA řadě restrikční enzym trávení a poté cirkulují výsledné fragmenty pomocí vlastní ligace. Primery jsou navrženy tak, aby byly prodlouženy vnější ze známého segmentu, což má za následek zesílení zbytku kruhu. To je zvláště užitečné při identifikaci sekvencí na obou stranách různých genomových inzertů.[26]
Podobně, termální asymetrická prokládaná PCR (nebo TAIL-PCR) se používá k izolaci neznámých sekvencí lemujících známou oblast genomu. Ve známé sekvenci používá TAIL-PCR vnořený pár primerů s různými teplotami žíhání. Pro degeneraci z neznámé sekvence v opačném směru se používá „degenerovaný“ primer.[27]
Izotermické metody amplifikace
Byly vyvinuty některé protokoly amplifikace DNA, které lze použít alternativně k PCR. Jsou izotermické, což znamená, že jsou provozovány při konstantní teplotě.[28]
Zesílení závislé na helikáze (HDA) je podobný tradiční PCR, ale používá spíše konstantní teplotu než cyklování denaturací a žíháním / prodlužováním. DNA helikáza, což je enzym, který odvíjí DNA, se používá místo tepelné denaturace.[29] Smyčkově zprostředkovaná izotermická amplifikace je podobný nápad, ale provádí se pomocí polymerázy vytěsňující vlákno.[30]
Reakce amplifikace enzymu Nicking (NEAR) a jeho bratranec zesílení posunutí vlákna (SDA) jsou izotermické, replikující DNA při konstantní teplotě pomocí polymerázy a enzymu nicking.[28]
Amplifikace rekombinázové polymerázy (RPA)[31] používá a rekombináza specificky párovat primery s dvouvláknovou DNA na základě homologie, a tak řídit syntézu DNA z definovaných sekvencí DNA přítomných ve vzorku. Přítomnost cílové sekvence iniciuje amplifikaci DNA a není nutné žádné tepelné ani chemické tavení DNA. Reakce postupuje rychle a má za následek specifickou amplifikaci DNA od několika cílových kopií až po detekovatelné hladiny obvykle během 5–10 minut. Celý reakční systém je stabilní jako sušená formulace a nevyžaduje chlazení. RPA lze použít k nahrazení PCR v různých laboratorních aplikacích a uživatelé si mohou navrhnout vlastní testy.[32]
Mezi další typy izotermického zesílení patří amplifikace celého genomu (WGA), Amplifikace na základě sekvence nukleové kyseliny (NASBA) a transkripcí zprostředkovaná amplifikace (TMA).[28]
Viz také
Reference
- ^ Hayden MJ, Nguyen TM, Waterman A, Chalmers KJ (2008). „Multiplex-Ready PCR: nová metoda pro multiplexované genotypování SSR a SNP“. BMC Genomics. 9: 80. doi:10.1186/1471-2164-9-80. PMC 2275739. PMID 18282271.
- ^ Innis MA, Myambo KB, Gelfand DH, Brow MA (prosinec 1988). „Sekvenování DNA pomocí DNA polymerázy Thermus aquaticus a přímé sekvenování DNA amplifikované polymerázovou řetězovou reakcí“. Proc. Natl. Acad. Sci. USA. 85 (24): 9436–40. Bibcode:1988PNAS ... 85.9436I. doi:10.1073 / pnas.85.24.9436. PMC 282767. PMID 3200828.
- ^ Pierce KE, Wangh LJ (2007). Lineární následná exponenciální polymerázová řetězová reakce a příbuzné technologie Strategie detekce v reálném čase pro rychlou a spolehlivou diagnostiku z jednotlivých buněk. Methods Mol Med. Metody v molekulární medicíně ™. 132. str. 65–85. doi:10.1007/978-1-59745-298-4_7. ISBN 978-1-58829-578-1. PMID 17876077.
- ^ A b C d Saiki RK, Gelfand DH, Stoffel S a kol. (Leden 1988). "Primerem řízená enzymatická amplifikace DNA termostabilní DNA polymerázou". Věda. 239 (4839): 487–91. Bibcode:1988Sci ... 239..487S. doi:10.1126 / science.239.4839.487. PMID 2448875.
- ^ Cheng S, Fockler C, Barnes WM, Higuchi R (červen 1994). „Efektivní amplifikace dlouhých cílů z klonovaných inzertů a lidské genomové DNA“. Proc. Natl. Acad. Sci. USA. 91 (12): 5695–9. Bibcode:1994PNAS ... 91.5695C. doi:10.1073 / pnas.91.12.5695. PMC 44063. PMID 8202550.
- ^ Chou Q, Russell M, Birch DE, Raymond J, Bloch W (duben 1992). „Prevence chybného primingu před a PCR a dimerizace primeru zlepšuje amplifikace s nízkým počtem kopií“. Nucleic Acids Res. 20 (7): 1717–23. doi:10.1093 / nar / 20.7.1717. PMC 312262. PMID 1579465.
- ^ Don RH, Cox PT, Wainwright BJ, Baker K, Mattick JS (červenec 1991). "'Touchdown 'PCR k obejití falešného primingu během genové amplifikace ". Nucleic Acids Res. 19 (14): 4008. doi:10.1093 / nar / 19.14.4008. PMC 328507. PMID 1861999.
- ^ Stemmer WP, Crameri A, Ha KD, Brennan TM, Heyneker HL (1995). "Jednostupňové sestavení genu a celého plazmidu z velkého počtu oligodeoxyribonukleotidů". Gen. 164 (1): 49–53. doi:10.1016/0378-1119(95)00511-4. PMID 7590320.
- ^ Pavlov AR, Pavlova NV, Kozyavkin SA, Slesarev AI (2006). „Termostabilní DNA polymerázy pro široké spektrum aplikací: Srovnání robustního hybridního TopoTaq s jinými enzymy“. In Kieleczawa J. (ed.). DNA Sequencing II: Optimizing Preparation and Cleanup. Jones a Bartlett. str. 241–257. ISBN 978-0-7637-3383-4.
- ^ Raoult, D; G Aboudharam; E Crubezy; G Larrouy; B Ludes; M Drancourt (07.11.2000). „Molekulární identifikace Yersinia pestis jako původce středověké černé smrti„ sebevražednou PCR “. Proc. Natl. Acad. Sci. USA. 97 (23): 12800–12803. Bibcode:2000PNAS ... 9712800R. doi:10.1073 / pnas.220225197. ISSN 0027-8424. PMC 18844. PMID 11058154.
- ^ Androvic, Peter; Valihrach, Lukas; Elling, Julie; Sjoback, Robert; Kubista, Mikael (2017). „Oboustranný RT-qPCR: nová metoda pro vysoce přesnou kvantifikaci miRNA“. Výzkum nukleových kyselin. 45 (15): e144. doi:10.1093 / nar / gkx588. ISSN 0305-1048. PMC 5587787. PMID 28911110.
- ^ Mueller PR, Wold B (listopad 1989). "In vivo stopa svalového specifického zesilovače ligací zprostředkovanou PCR". Věda. 246 (4931): 780–6. Bibcode:1989Sci ... 246..780M. doi:10.1126 / science.2814500. PMID 2814500.
- ^ Herman JG, Graff JR, Myöhänen S, Nelkin BD, Baylin SB (Září 1996). „Methylation-specific PCR: a new PCR assay for methylation status of CpG Islands“. Proc. Natl. Acad. Sci. USA. 93 (18): 9821–6. Bibcode:1996PNAS ... 93.9821H. doi:10.1073 / pnas.93.18.9821. PMC 38513. PMID 8790415.
- ^ Hernández, H; Tse, MY; Pang, SC; Arboleda, H; Forero, DA (říjen 2013). „Optimalizace metodik pro metylační analýzu DNA založenou na PCR“. Biotechniky. 55 (4): 181–197. doi:10.2144/000114087. PMID 24107250.
- ^ Markoulatos, P; Siafakas, N; Moncany, M (2002). „Multiplexní polymerázová řetězová reakce: praktický přístup“. Deník klinické laboratorní analýzy. 16 (1): 47–51. doi:10.1002 / jcla.2058. PMC 6808141. PMID 11835531.
- ^ E. Zietkiewicz; A. Rafalski a D. Labuda (1994). "Otisk genomu pomocí amplifikace polymerázové řetězové reakce s jednoduchým sekvenčním opakováním (SSR)". Genomika. 20 (2): 176–83. doi:10.1006 / geno.1994.1151. PMID 8020964.
- ^ Dobosy JR, Rose SD, Beltz KR, Rupp SM, Powers KM, Behlke MA, Walder JA (srpen 2011). „RNáza H-dependentní PCR (rhPCR): zlepšená specificita a detekce polymorfismu jednoho nukleotidu pomocí blokovaných štěpitelných primerů“. BMC biotechnologie. 11: 80. doi:10.1186/1472-6750-11-80. PMC 3224242. PMID 21831278.
- ^ Venkitaraman AR (duben 1989). „Použití modifikované T7 DNA polymerázy (sekvenáza verze 2.0) pro místně řízenou oligonukleotidovou mutagenezi“. Výzkum nukleových kyselin. 17 (8): 3314. doi:10.1093 / nar / 17.8.3314. PMC 317753. PMID 2726477.
- ^ „Thermo Sequenase DNA Polymerase“.
- ^ Lawyer, F. C .; Stoffel, S .; Saiki, R. K.; Chang, S. Y .; Landre, P. A .; Abramson, R. D .; Gelfand, D. H. (01.05.1993). „Exprese na vysoké úrovni, čištění a enzymatická charakterizace DNA polymerázy Thermus aquaticus plné délky a zkrácené formy s nedostatkem aktivity 5 'až 3' exonukleázy". Metody a aplikace PCR. 2 (4): 275–287. doi:10,1101 / gr. 2.4.275. ISSN 1054-9803. PMID 8324500.
- ^ „Applied Biosystems - Support“. www6.appliedbiosystems.com.
- ^ Villbrandt, B; Sobek, H; Frey, B; Schomburg, D (září 2000). „Výměna domén: chiméry DNA polymerázy Thermus aquaticus, DNA polymerázy I z Escherichia coli a DNA polymerázy Thermotoga neapolitana“. Proteinové inženýrství. 13 (9): 645–54. doi:10.1093 / protein / 13.9.645. PMID 11054459.
- ^ https://www.promega.in/products/pcr/rt-pcr/tth-dna-polymerase/
- ^ Cline J, Braman JC, Hogrefe HH (září 1996). „PCR věrnost pfu DNA polymerázy a dalších termostabilních DNA polymeráz“. Nucleic Acids Res. 24 (18): 3546–51. doi:10.1093 / nar / 24.18.3546. PMC 146123. PMID 8836181.
- ^ A b van Pelt-Verkuil E, van Belkum A, Hays JP (2008). "Taq a další termostabilní DNA polymerázy". Principy a technické aspekty amplifikace PCR. 103–18. doi:10.1007/978-1-4020-6241-4_7. ISBN 978-1-4020-6240-7.
- ^ Ochman H, Gerber AS, Hartl DL (1. listopadu 1988). „Genetické aplikace inverzní polymerázové řetězové reakce“. Genetika. 120 (3): 621–3. PMC 1203539. PMID 2852134.
- ^ Liu YG, Whittier RF (únor 1995). "Termální asymetrická prokládaná PCR: automatická amplifikace a sekvenování fragmentů inzertního konce z klonů P1 a YAC pro chůzi chromozomu". Genomika. 25 (3): 674–81. doi:10.1016 / 0888-7543 (95) 80010-J. PMID 7759102.
- ^ A b C „Izotermická amplifikace - přehled aplikace“. New England BioLabs, Inc. 2020. Citováno 13. srpna 2020.
- ^ Vincent M, Xu Y, Kong H (srpen 2004). „Zesílení izotermické DNA závislé na helikáze“. EMBO Rep. 5 (8): 795–800. doi:10.1038 / sj.embor.7400200. PMC 1249482. PMID 15247927.
- ^ Notomi T, Okayama H, Masubuchi H, Yonekawa T, Watanabe K, Amino N, Hase T (2000). „Smyčkově zprostředkovaná izotermická amplifikace DNA“. Nucleic Acids Res. 28 (12): 63e – 63. doi:10.1093 / nar / 28.12.e63. PMC 102748. PMID 10871386.
- ^ Piepenburg O, Williams CH, Stemple DL, Armes NA (2006). „Detekce DNA pomocí rekombinačních proteinů“. PLOS Biol. 4 (7): e204. doi:10.1371 / journal.pbio.0040204. PMC 1475771. PMID 16756388.
- ^ Lutz S, Weber P, Focke M, Faltin B, Hoffmann J, Müller C, Mark D, Roth G, Munday P, Armes N, Piepenburg O, Zengerle R, von Stetten F (duben 2010). „Mikrofluidní laboratoř na folii pro analýzu nukleových kyselin na základě izotermické rekombinázové polymerázové amplifikace (RPA)“. Laboratorní čip. 10 (7): 887–93. doi:10.1039 / b921140c. PMID 20300675.
