Digitální polymerázová řetězová reakce - Digital polymerase chain reaction
Zdá se, že hlavní přispěvatel do tohoto článku má úzké spojení s jeho předmětem. (Srpna 2019) (Zjistěte, jak a kdy odstranit tuto zprávu šablony) |
Digitální polymerázová řetězová reakce (digitální PCR, DigitalPCR, dPCRnebo dePCR) je biotechnologické zdokonalení konvenčních polymerázová řetězová reakce metody, které lze použít k přímé kvantifikaci a klonální amplifikaci řetězců nukleových kyselin, včetně DNA, cDNA nebo RNA. Klíčový rozdíl mezi dPCR a tradiční PCR spočívá v metodě měření množství nukleových kyselin, přičemž první z nich je přesnější metodou než PCR, ačkoli je také náchylnější k chybám v rukou nezkušených uživatelů.[1] „Digitální“ měření kvantitativně a diskrétně měří určitou proměnnou, zatímco „analogové“ měření extrapoluje určitá měření na základě měřených vzorců. PCR provádí jednu reakci na jeden vzorek. dPCR také provádí jedinou reakci ve vzorku, avšak vzorek je rozdělen na velké množství oddílů a reakce se provádí v každém oddílu samostatně. Tato separace umožňuje spolehlivější sběr a citlivější měření množství nukleových kyselin. Metoda byla prokázána jako užitečná pro studium variací genových sekvencí - jako jsou varianty počtu kopií a bodové mutace - a běžně se používá pro klonální amplifikaci vzorků pro sekvenování nové generace.
Zásady

Ke kvantifikaci se používá metoda polymerázové řetězové reakce nukleové kyseliny amplifikací molekuly nukleové kyseliny s enzymem DNA polymeráza.[2] Konvenční PCR je založeno na teorii, že zesílení je exponenciální. Nukleové kyseliny proto mohou být kvantifikovány porovnáním počtu amplifikačních cyklů a množství koncového produktu PCR s množstvím referenčního vzorku. Mnoho faktorů však tento výpočet komplikuje a vytváří nejistoty a nepřesnosti. Mezi tyto faktory patří následující: počáteční amplifikační cykly nemusí být exponenciální; Amplifikace PCR případně náhorní plošiny po nejistém počtu cyklů; a nízké počáteční koncentrace molekul cílové nukleové kyseliny se nemusí amplifikovat na detekovatelné úrovně. Nejvýznamnějším omezením PCR je však to, že účinnost amplifikace PCR ve sledovaném vzorku se může lišit od účinnosti referenčních vzorků. Protože PCR je exponenciální proces, lze pozorovat pouze dvojnásobné rozdíly v amplifikaci, což výrazně ovlivňuje platnost a přesnost výsledků.

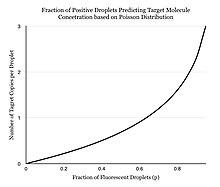
Místo provedení jedné reakce na jamku zahrnuje dPCR rozdělení PCR roztoku na desítky tisíc kapiček o velikosti nano litrů, kde v každé z nich probíhá samostatná PCR reakce.[3][4] Roztok PCR je vyroben podobně jako a TaqMan test, který se skládá z templátové DNA (nebo RNA), fluorescenčních zhášecích sond, primerů a PCR hlavní mix, který obsahuje DNA polymeráza, dNTP, MgCl2a reakční pufry v optimálních koncentracích. K dělení vzorků lze použít několik různých metod, včetně mikrojamkových destiček, kapilár, olejové emulze a polí miniaturizovaných komor s povrchy vázajícími nukleové kyseliny.[5] Roztok PCR je rozdělen na menší reakce a poté je proveden jednotlivě PCR. Po několika cyklech amplifikace PCR se vzorky kontrolují na fluorescenci s binárním odečtem „0“ nebo „1“. Zaznamená se zlomek fluoreskujících kapiček.[4] Rozdělení vzorku umožňuje odhadnout počet různých molekul za předpokladu, že populace molekul následuje Poissonovo rozdělení, což odpovídá možnosti více cílových molekul obývajících jednu kapičku. Použitím Poissonova zákona malých čísel lze distribuci cílové molekuly ve vzorku přesně aproximovat, což umožňuje kvantifikaci cílového řetězce v produktu PCR.[6] Tento model jednoduše předpovídá, že jak se zvyšuje počet vzorků obsahujících alespoň jednu cílovou molekulu, zvyšuje se pravděpodobnost vzorků obsahujících více než jednu cílovou molekulu. V konvenční PCR je počet cyklů amplifikace PCR úměrný počátečnímu počtu kopií. Na rozdíl od víry mnoha lidí, že dPCR poskytuje absolutní kvantifikaci, používá digitální PCR statistickou sílu k zajištění relativní kvantifikace. Například pokud vzorek A, pokud je testován na 1 milionu oddílů, dává jednu pozitivní reakci, neznamená to, že vzorek A má jednu výchozí molekulu.
Mezi výhody dPCR patří zvýšená přesnost díky masivnímu dělení vzorků, které zajišťuje spolehlivé měření v požadované sekvenci DNA díky reprodukovatelnosti.[4] Míra chyb je větší při detekci rozdílů malé změny u základní PCR, zatímco míra chyb je menší u dPCR kvůli rozdílům malé změny, které lze detekovat v sekvenci DNA. Samotná technika snižuje použití většího množství potřebného činidla, což nevyhnutelně sníží náklady na experiment. Také dPCR je vysoce kvantitativní, protože se při stanovení množství amplifikované cílové DNA nespoléhá na relativní fluorescenci roztoku.
Srovnání mezi dPCR a real-time PCR (qPCR)
dPCR měří skutečný počet molekul (cílová DNA), protože každá molekula je v jedné kapičce, což z ní činí diskrétní „digitální“ měření. Poskytuje absolutní kvantifikaci, protože dPCR měří pozitivní zlomek vzorků, což je počet kapiček, které fluoreskují v důsledku správné amplifikace. Tato pozitivní frakce přesně označuje počáteční množství templátové nukleové kyseliny. Podobně, qPCR využívá fluorescence; měří však intenzitu fluorescence ve specifických časech (obvykle po každém amplifikačním cyklu), aby se určilo relativní množství cílové molekuly (DNA), ale nemůže určit přesné množství bez konstrukce standardní křivky s použitím různých množství definovaného standardu. Udává prahovou hodnotu na cyklus (CT) a rozdíl v CT se používá k výpočtu množství počáteční nukleové kyseliny. Jako takový je qPCR analogovým měřením, které nemusí být tak přesné kvůli extrapolaci potřebné k dosažení měření.[5][7]
dPCR měří množství DNA po dokončení amplifikace a poté určuje podíl replikátů. Toto je reprezentativní pro měření koncového bodu, protože vyžaduje pozorování dat po dokončení experimentu. Naproti tomu qPCR zaznamenává relativní fluorescenci DNA ve specifických bodech během procesu amplifikace, což vyžaduje zastavení experimentálního procesu. Tento aspekt qPCR „v reálném čase“ může teoreticky ovlivnit výsledky v důsledku zastavení experimentu.[Citace je zapotřebí ] V praxi však většina qPCR termální cyklisty přečtěte si fluorescenci každého vzorku velmi rychle na konci kroku žíhání / prodloužení, než přejdete k dalšímu kroku tavení, což znamená, že tato hypotetická obava není ve skutečnosti relevantní ani použitelná pro drtivou většinu výzkumníků.
qPCR není schopen rozlišit rozdíly v genové expresi nebo kopírovat variace počtu čísel, které jsou menší než dvojnásobné. Je obtížné identifikovat alely s frekvencemi menšími než 1%, protože vysoce hojné běžné alely by se shodovaly s podobnými sekvencemi.[je zapotřebí objasnění ] Na druhou stranu se ukázalo, že dPCR detekuje rozdíly v genové expresi menší než 30%, rozlišuje mezi variacemi počtu kopií, které se liší pouze o 1 kopii, a identifikuje alely, které se vyskytují při frekvencích menších než 0,1%.[8]
Aplikace
Digitální PCR má mnoho aplikací základní výzkum, klinická diagnostika a testování prostředí. Jeho použití zahrnuje patogen detekce a zdraví trávicího ústrojí analýza;[9][10] kapalná biopsie pro rakovina monitorování, varhany odmítnutí transplantátu monitorovací a neinvazivní prenatální testování vážně genetické abnormality;[11][12][13][14][15][16][17][18] variace počtu kopií analýza,[19][20][21] analýza exprese jednoho genu,[22] detekce vzácných sekvencí,[18][23][24] profilování genové exprese a jednobuněčná analýza;[25][26][24][27][28][29][30] detekce DNA kontaminující látky v biologickém zpracování,[31] validace genové úpravy a detekce konkrétních změny methylace v DNA tak jako biomarkery rakoviny.[32][33][34][35] dPCR se také často používá jako ortogonální metoda k potvrzení vzácných mutací detekovaných prostřednictvím sekvenování nové generace (NGS) a ověřit NGS knihovny.[36][37][38]
Absolutní kvantifikace
dPCR umožňuje absolutní a reprodukovatelnou kvantifikaci cílových nukleových kyselin při rozlišení jedné molekuly.[24][39][40][41] Na rozdíl od analogového kvantitativní PCR (qPCR) však absolutní kvantifikace pomocí dPCR nevyžaduje a standardní křivka ).[39] dPCR má také větší toleranci vůči inhibitorovým látkám a PCR testům, které se neefektivně amplifikují ve srovnání s qPCR.[42][43]
dPCR může kvantifikovat například přítomnost specifických sekvencí z kontaminace geneticky modifikované organismy v potravinách,[44] virová zátěž v krvi,[45] PBMC,[46][47] vzorky séra,[48] tkáně choriových klků,[49][50] biomarkery neurodegenerativního onemocnění v mozkové míchě,[51] a fekální kontaminace v pitné vodě. [52]
Zkopírujte variaci čísla
Změna stavu počtu kopií s ohledem na referenční lokus pro jednu kopii se označuje jako „variace počtu kopií „(CNV), pokud se objeví v zárodečných buňkách, nebo změna počtu kopií (CNA), pokud se objeví v somatických buňkách.[53] CNV nebo CNA mohou být způsobeny delecí nebo amplifikací lokusu s ohledem na počet kopií referenčního lokusu přítomného v buňce a společně jsou hlavními přispěvateli k variabilitě v lidský genom.[54][55][56] Byly spojovány s rakovinou;[57][58][59] neurologický,[60] psychiatrické,[61][62] a autoimunitní onemocnění;[63] a nežádoucí účinky léků.[64] Je však obtížné měřit tyto alelické variace s vysokou přesností za použití jiných metod, jako je qPCR, což ztěžuje fenotypové a chorobné asociace se změněným stavem CNV.[65][66]
Velký počet „digitalizovaných“ měření koncových bodů umožněných rozdělením vzorků umožňuje dPCR řešit malé rozdíly v počtu kopií s lepšími přesnost a preciznost ve srovnání s jinými metodami, jako jsou mikropole založené na SNP[67] nebo qPCR.[68][69] qPCR má omezenou schopnost přesně kvantifikovat genové amplifikace u několika nemocí, včetně Crohnovy choroby, infekce HIV-1 a obezity.[70][66][69]
dPCR byl navržen k měření koncentrace cíle nukleové kyseliny v kopiích na jednotku objemu vzorku. Pokud pracujete ve zředěných reakcích, kde méně než ~ 10% oddílů obsahuje požadovaný cíl (označovaný jako „limitní ředění“), lze počet kopií odhadnout porovnáním počtu fluorescenčních kapiček vznikajících z cílové CNV s počtem fluorescenčních kapičky vznikající z neměnného referenčního lokusu s jednou kopií.[19] Ve skutečnosti, jak při těchto nižších cílových koncentracích, tak při vyšších koncentracích, kde lze více kopií stejného cíle lokalizovat do jednoho oddílu, Poissonova statistika se používají pro korekci těchto vícenásobných povolání, aby byla zajištěna přesnější hodnota koncentrace každého cíle.[71][72]
K odhalení zárodečné i somatické variace v počtu kopií genů mezi lidmi byla použita digitální PCR[73] a studovat souvislost mezi zesílením HER2 (ERBB2) a rakovina prsu postup.[74][75][76][21]
Vzácná mutace a detekce vzácných alel
Rozdělení do oddílů v digitální PCR zvyšuje citlivost a umožňuje detekci zvláště vzácných událostí jednotlivé nukleotidové varianty (SNV), izolováním nebo výrazným zmenšením cíle biomarker signál z potenciálně konkurenčního pozadí.[7][5] Tyto události lze rozdělit do dvou tříd: detekce vzácných mutací a detekce vzácných sekvencí.
Detekce vzácných mutací
K detekci vzácných mutací dochází, když biomarker existuje na pozadí vysoce hojného protějšku, který se liší pouze jedinou nukleotidovou variantou (SNV). Ukázalo se, že digitální PCR je schopna detekovat mutantní DNA v přítomnosti 200 000násobného přebytku DNA divoký typ pozadí, které je 2 000krát citlivější než dosažitelné s konvenčním qPCR.[7]
Detekce vzácných sekvencí
Digitální PCR dokáže detekovat vzácné sekvence, jako je HIV DNA u pacientů s HIV,[18] a DNA z fekálních bakterií v oceánu a dalších vzorcích vody pro hodnocení kvality vody.[77] dPCR dokáže detekovat sekvence tak vzácné jako 1 z každých 1 250 000 buněk.[18]
Tekutá biopsie
Schopnost dPCR detekovat vzácné mutace může být na klinice zvláště výhodná díky použití kapalná biopsie, obecně neinvazivní strategie pro detekci a monitorování onemocnění pomocí tělesných tekutin.[11][78] Vědci použili tekutou biopsii ke sledování nádorové zátěže, odezvy na léčbu a progrese onemocnění v rakovina pacientů měřením vzácných mutací v cirkulující nádorová DNA (ctDNA) v různých biologických tekutinách od pacientů včetně krev, moč a mozkomíšní mok.[11][79][80] Včasná detekce ctDNA (jako u molekulárních relaps ) může vést k dřívější administraci imunoterapie nebo cílenou terapii specifickou pro pacientův podpis mutace, která potenciálně zvyšuje šance na účinnost léčby, spíše než čekat na klinický relaps před změnou léčby. Kapalné biopsie mohou mít dobu zpracování několik dní, ve srovnání s dvěma až čtyřmi týdny nebo déle pro tkáňové testy.[81][82] Tuto zkrácenou dobu potřebnou k dosažení výsledků využili lékaři k urychlení léčby šité na míru biopsie data.[81]
V roce 2016 ověřila prospektivní studie používající dPCR na Dana-Farber Cancer Institute klinický přínos tekuté biopsie jako prediktivního diagnostického nástroje pro pacienty s nemalobuněčný karcinom plic.[83] Aplikace tekutých bioptických testů byla také studována u pacientů s prsa,[84] kolorektální,[85][86] gynekologické,[87] a měchýř rakoviny[79][88] sledovat zátěž nemoci a reakci nádoru na léčbu.
Genová exprese a kvantifikace RNA
Genový výraz a RNA kvantifikační studie těží ze zvýšené přesnosti a absolutní kvantifikace dPCR. Kvantifikace RNA může být provedena pomocí RT-PCR, kde RNA je reverzně transkribována cDNA v samotné dělené reakci a počet molekul RNA pocházejících z každého transkriptu (nebo alelického transkriptu) je kvantifikován pomocí dPCR (ref).[25]
Větší citlivost a přesnost lze často dosáhnout použitím dPCR namísto qPCR k částečné kvantifikaci molekul RNA, protože pro kvantifikaci nevyžaduje použití standardní křivky.[89] dPCR je také odolnější vůči inhibitorům PCR pro kvantifikaci RNA než qPCR.[42][10]
dPCR může detekovat a kvantifikovat více jednotlivých cílových druhů na detekční kanál než qPCR na základě schopnosti rozlišit cíle na základě jejich diferenciální amplitudy fluorescence nebo použitím odlišných barevných kombinací pro jejich detekci.[90] Jako příklad toho lze použít dvoukanálový dPCR systém k detekci exprese čtyř různých sestřihových variant lidské v jedné jamce reverzní transkriptáza telomerázy, protein, který je aktivnější ve většině nádorových buněk než ve zdravých buňkách.[91]
Alternativní použití pro oddíly
Pomocí schopností dynamického dělení použitých v dPCR lze dosáhnout zlepšeného sekvenování NGS rozdělením komplexních reakcí PCR před amplifikací, aby se dosáhlo rovnoměrnější amplifikace v mnoha odlišných amplikony pro NGS analýza.[92][93] Navíc se ukázalo, že vylepšená specificita komplexních reakcí amplifikace PCR v kapičkách výrazně snižuje počet iterací potřebných k výběru vysoké afinity. aptamery v SELEX metoda.[94] Rozdělení může také umožnit robustnější měření aktivity telomerázy z buněčných lyzátů.[95][96] Možnosti dynamického dělení dPCR lze také použít k rozdělení tisíců jader nebo celých buněk na jednotlivé kapičky, aby se usnadnila příprava knihovny pro jednu buňku test na chromatin přístupný k transposáze pomocí sekvenování (scATAC-seq).[97]
Droplet Digital PCR
Droplet Digital PCR (ddPCR) je metoda dPCR, při které je 20 mikrolitrová reakce vzorku včetně testovacích primerů a buď Taqmanových sond nebo interkalačního barviva rozdělena na ~ 20 000 nanoliterních olejových kapiček přes vodní olej emulze technika, termocyklovaná do koncového bodu na 96jamkové PCR destičce a amplituda fluorescence byla odečtena pro všechny kapičky v každé jamce vzorku v průtokovém cytometru.[98]
Dějiny
dPCR vzešel z přístupu poprvé publikovaného v roce 1988 autorem Cetus Corporation když vědci prokázali, že jednotlivé molekuly β-globinu lze detekovat a amplifikovat pomocí PCR.[99][100] Toho bylo dosaženo rozdělením vzorku, takže některé reakce obsahovaly molekulu a jiné nikoli. V roce 1990 Peter Simmonds a AJ Brown poprvé použili tento koncept ke kvantifikaci molekuly.[101] Alex Morley a Pamela Sykes formálně zavedli metodu jako kvantitativní techniku v roce 1992.[40]
V roce 1999 vytvořili Bert Vogelstein a Kenneth Kinzler termín „digitální PCR“ a ukázali, že tuto techniku lze použít k nalezení vzácných mutací rakoviny.[102] Bylo však obtížné provést dPCR; bylo to náročné na práci, vyžadovalo to hodně školení, aby to bylo správné, a bylo těžké to udělat ve velkém množství. [102] V roce 2003 Kinzler a Vogelstein pokračovali v zdokonalování dPCR a vytvořili vylepšenou metodu, kterou nazvali BEAMing technologie, zkratka pro „korálky, emulze, zesílení a magnetika“. Nový protokol používal emulzi k rozdělení amplifikačních reakcí do jedné zkumavky. Tato změna umožnila vědcům škálovat metodu na tisíce reakcí v jednom běhu.[103][104][105]
Společnosti vyvíjející komerční systémy dPCR mají integrované technologie, jako je automatické dělení vzorků, digitální počítání cílů nukleových kyselin a zvyšování počtu kapiček, které mohou pomoci zefektivnit proces.[106][107][108] V posledních letech vědci vyvinuli a komercializovali diagnostiku založenou na dPCR pro několik podmínek, včetně nemalobuněčný karcinom plic a Downův syndrom.[109][110] První systém dPCR pro klinické použití byl v roce 2017 označen značkou CE a schválen USA Úřad pro kontrolu potravin a léčiv v roce 2019, pro diagnostiku chronická myeloidní leukémie.[111]
Reference
- ^ Perkel J (květen 2015). „Vedení našich experimentů s PCR“. Biotechniky. 58 (5): 217–21. doi:10.2144/000114283. PMID 25967899.
- ^ „Polymerase Chain Reaction (PCR)“. Národní centrum pro biotechnologické informace, Americká národní lékařská knihovna.
- ^ Duewer, David L .; et al. (2018). „Hodnocení kapkové digitální PCR pro kvantifikaci lidské genomové DNA: převod kopií na nanoliter na nanogramy nukleární DNA na mikroliter“. Analytická a bioanalytická chemie. 410 (12): 2879–2887. doi:10.1007 / s00216-018-0982-1. ISSN 1618-2642. PMC 5996397. PMID 29556737.
- ^ A b C Baker, Monya (2012). Msgstr "Digitální PCR zasáhne svůj krok". Přírodní metody. 9 (6): 541–544. doi:10.1038 / nmeth.2027. S2CID 46347563.
- ^ A b C Quan, Phenix-Lan; Sauzade, Martin; Brouzes, Eric (2018). „dPCR: Recenze technologie“. Senzory. 18 (4): 1271. doi:10,3390 / s18041271. ISSN 1424-8220. PMC 5948698. PMID 29677144.
- ^ Prediger E. „Digitální PCR (dPCR) - co to je a proč ji používat?“. Integrované technologie DNA.
- ^ A b C Pekin, Deniz; et al. (2011). „Kvantitativní a citlivá detekce vzácných mutací pomocí mikrofluidik na bázi kapiček“. Laboratoř na čipu. 11 (13): 2156–66. doi:10.1039 / c1lc20128j. ISSN 1473-0197. PMID 21594292.
- ^ Baker, Monya (01.06.2012). Msgstr "Digitální PCR zasáhne svůj krok". Přírodní metody. 9 (6): 541–544. doi:10.1038 / nmeth.2027. S2CID 46347563.
- ^ Witte, Anna Kristina; et al. (2016). "Hodnocení výkonnosti kvantitativní detekce lokusu Listeria monocytogenes prfA s kapičkovou digitální PCR". Analytická a bioanalytická chemie. 408 (27): 7583–7593. doi:10.1007 / s00216-016-9861-9. ISSN 1618-2642. PMC 5061835. PMID 27558101.
- ^ A b Stauber, Jennifer; et al. (2016). „Droplet digital PCR spolehlivě a reprodukovatelně kvantifikuje zánětlivé transkripty hostitele ve stolici.“. Buněčná imunologie. 303: 43–49. doi:10.1016 / j.cellimm.2016.03.007. ISSN 0008-8749. PMC 4863679. PMID 27063479.
- ^ A b C Skibo, Scott (23. února 2018). „Přistihlo se profilování nádoru k rakovině?“. Citováno 23. července 2019.
- ^ Hirsch, Fred (27. července 2018). „Pokyny zdůrazňují„ osvědčené postupy “pro tekutou biopsii během léčby nemalobuněčného karcinomu plic.“. Citováno 23. července 2019.
- ^ Johnson, Madeleine (12. ledna 2018). „Bio-Rad pokračuje v rozvoji technologie Digital PCR Tech, testy kapalné biopsie na komerční klinický trh“. Citováno 23. července 2019.
- ^ Oxnard, G. R .; et al. (2014). „Neinvazivní detekce odpovědi a rezistence u EGFR-mutantního karcinomu plic pomocí kvantitativní genotypizace nové generace bezbuněčné plazmové DNA nové generace“. Klinický výzkum rakoviny. 20 (6): 1698–1705. doi:10.1158 / 1078-0432.CCR-13-2482. ISSN 1078-0432. PMC 3959249. PMID 24429876.
- ^ Schütz, E .; et al. (2017). „Bezbuněčná DNA odvozená od štěpu, neinvazivní brzká rejekce a poškození štěpu při transplantaci jater: prospektivní, observační, multicentrická kohortní studie“. PLOS Medicine. 14 (4): e1002286. doi:10.1371 / journal.pmed.1002286. PMC 5404754. PMID 28441386.
- ^ Lee, S.Y .; Hwang, S.Y. (2015). „Aplikace digitální technologie polymerázové řetězové reakce pro neinvazivní prenatální test“. Journal of Genetic Medicine. 12 (2): 72–78. doi:10.5734 / JGM.2015.12.2.72. ISSN 2383-8442.
- ^ Gu, W .; et al. (2014). „Neinvazivní prenatální diagnostika u plodu ohroženého methylmalonovou acidemií“. Genetika v medicíně. 16 (7): 564–567. doi:10.1038 / gim.2013.194. PMC 4079742. PMID 24406457.
- ^ A b C d Strain, M .; et al. (2013). „Vysoce přesné měření HIV DNA pomocí Droplet Digital PCR“. PLOS ONE. 8 (4): e55943. Bibcode:2013PLoSO ... 855943S. doi:10.1371 / journal.pone.0055943. PMC 3616050. PMID 23573183.
- ^ A b Bell, Avery Davis; Usher, Christina L .; McCarroll, Steven A. (2018). Msgstr "Analýza variace počtu kopií pomocí Droplet Digital PCR". Digitální PCR. Metody v molekulární biologii. 1768. 143–160. doi:10.1007/978-1-4939-7778-9_9. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717442.
- ^ Shoda, Katsutoshi; et al. (2016). „Monitorování stavu počtu kopií HER2 v cirkulující nádorové DNA pomocí kapičkové digitální PCR u pacientů s rakovinou žaludku“. Rakovina žaludku. 20 (1): 126–135. doi:10.1007 / s10120-016-0599-z. ISSN 1436-3291. PMID 26874951.
- ^ A b Gevensleben, H .; et al. (2013). "Neinvazivní detekce amplifikace HER2 pomocí plazmové DNA digitální PCR". Klinický výzkum rakoviny. 19 (12): 3276–3284. doi:10.1158 / 1078-0432.CCR-12-3768. ISSN 1078-0432. PMC 6485473. PMID 23637122.
- ^ Torreggiani E, Rossini M, Bononi I, Pietrobon S, Mazzoni E, Iaquinta MR, Feo C, Rotondo JC, Rizzo P, Tognon M, Martini F (2019). „Protokol pro dlouhodobou kultivaci lidských primárních keratinocytů z normální kolorektální sliznice“. J Cell Physiol. 234 (7): 9895–9905. doi:10,1002 / jcp.27490. PMID 30362540.
- ^ Uchiyama, Yuri; et al. (2016). „Extrémně citlivá kapková digitální PCR pro detekci somatické mutace GNAQ s nízkou prevalencí u Sturge-Weberova syndromu“. Vědecké zprávy. 6 (1): 22985. Bibcode:2016NatSR ... 622985U. doi:10.1038 / srep22985. ISSN 2045-2322. PMC 4783707. PMID 26957145.
- ^ A b C Marusina, Kate (1. října 2017). „Positioning Digital PCR for Sharper Genomic Views“. Citováno 23. července 2019.
- ^ A b Kamitaki, Nolan; et al. (2018). "Použití Droplet Digital PCR k analýze alelově specifické RNA exprese". Digitální PCR. Metody v molekulární biologii. 1768. 401–422. doi:10.1007/978-1-4939-7778-9_23. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717456.
- ^ Millier, Melanie J .; et al. (2017). „Digitální PCR pro genovou expresi: dopad inherentní degradace tkáňové RNA“. Vědecké zprávy. 7 (1): 17235. Bibcode:2017NatSR ... 717235M. doi:10.1038 / s41598-017-17619-0. ISSN 2045-2322. PMC 5722939. PMID 29222437.
- ^ "Vysoce citlivá detekce hepatitidy B pomocí ddPCR". 12. dubna 2018. Citováno 23. července 2019.
- ^ Jang, Minjeong; et al. (2017). „Digitální systém PCR založený na kapkách pro detekci úrovně jednobuněčných patogenů přenášených potravinami“. BioChip Journal. 11 (4): 329–337. doi:10.1007 / s13206-017-1410-x. ISSN 2092-7843. S2CID 89829687.
- ^ Igarashi, Yuka; et al. (2017). „Jednobuněčné trasování vektorů u pacientů s ADA-SCID léčených genovou terapií kmenových buněk“. Molekulární terapie - metody a klinický vývoj. 6: 8–16. doi:10.1016 / j.omtm.2017.05.005. ISSN 2329-0501. PMC 5466583. PMID 28626778.
- ^ Albayrak, Cem; et al. (2016). „Digitální kvantifikace proteinů a mRNA v jednotlivých savčích buňkách“. Molekulární buňka. 61 (6): 914–24. doi:10.1016 / j.molcel.2016.02.030. ISSN 1097-2765. PMID 26990994.
- ^ Hussain, Musaddeq; et al. (2016). "Metoda přímé kapkové digitální PCR pro kvantifikaci zbytkové DNA v proteinových léčivech produkovaných v kvasinkových buňkách". Journal of Pharmaceutical and Biomedical Analysis. 123: 128–131. doi:10.1016 / j.jpba.2016.01.050. ISSN 0731-7085. PMID 26896631.
- ^ Miyaoka, Yuichiro; et al. (2014). "Izolace lidských bází editovaných lidských iPS buněk s jednou bází bez výběru antibiotik". Přírodní metody. 11 (3): 291–293. doi:10.1038 / nmeth.2840. PMC 4063274. PMID 24509632.
- ^ Mock, Ulrike; et al. (2016). „Digitální PCR k hodnocení frekvencí úprav genů (GEF-dPCR) zprostředkovaných návrhářskými nukleázami“. Přírodní protokoly. 11: 598–615. doi:10.1038 / nmeth.2840. PMC 4063274. PMID 24509632.
- ^ Nelson, C.E .; et al. (2015). „Úpravy genomu in vivo zlepšují funkci svalů na myším modelu Duchennovy svalové dystrofie“. Věda. 351 (6271): 403–407. doi:10.1126 / science.aad5143. ISSN 0036-8075. PMC 4883596. PMID 26721684.
- ^ Miyaoka, Yuichiro; et al. (2016). „Systematická kvantifikace HDR a NHEJ odhaluje účinky lokusu, nukleázy a buněčného typu na editaci genomu“. Vědecké zprávy. 61: 23549. Bibcode:2016NatSR ... 623549M. doi:10.1038 / srep23549. PMC 4814844. PMID 27030102.
- ^ Guttery, D. S .; et al. (2015). „Neinvazivní detekce aktivujících mutací estrogenového receptoru 1 (ESR1) v metastatickém karcinomu prsu pozitivním na estrogenový receptor“. Klinická chemie. 61 (7): 974–982. doi:10.1373 / clinchem.2015.238717. ISSN 0009-9147. PMID 25979954.
- ^ Robin, Jérôme D .; et al. (2016). „Porovnání metod kvantifikace DNA pro sekvenování příští generace“. Vědecké zprávy. 6 (1): 24067. Bibcode:2016NatSR ... 624067R. doi:10.1038 / srep24067. ISSN 2045-2322. PMC 4822169. PMID 27048884.
- ^ Aigrain, Louise; et al. (2016). „Kvantifikace efektivnosti protokolu přípravy knihovny sekvenování nové generace pomocí kapičkových digitálních PCR testů - systematické srovnání sad pro přípravu knihovny DNA pro sekvenování Illumina“. BMC Genomics. 17 (1): 458. doi:10.1186 / s12864-016-2757-4. ISSN 1471-2164. PMC 4906846. PMID 27297323.
- ^ A b Brunetto, Giovanna S .; et al. (2014). „Digital droplet PCR (ddPCR) pro přesnou kvantifikaci provirové zátěže lidského T-lymfotropního viru 1 v periferní krvi a mozkomíšním moku pacientů s HAM / TSP a identifikaci virových mutací“. Journal of NeuroVirology. 20 (4): 341–351. doi:10.1007 / s13365-014-0249-3. ISSN 1355-0284. PMC 4085507. PMID 24781526.
- ^ A b Sykes PJ, Neoh SH, Brisco MJ, Hughes E, Condon J, Morley AA (září 1992). Msgstr "Kvantifikace cílů pro PCR pomocí omezujícího ředění". Biotechniky. 13 (3): 444–9. PMID 1389177.
- ^ Vogelstein, B .; Kinzler, K. W. (1999). „Digital PCR“. Sborník Národní akademie věd. 96 (16): 9236–9241. Bibcode:1999PNAS ... 96.9236V. doi:10.1073 / pnas.96.16.9236. ISSN 0027-8424. PMC 17763. PMID 10430926.
- ^ A b Rački, Nejc; et al. (2014). „Digitální PCR s kapičkami reverzní transkriptázy vykazuje vysokou odolnost vůči inhibitorům PCR ze vzorků rostlin, půdy a vody“. Rostlinné metody. 10 (1): 42. doi:10.1186 / s13007-014-0042-6. ISSN 1746-4811. PMC 4307183. PMID 25628753.
- ^ Dingle, T. C .; et al. (2013). „Tolerance Droplet-Digital PCR vs. kvantitativní PCR v reálném čase na inhibiční látky“. Klinická chemie. 59 (11): 1670–1672. doi:10.1373 / clinchem.2013.211045. ISSN 0009-9147. PMC 4247175. PMID 24003063.
- ^ Dobnik, David; et al. (2018). "Multiplex Droplet Digital PCR Protocols for Quantification of GM Maize Events". Digitální PCR. Metody v molekulární biologii. 1768. str. 69–98. doi:10.1007/978-1-4939-7778-9_5. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717438.
- ^ Vellucci, Ashley; et al. (2018). "Použití Droplet Digital PCR k detekci koinfekce lidských herpesvirů 6A a 6B (HHV-6A a HHV-6B) v klinických vzorcích". Digitální PCR. Metody v molekulární biologii. 1768. 99–109. doi:10.1007/978-1-4939-7778-9_6. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717439.
- ^ Tagliapietra A, Rotondo JC, Bononi I, Mazzoni E, Magagnoli F, Maritati M (2019). "Droplet-digital PCR test k detekci polyomavirových sekvencí Merkelových buněk v choriových klcích ze samic potratu postižených samicemi". J Cell Physiol. 235 (3): 1888–1894. doi:10,1002 / jcp.29213. PMID 31549405.
- ^ Tagliapietra A, Rotondo JC, Bononi I, Mazzoni E, Magagnoli F, Maritati M, Contini C, Vesce F, Tognon M, Martini F (2019). „Stopy BK a JC polyomavirů ve vzorcích od žen postižených spontánním potratem“. Hum Reprod. 34 (3): 433–440. doi:10,1002 / jcp.27490. PMID 30590693.
- ^ Mazzoni E, Rotondo JC, Marracino L, Selvatici R, Bononi I, Torreggiani E, Touzé A, Martini F, Tognon MG (2017). „Detekce DNA polyomaviru z Merkelových buněk ve vzorcích séra zdravých dárců krve“. Přední Oncol. 7: 433–440. doi:10.3389 / fonc.2017.00294. PMC 5712532. PMID 29238698.
- ^ Tagliapietra A, Rotondo JC, Bononi I, Mazzoni E, Magagnoli F, Maritati M (2019). "Droplet-digital PCR test k detekci polyomavirových sekvencí Merkelových buněk v choriových klcích ze samic potratu postižených samicemi". J Cell Physiol. 235 (3): 1888–1894. doi:10,1002 / jcp.29213. PMID 31549405.
- ^ Tagliapietra A, Rotondo JC, Bononi I, Mazzoni E, Magagnoli F, Maritati M, Contini C, Vesce F, Tognon M, Martini F (2019). „Stopy BK a JC polyomavirů ve vzorcích od žen postižených spontánním potratem“. Hum Reprod. 34 (3): 433–440. doi:10,1002 / jcp.27490. PMID 30590693.
- ^ Podlesniy, Petar; et al. (2018). "Biomarkery v mozkomíšním moku: Analýza bezbuněčné cirkulující mitochondriální DNA pomocí digitální PCR". Digitální PCR. Metody v molekulární biologii. 1768. 111–126. doi:10.1007/978-1-4939-7778-9_7. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717440.
- ^ Cao, Yiping; et al. (2018). „Testování obecné a lidské fekální kontaminace ve vodách“. Digitální PCR. Metody v molekulární biologii. 1768. str. 127–140. doi:10.1007/978-1-4939-7778-9_8. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717441.
- ^ Li, Wentian; et al. (2009). "Detekce oblasti s variací počtu kopií a změnou počtu kopií pomocí kumulativních grafů". BMC bioinformatika. 10 (S1): S67. arXiv:0909.3129. Bibcode:2009arXiv0909.3129L. doi:10.1186 / 1471-2105-10-S1-S67. ISSN 1471-2105. PMC 2648736. PMID 19208171.
- ^ Koren, Amnon; et al. (2014). „Genetická variace v načasování replikace lidské DNA“. Buňka. 159 (5): 1015–1026. doi:10.1016 / j.cell.2014.10.025. ISSN 0092-8674. PMC 4359889. PMID 25416942.
- ^ Sanders, Sean (16. července 2008). „CNVs vs. SNPs: Understanding Human Structural Variation in Disease“. Citováno 24. července 2019.
- ^ Marshall, Christian R; et al. (2016). „Příspěvek variant počtu kopií ke schizofrenii ze studie zaměřené na celý genom zahrnující 41 321 subjektů“. Genetika přírody. 49 (1): 27–35. doi:10.1038 / ng.3725. ISSN 1061-4036. PMC 5737772. PMID 27869829.
- ^ Shlien, Adam; Malkin, David (2009). „Kopírovat variace počtu a rakovinu“. Genomová medicína. 1 (6): 62. doi:10,1186 / gm62. ISSN 1756-994X. PMC 2703871. PMID 19566914.
- ^ Lauer, Stephanie; Gresham, David (2019). Msgstr "Vyvíjející se pohled na varianty počtu kopií". Současná genetika. 65 (6): 1287–1295. doi:10.1007 / s00294-019-00980-0. ISSN 0172-8083. PMID 31076843. S2CID 149444714.
- ^ „Bylo zjištěno, že změna čísla souvisí s úmrtností na rakovinu“. 5. září 2018. Citováno 24. července 2019.
- ^ Gu, W .; Lupski, J. R. (2008). „CNV a nemoci nervového systému - co je nového?“. Cytogenetický a genomový výzkum. 123 (1–4): 54–64. doi:10.1159/000184692. ISSN 1424-8581. PMC 2920183. PMID 19287139.
- ^ Thapar, Anita; Cooper, Miriam (2013). „Varianta kopírování čísel: Co to je a co nás informovalo o dětských psychiatrických poruchách?“. Journal of the American Academy of Child & Adolescent Psychiatry. 52 (8): 772–774. doi:10.1016 / j.jaac.2013.05.013. ISSN 0890-8567. PMC 3919207. PMID 23880486.
- ^ Sekar, Aswin; et al. (2016). „Riziko schizofrenie z komplexní variace složky komplementu 4“. Příroda. 530 (7589): 177–183. Bibcode:2016Natur.530..177.. doi:10.1038 / příroda16549. ISSN 0890-8567. PMC 4752392. PMID 26814963.
- ^ Yim, Seon-Hee; et al. (2015). „Klinické důsledky variací počtu kopií u autoimunitních poruch“. Korean Journal of Internal Medicine. 30 (3): 294–304. doi:10.3904 / kjim.2015.30.3.294. ISSN 1226-3303. PMC 4438283. PMID 25995659.
- ^ On, Yijing; Hoskins, Janelle M .; McLeod, Howard L. (2011). „Kopírovat varianty čísel ve farmakogenetických genech“. Trendy v molekulární medicíně. 17 (5): 244–251. doi:10.1016 / j.molmed.2011.01.007. ISSN 1471-4914. PMC 3092840. PMID 21388883.
- ^ Gonzalez, E. (2005). „Vliv segmentových duplikací genů obsahujících CCL3L1 na citlivost na HIV-1 / AIDS“. Věda. 307 (5714): 1434–1440. Bibcode:2005Sci ... 307.1434G. doi:10.1126 / science.1101160. ISSN 0036-8075. PMID 15637236. S2CID 8815153.
- ^ A b Unutmaz, Derya; et al. (2010). „Varianta počtu kopií CCL3L1 a náchylnost k infekci HIV-1: metaanalýza“. PLOS ONE. 5 (12): e15778. Bibcode:2010PLoSO ... 515778L. doi:10.1371 / journal.pone.0015778. ISSN 1932-6203. PMC 3012711. PMID 21209899.
- ^ Dube, Simant; Qin, Jian; Ramakrishnan, Ramesh (2008). „Matematická analýza variace počtu kopií ve vzorku DNA pomocí digitální PCR na nanofluidním zařízení“. PLOS ONE. 3 (8): e2876. Bibcode:2008PLoSO ... 3.2876D. doi:10,1371 / journal.pone.0002876. ISSN 1932-6203. PMC 2483940. PMID 18682853.
- ^ Hughesman, Curtis B .; et al. (2017). „Detekce klinicky relevantních změn počtu kopií v progresi rakoviny ústní pomocí multiplexované kapky digitální PCR“. Vědecké zprávy. 7 (1): 11855. Bibcode:2017NatSR ... 711855H. doi:10.1038 / s41598-017-11201-4. ISSN 2045-2322. PMC 5605662. PMID 28928368.
- ^ A b Usher, Christina; et al. (2015). „Strukturální formy lokusu lidské amylázy a jejich vztah k SNP, haplotypům a obezitě“. Genetika přírody. 47 (8): 921–925. doi:10,1038 / ng. 3340. PMC 4712930. PMID 26098870.
- ^ Aldhous, Marian C .; et al. (2010). „Metody měření a přesnost při změně počtu kopií: nereplikace asociací počtu kopií beta-defensinu s Crohnovou chorobou“. Lidská molekulární genetika. 19 (24): 4930–4938. doi:10,1093 / hmg / ddq411. ISSN 1460-2083. PMC 2989891. PMID 20858604.
- ^ Pinheiro, Leonardo; Emslie, Kerry R. (2018). "Základní koncepty a validace digitálních PCR měření". Digitální PCR. Metody v molekulární biologii. 1768. str. 11–24. doi:10.1007/978-1-4939-7778-9_2. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717435.
- ^ Quan, Phenix-Lan; Sauzade, Martin; Brouzes, Eric (2018). „dPCR: Recenze technologie“. Senzory. 18 (4): 1271. doi:10,3390 / s18041271. ISSN 1424-8220. PMC 5948698. PMID 29677144.
- ^ Handsaker, Robert E; et al. (2015). „Velké variace počtu multiallelických kopií u lidí“. Genetika přírody. 47 (3): 296–303. doi:10.1038 / ng.3200. ISSN 1061-4036. PMC 4405206. PMID 25621458.
- ^ Garcia-Murillas, Isaac; Turner, Nicholas C. (2018). "Hodnocení amplifikace HER2 v plazmové cfDNA". Digitální PCR. Metody v molekulární biologii. 1768. 161–172. doi:10.1007/978-1-4939-7778-9_10. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717443.
- ^ Christgen, Matthias; van Luttikhuizen; et al. (2016). "Přesný ERBB2 hodnocení počtu kopií u rakoviny prsu pomocí analýzy pole sondy molekulární inverze ". Cílový cíl. 7 (50): 82733–82740. doi:10.18632 / oncotarget.12421. ISSN 1949-2553. PMC 5347728. PMID 27716627.
- ^ Borley, A; et al. (2014). „Dopad počtu kopií HER2 u IHC2 + / FISH-amplifikovaného karcinomu prsu na výsledek adjuvantní léčby trastuzumabem ve velké britské síti proti rakovině“. British Journal of Cancer. 110 (8): 2139–2143. doi:10.1038 / bjc.2014.147. ISSN 0007-0920. PMC 3992505. PMID 24691421.
- ^ Cao, Yiping; Raith, Meredith R .; Griffith, John F. (2015). "Droplet digital PCR pro simultánní kvantifikaci obecných a lidských fekálních indikátorů pro hodnocení kvality vody". Vodní výzkum. 70: 337–349. doi:10.1016 / j.watres.2014.12.008. ISSN 0043-1354. PMID 25543243.
- ^ Evropská společnost pro lékařskou onkologii (17. listopadu 2017). "Studie analyzuje mutace v mozkomíšním moku u rakoviny plic s mozkovými metastázami". Citováno 24. července 2019.
- ^ A b Petrone, Justin (8. června 2017). „Norský tým plánuje do konce roku debutovat s digitálním PCR založeným na testu rakoviny močového měchýře. Citováno 24. července 2019.
- ^ Hiemcke-Jiwa, Laura S .; et al. (2018). „Využití digitální metody kapiček v kapalných biopsiích: Vysoce citlivá technika pro detekci MYD88 str. (L265P) v mozkomíšním moku“. Hematologická onkologie. 36 (2): 429–435. doi:10.1002 / hon.2489. PMID 29210102. S2CID 4968214.
- ^ A b Paxton, Anne (říjen 2017). „Oživené naděje, nové výzvy s tekutou biopsií“. Citováno 24. července 2019.
- ^ Bhadra, Krish; Mellert, Hestia; Pestano, Gary (5. června 2017). „Přijetí testů kapalné biopsie pro NSCLC“. Citováno 24. července 2019.
- ^ Sacher, Adrian G .; Paweletz, Cloud; Dahlberg, Suzanne E. (2016). „Prospektivní validace genotypu rychlé plazmy pro detekci mutací EGFR a KRAS u pokročilého karcinomu plic“. JAMA Onkologie. 2 (8): 1014–1022. doi:10.1001 / jamaoncol.2016.0173. PMC 4982795. PMID 27055085.
- ^ Olsson, Eleonor; et al. (2015). „Sériové monitorování cirkulující nádorové DNA u pacientek s primárním karcinomem prsu za účelem detekce okultního metastatického onemocnění“. EMBO Molekulární medicína. 7 (8): 1034–1047. doi:10,15252 / emmm.201404913. ISSN 1757-4676. PMC 4551342. PMID 25987569.
- ^ Carpinetti, Paola; et al. (2015). „Použití personalizovaných biomarkerů a tekutých biopsií k monitorování odpovědi na léčbu a recidivy onemocnění u lokálně pokročilého karcinomu konečníku po neoadjuvantní chemoradiaci“. Cílový cíl. 6 (35): 38360–71. doi:10,18632 / oncotarget.5256. ISSN 1949-2553. PMC 4742005. PMID 26451609.
- ^ Reinert, Thomas; et al. (2016). „Analýza cirkulující nádorové DNA ke sledování chorobnosti po operaci kolorektálního karcinomu“. Střevo. 65 (4): 625–634. doi:10.1136 / gutjnl-2014-308859. ISSN 0017-5749. PMID 25654990.
- ^ Samimi, Goli; et al. (2015). „Personalizované biomarkery cirkulující nádorové DNA dynamicky předpovídají odpověď na léčbu a přežití u gynekologických nádorů“. PLOS ONE. 10 (12): e0145754. Bibcode:2015PLoSO..1045754P. doi:10.1371 / journal.pone.0145754. ISSN 1932-6203. PMC 4696808. PMID 26717006.
- ^ Dahmcke, Christina M .; et al. (2016). „Prospektivní slepé hodnocení testování DNA močí pro detekci karcinomu močového měchýře urotelu u pacientů s hrubou hematurií“. Evropská urologie. 70 (6): 916–919. doi:10.1016 / j.eururo.2016.06.035. ISSN 0302-2838. PMID 27417036.
- ^ Taylor, Sean S .; et al. (2015). „Optimalizace Droplet Digital PCR z RNA a DNA extraktů s přímým porovnáním s RT-qPCR: Klinické důsledky pro kvantifikaci subpopulací rezistentních na Oseltamivir“. Journal of Virological Methods. 224: 58–66. doi:10.1016 / j.jviromet.2015.08.014. PMID 26315318.
- ^ Whale, Alexandra S .; Huggett, Jim F .; Tzonev, Svilen (2016). „Základy multiplexování pomocí digitální PCR“. Biomolekulární detekce a kvantifikace. 10: 15–23. doi:10.1016 / j.bdq.2016.05.002. ISSN 2214-7535. PMC 5154634. PMID 27990345.
- ^ Slunce, Bing; Tao, Lian; Zheng, Yung-Ling (2014). „Simultánní kvantifikace alternativně sestřižených transkriptů v digitální PCR reakci s jednou kapkou“. Biotechniky. 56 (6): 319–325. doi:10.2144/000114179. PMID 24924392.
- ^ Valencia, C. Alexander; et al. (2012). „Hodnocení platforem pro obohacování cíle pomocí masivně paralelního sekvenování pro detekci mutací u vrozené svalové dystrofie“. The Journal of Molecular Diagnostics. 14 (3): 233–246. doi:10.1016 / j.jmoldx.2012.01.009. ISSN 1525-1578. PMC 3349841. PMID 22426012.
- ^ Brusgaard, Klaus; et al. (2015). „Jaká je nejlepší metoda obohacení NGS pro molekulární diagnostiku monogenního diabetu a obezity?“. PLOS ONE. 10 (11): e0143373. Bibcode:2015PLoSO..1043373P. doi:10.1371 / journal.pone.0143373. ISSN 1932-6203. PMC 4657897. PMID 26599467.
- ^ Ouellet, Eric; et al. (2015). „Hi-Fi SELEX: Vysoce věrná platforma pro objevování terapeutických aptamerů na bázi digitální PCR“. Biotechnologie a bioinženýrství. 112 (8): 1506–1522. doi:10,1002 / bit. 25881. ISSN 0006-3592. PMID 25727321. S2CID 39450798.
- ^ Ludlow, Andrew T .; et al. (2018). „ddTRAP: Metoda pro citlivou a přesnou kvantifikaci aktivity telomerázy“. Digitální PCR. Metody v molekulární biologii. 1768. str. 513–529. doi:10.1007/978-1-4939-7778-9_29. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMC 6046637. PMID 29717462.
- ^ Sayed, Mohammed E .; Slusher, Aaron L .; Ludlow, Andrew T. (2019). „Droplet Digital TRAP (ddTRAP): Přizpůsobení protokolu Telomere Repeat Amplification Protocol na Droplet Digital Polymerase Chain Reaction“. Žurnál vizualizovaných experimentů (147). doi:10.3791/59550. ISSN 1940-087X. PMID 31107456.
- ^ Stein, Richard A. (1. července 2019). „Single-Cell Sequencing Sifts through Multiple Omics“. Citováno 1. srpna 2019.
- ^ Wood-Bouwens, Christina M .; Ji, Hanlee P. (2018). "Jednobarevná multiplexovaná měření počtu kopií ddPCR a genotypování jednotlivých nukleotidových variant". Digitální PCR. Metody v molekulární biologii. 1768. 323–333. doi:10.1007/978-1-4939-7778-9_18. ISBN 978-1-4939-7776-5. ISSN 1064-3745. PMID 29717451.
- ^ Erlich, H. A .; Mullis, K. B .; Horn, G. T .; Higuchi, R .; Scharf, S. J .; Stoffel, S .; Gelfand, D. H .; Saiki, R. K. (29. ledna 1988). "Primerem řízená enzymatická amplifikace DNA termostabilní DNA polymerázou". Věda. 239 (4839): 487–491. Bibcode:1988Sci ... 239..487S. doi:10.1126 / science.239.4839.487. ISSN 0036-8075. PMID 2448875.
- ^ Morley, Alexander A. (1. září 2014). „Digital PCR: A brief history“. Biomolekulární detekce a kvantifikace. 1 (1): 1–2. doi:10.1016 / j.bdq.2014.06.001. ISSN 2214-7535. PMC 5129430. PMID 27920991.
- ^ Rutsaert, Sofie; Bosman, Kobus; Trypsteen, Wim; Nijhuis, Monique; Vandekerckhove, Linos (30. ledna 2018). „Digitální PCR jako nástroj pro měření perzistence HIV“. Retrovirologie. 15 (1): 16. doi:10.1186 / s12977-018-0399-0. ISSN 1742-4690. PMC 5789538. PMID 29378600.
- ^ A b Perkel, Jeff (11. dubna 2014). „Revoluce digitální PCR“. Citováno 22. července 2019.
- ^ Pohl G, Shih I (leden 2004). "Princip a aplikace digitální PCR". Odborný přehled molekulární diagnostiky. 4 (1): 41–7. doi:10.1586/14737159.4.1.41. PMID 14711348. S2CID 28271641.
- ^ Dressman D, Yan H, Traverso G, Kinzler KW, Vogelstein B (červenec 2003). „Transformace jednotlivých molekul DNA na fluorescenční magnetické částice pro detekci a stanovení počtu genetických variací“. Sborník Národní akademie věd Spojených států amerických. 100 (15): 8817–22. Bibcode:2003PNAS..100.8817D. doi:10.1073 / pnas.1133470100. PMC 166396. PMID 12857956.
- ^ Diehl F, Li M, Kinzler, KW, Vogelstein B, Dressman D (2006). „BEAMing: jednokomolekulární PCR na mikročásticích v emulzích voda v oleji“. Přírodní metody. 3 (7): 551–559. doi:10.1038 / nmeth898. PMID 16791214. S2CID 7059151.
- ^ Butkus, Ben (8. července 2010). „Digitální PCR prostor se zahřívá, protože prodejci nástrojů pro biologické vědy začínají sázet“. Citováno 22. července 2019.
- ^ Ramakrishnan R, Qin J, Jones RC, Weaver LS (2013). "Integrated Fluidic Circuits (IFCs) for digital PCR". Microfluidic Diagnostics. Metody v molekulární biologii. 949. pp. 423–31. doi:10.1007/978-1-62703-134-9_27. ISBN 978-1-62703-133-2. PMID 23329458.
- ^ Butkus, Ben (29 Mar 2012). "RainDance Launches Digital PCR Platform; Claims Sensitivity, Operating Cost Superiority". Citováno 22. července 2019.
- ^ "'Liquid biopsy' blood test detects genetic mutations in common form of lung cancer". 7 Apr 2016. Citováno 22. července 2019.
- ^ "Korea's BioCore First to Commercialize NIPT Based on Digital PCR". 2. března 2018. Citováno 22. července 2019.
- ^ "Bio-Rad Gets First CE Mark on Clinical ddPCR Test". 5. prosince 2017. Citováno 22. července 2019.
