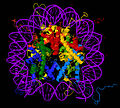
Nukleosom - Nucleosome

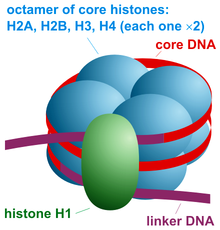
A nukleosom je základní strukturální jednotka DNA balení v eukaryoty. Struktura nukleosomu se skládá ze segmentu DNA navinutého kolem osmi histonové proteiny[1] a připomíná nit omotanou kolem cívky. Nukleosom je základní podjednotkou chromatin. Každý nukleosom se skládá z o něco méně než dvou závitů DNA omotaných kolem sady osmi proteinů zvaných histony, které jsou známé jako histonový oktamer. Každý histonový oktamer se skládá ze dvou kopií histonových proteinů H2A, H2B, H3, a H4.
DNA musí být zhuštěna do nukleosomů, aby se vešla do DNA buněčné jádro.[2] Kromě obalení nukleosomů eukaryotické chromatin se dále zhutňuje složením do řady složitějších struktur, případně tvoří a chromozóm. Každá lidská buňka obsahuje asi 30 milionů nukleosomů.[3]
Předpokládá se, že nukleozomy nesou epigeneticky zděděné informace v podobě kovalentní úpravy jejich jádra histony. Pozice nukleosomů v genomu nejsou náhodné a je důležité vědět, kde se každý nukleosom nachází, protože to určuje přístup DNA k regulační proteiny.[4]
Nukleosomy byly poprvé pozorovány jako částice v elektronovém mikroskopu Donem a Adou Olinsovými v roce 1974,[5] a jejich existenci a strukturu (jako histonové oktamery obklopené přibližně 200 bazickými páry DNA) navrhl Roger Kornberg.[6][7] Role nukleosomu jako obecného represoru genů byla prokázána Lorch et al. in vitro,[8] a Han a Grunstein in vivo v letech 1987 a 1988.[9]
Částice jádra nukleosomu se skládá z přibližně 146 základní páry (bp) z DNA[10] zabaleno v 1,67 levou rukou superhelikální zatáčky kolem a histon oktamer, skládající se ze 2 kopií každého z hlavních histonů H2A, H2B, H3, a H4.[11] Částice jádra jsou spojeny úseky spojovací DNA, který může mít délku až asi 80 bp. Technicky je nukleosom definován jako základní částice plus jedna z těchto spojovacích oblastí; toto slovo je však často synonymem pro jádrovou částici.[12] Mapy polohy nukleosomů v celém genomu jsou nyní k dispozici pro mnoho modelových organismů, včetně myších jater a mozku.[13]
Linkerové histony jako např H1 a jeho izoformy se podílejí na zhutňování chromatinu a sedí na bázi nukleosomu poblíž vstupu a výstupu DNA vázajícího se na spojovací oblast DNA.[14] Nekondenzované nukleosomy bez linkeru histonu připomínají „kuličky na řetězci DNA“ pod elektronový mikroskop.[15]
Na rozdíl od většiny eukaryotických buněk se z velké části používají zralé spermie protaminy aby zabalili svou genomovou DNA, s největší pravděpodobností dosáhnou ještě vyššího poměru balení.[16] Ekvivalenty histonu a zjednodušená struktura chromatinu byly také nalezeny v Archaea,[17] což naznačuje, že eukaryota nejsou jedinými organismy, které používají nukleosomy.
Struktura
Struktura jádrové částice

Přehled
Průkopnické strukturální studie skupiny Aarona Kluga v 80. letech poskytly první důkaz, že oktamer histonových proteinů obaluje DNA kolem sebe v přibližně 1,7 otáčkách levotočivého superhelixu.[18] V roce 1997 první téměř atomové rozlišení Krystalická struktura skupina nukleosomů byla vyřešena skupinou Richmond, ukazující nejdůležitější podrobnosti částice. Člověk alfa satelit palindromická DNA rozhodující pro dosažení krystalové struktury nukleosomů z roku 1997 byla vyvinuta skupinou Bunick v Národní laboratoři Oak Ridge v Tennessee.[19][20][21][22][23] Struktury více než 20 různých jaderných částic nukleosomu byly dosud vyřešeny,[24] včetně těch, které obsahují histonové varianty a histony z různých druhů. Struktura jádrové částice nukleosomu je pozoruhodně zachována a dokonce i změna více než 100 zbytků mezi histony žáby a kvasinek vede k mapám elektronové hustoty s celkovou odchylka od odmocniny pouze 1,6 Å.[25]
Částice jádra nukleosomu (NCP)
Částice jádra nukleosomu (znázorněná na obrázku) sestává z přibližně 146 základní pár DNA[10] zabaleno v 1,67 levou rukou superhelikální zatáčky okolo histonový oktamer, skládající se ze 2 kopií každého z hlavních histonů H2A, H2B, H3, a H4. Sousední nukleosomy jsou spojeny úsekem volné DNA nazývané spojovací DNA (jehož délka se pohybuje od 10 do 80 bp v závislosti na druhu a typu tkáně[17]Celá struktura generuje válec o průměru 11 nm a výšce 5,5 nm.

Částice jádra nukleosomu jsou pozorovány, když je chromatin v mezifázi zpracován tak, že způsobí částečné rozvinutí chromatinu. Výsledný obraz pomocí elektronového mikroskopu je „korálky na provázku“. Řetězec je DNA, zatímco každá kulička v nukleosomu je jádrová částice. Částice jádra nukleosomu se skládá z DNA a histonových proteinů.[29]
Částečný DNAse trávení chromatin odhaluje jeho strukturu nukleosomů. Protože části DNA jádra nukleosomových částic jsou pro DNAse méně přístupné než spojovací sekce, DNA se štěpí na fragmenty o délce rovnající se multiplicitě vzdálenosti mezi nukleosomy (180, 360, 540 párů bází atd.). Proto velmi charakteristický vzor podobný žebříku je viditelný během Gelová elektroforéza té DNA.[26] K takovému trávení může dojít také za přírodních podmínek během apoptóza ("buněčná sebevražda" nebo programovaná buněčná smrt), protože autodestrukce DNA obvykle je to jeho role.
Interakce proteinů v nukleosomu
Jádrové histonové proteiny obsahují charakteristický strukturální motiv označovaný jako „histonový záhyb“, který se skládá ze tří alfa-šroubovic (al-3) oddělených dvěma smyčkami (L1-2). V roztoku tvoří histony heterodimery H2A-H2B a heterotetramery H3-H4. Histony dimerizují kolem svých dlouhých a2 šroubovic v antiparalelní orientaci a v případě H3 a H4 tvoří dva takové dimery 4šroubovicový svazek stabilizovaný rozsáhlou interakcí H3-H3 '. Dimer H2A / H2B se váže na tetramer H3 / H4 v důsledku interakcí mezi H4 a H2B, které zahrnují tvorbu hydrofobního klastru.[11] Histonový oktamer je tvořen centrálním tetramerem H3 / H4 vloženým mezi dva dimery H2A / H2B. Vzhledem k vysoce základnímu náboji všech čtyř jádrových histonů je histonový oktamer stabilní pouze v přítomnosti DNA nebo velmi vysokých koncentrací solí.
Interakce histon - DNA
Nukleosom obsahuje více než 120 přímých interakcí protein-DNA a několik stovek vodou zprostředkovaných interakcí.[30] Přímé interakce protein - DNA se nešíří rovnoměrně po povrchu oktameru, ale spíše se nacházejí na diskrétních místech. Je to způsobeno tvorbou dvou typů vazebných míst pro DNA v oktameru; místo α1α1, které využívá spirálu α1 ze dvou sousedních histonů, a místo L1L2 tvořené smyčkami L1 a L2. Slané odkazy a vodíkové vazby mezi bazickými a hydroxylovými skupinami postranního řetězce a amidy hlavního řetězce s fosfáty páteře DNA tvoří podstatnou část interakcí s DNA. To je důležité vzhledem k tomu, že všudypřítomná distribuce nukleosomů podél genomů vyžaduje, aby se jednalo o nesekvenčně specifický faktor vázající DNA. Ačkoli mají nukleosomy tendenci upřednostňovat některé sekvence DNA před jinými,[31] jsou schopné vázat se prakticky na jakoukoli sekvenci, o které se předpokládá, že je to způsobeno flexibilitou při vytváření těchto vodou zprostředkovaných interakcí. Kromě toho dochází k nepolárním interakcím mezi postranními řetězci proteinů a deoxyribózovými skupinami a postranní řetězec argininu se interkaluje do menší drážky DNA na všech 14 místech, kde čelí povrchu oktameru. místa kolem povrchu oktameru narušují DNA uvnitř jádra nukleosomu. DNA je nerovnoměrně ohnutá a obsahuje také chyby zkroucení. Zákrut volné DNA ve formě B v roztoku je 10,5 bp za kolo. Celkový zkroucení nukleosomální DNA je však pouze 10,2 bp za kolo, lišící se od hodnoty 9,4 až 10,9 bp za kolo.
Histonové ocasní domény
Prodloužení histonového ocasu tvoří až 30% hmotnostních histonů, ale nejsou viditelné v krystalových strukturách nukleosomů kvůli jejich vysoké vnitřní flexibilitě a byly považovány za převážně nestrukturované.[32] N-koncové konce histonů H3 a H2B procházejí kanálem tvořeným vedlejšími drážkami dvou řetězců DNA, vyčnívajících z DNA každých 20 bp. The N-terminál ocas histonu H4, na druhé straně, má oblast vysoce bazických aminokyselin (16-25), která v krystalové struktuře tvoří interakci s vysoce kyselou povrchovou oblastí dimeru H2A-H2B jiného nukleosomu, být potenciálně relevantní pro strukturu vyšších řádů nukleosomů. Předpokládá se, že k této interakci dochází také za fyziologických podmínek acetylace ocasu H4 narušuje strukturu vyššího řádu chromatinu.
Struktura vyšší objednávky

Organizace DNA, jíž je dosaženo nukleosomem, nemůže plně vysvětlit obal DNA pozorovaný v buněčném jádru. Další zhutnění chromatin do buněčného jádra je nezbytný, ale dosud není dobře pochopen. Současné porozumění[24] je to, že opakující se nukleosomy s intervenující „linkerovou“ DNA tvoří a 10-nm vlákno, popsané jako „korálky na provázku“, a mají poměr balení asi pět až deset.[17] Řetězec nukleosomů může být uspořádán do a 30 nm vlákno, zhutněná struktura s poměrem balení ~ 50[17] a jehož tvorba závisí na přítomnosti H1 histon.
Byla představena krystalová struktura tetranukleosomu, která byla použita k vytvoření navrhované struktury 30 nm vlákna jako dvousložkové šroubovice.[33]O tomto modelu stále existuje určité množství sporů, protože je neslučitelný s nedávným elektronová mikroskopie data.[34] Kromě toho je struktura chromatinu špatně pochopena, ale klasicky se navrhuje, že 30 nm vlákno je uspořádáno do smyček podél centrálního proteinového lešení za vzniku transkripčně aktivního euchromatin. Další zhutnění vede k transkripčně neaktivnímu heterochromatin.
Dynamika
I když je nukleosom velmi stabilní komplex protein-DNA, není statický a bylo prokázáno, že prochází řadou různých strukturálních úprav, včetně klouzání nukleosomů a expozice místa DNA. V závislosti na kontextu mohou nukleosomy inhibovat nebo usnadňovat vazbu transkripčního faktoru. Polohy nukleosomů jsou řízeny třemi hlavními příspěvky: Zaprvé, vnitřní vazebná afinita histonového oktameru závisí na sekvenci DNA. Za druhé, nukleosom může být přemístěn nebo rekrutován kompetitivní nebo kooperativní vazbou jiných proteinových faktorů. Za třetí, nukleosom může být aktivně translokován remodelačními komplexy závislými na ATP.[35]
Klouzání nukleosomu
Práce prováděné v Bradburyho laboratoři ukázaly, že nukleosomy rekonstituované na 5S DNA poziční sekvenci se dokázaly translačně přemístit na sousední sekvence, když byly inkubovány tepelně.[36] Pozdější práce ukázaly, že toto přemístění nevyžadovalo narušení histonového oktameru, ale bylo v souladu s tím, že nukleosomy byly schopné „klouzat“ po DNA v cis. V roce 2008 se dále ukázalo, že CTCF vazebná místa fungují jako kotvy umisťující nukleosomy, takže při použití k seřazení různých genomových signálů lze snadno identifikovat více sousedících nukleosomů.[37] Ačkoli jsou nukleosomy vnitřně mobilní, eukaryoty vyvinuly velkou rodinu ATP-závislých enzymů remodelujících chromatiny, aby změnily strukturu chromatinu, z nichž mnohé to dělají klouzáním nukleosomů. V roce 2012 laboratoř Beeny Pillai prokázala, že klouzání nukleosomů je jedním z možných mechanismů pro tkáňově specifickou expresi genů ve velkém měřítku. Práce ukazuje, že počáteční místo transkripce pro geny exprimované v konkrétní tkáni jsou vyčerpány nukleosomy, zatímco stejná sada genů v jiné tkáni, kde nejsou exprimovány, je vázána na nukleosomy.[13]
Expozice místa DNA
Práce laboratoře Widom ukázala, že nukleosomální DNA je v rovnováze mezi zabaleným a nezabaleným stavem. Měření těchto rychlostí pomocí časově rozlišeného FRET odhalili, že DNA v nukleosomu zůstává plně zabalená pouze 250 ms, než je rozbalena po dobu 10-50 ms a poté rychle znovu zabalena.[38] To znamená, že DNA nemusí být aktivně oddělena od nukleosomu, ale že existuje značný zlomek času, během kterého je plně přístupná. To lze skutečně rozšířit na pozorování, že zavedení sekvence vázající DNA do nukleosomu zvyšuje dostupnost sousedních oblastí DNA, když jsou vázány.[39] Tato tendence DNA „dýchat“ v nukleosomu má důležité funkční důsledky pro všechny proteiny vázající DNA, které působí v prostředí chromatinu.[38] Zejména dynamické dýchání nukleosomů hraje důležitou roli při omezování pokroku RNA polymeráza II během prodloužení transkripce.[40]
Oblast bez nukleosomů
Promotory aktivních genů mají oblasti bez nukleosomů (NFR). To umožňuje přístup promotorové DNA k různým proteinům, jako jsou transkripční faktory. Oblast bez nukleosomů obvykle zahrnuje 200 nukleotidů v S. cerevisae[41] Dobře umístěné nukleosomy tvoří hranice NFR. Tyto nukleosomy se nazývají + 1-nukleosom a -1-nukleosom a jsou umístěny v kanonických vzdálenostech po proudu a proti směru od startovacího místa transkripce.[42] + 1-nukleosom a několik downstream nukleosomů mají také tendenci inkorporovat histonovou variantu H2A.Z.[42]
Modulační struktura nukleosomů
Eukaryotické genomy jsou všudypřítomně asociovány s chromatinem; buňky však musí prostorově a časově regulovat specifické lokusy nezávisle na objemovém chromatinu. Za účelem dosažení vysoké úrovně kontroly potřebné ke koordinaci jaderných procesů, jako je replikace, oprava a transkripce DNA, vyvinuli buňky řadu prostředků k lokální a specifické modulaci struktury a funkce chromatinu. To může zahrnovat kovalentní modifikaci histonů, inkorporaci histonových variant a nekovalentní remodelaci pomocí ATP závislých remodelačních enzymů.
Posttranslační modifikace histonu

Od té doby, co byly objeveny v polovině 60. let, se předpokládalo, že modifikace histonu ovlivní transkripci.[43] Skutečnost, že většina nalezených časných posttranslačních modifikací byla koncentrována v prodloužení ocasu, které vyčnívají z jádra nukleosomu, vedla ke dvěma hlavním teoriím týkajícím se mechanismu modifikace histonu. První z teorií naznačovala, že mohou ovlivnit elektrostatické interakce mezi histonovými konci a DNA a „uvolnit“ strukturu chromatinu. Později bylo navrženo, že kombinace těchto modifikací mohou vytvářet vazebné epitopy, kterými se získávají další proteiny.[44] Nedávno, vzhledem k tomu, že ve strukturovaných oblastech histonů bylo nalezeno více modifikací, bylo předloženo, že tyto modifikace mohou ovlivnit histon-DNA[45] a histon-histon[46] interakce uvnitř jádra nukleosomu. Předpokládá se, že modifikace (jako je acetylace nebo fosforylace), které snižují náboj globulárního histonového jádra, „uvolní“ asociaci jádro-DNA; síla účinku závisí na umístění modifikace uvnitř jádra.[47]Ukázalo se, že některé modifikace korelují s umlčováním genů; jiné se zdají být v korelaci s aktivací genů. Mezi běžné úpravy patří acetylace, methylace nebo ubikvitinace z lysin; methylace z arginin; a fosforylace z serin. Takto uložené informace jsou brány v úvahu epigenetický, protože není kódován v DNA, ale je stále zděděn v dceřiných buňkách. Udržování potlačeného nebo aktivovaného stavu genu je často nutné buněčná diferenciace.[17]
Histonové varianty
Ačkoli jsou histony v průběhu evoluce pozoruhodně konzervovány, bylo identifikováno několik variantních forem. Tato diverzifikace funkce histonu je omezena na H2A a H3, přičemž H2B a H4 jsou většinou neměnné. H2A lze nahradit H2AZ (což vede ke snížené stabilitě nukleosomů) nebo H2AX (který je spojen s opravou DNA a T buňka diferenciace), zatímco neaktivní X chromozomy u savců jsou obohaceny o makroH2A. H3 může být nahrazen H3.3 (který koreluje s aktivovanými geny a regulačními prvky) a v centromery H3 se nahrazuje CENPA.[17]
Remodelace nukleosomů závislá na ATP
S výrazem je spojena řada odlišných reakcí Remodelace chromatinu závislá na ATP. Bylo prokázáno, že remodelační enzymy posouvají nukleosomy podél DNA,[48] narušit kontakty histon-DNA do té míry, že destabilizuje dimer H2A / H2B[49][50] a generovat negativní superhelikální torzi v DNA a chromatinu.[51] Nedávno bylo prokázáno, že enzym remodelace Swr1 zavádí variantu histonu H2A.Z do nukleosomů.[52] V současné době není jasné, zda všechny tyto reakce představují odlišné reakce nebo pouze alternativní výsledky společného mechanismu. To, co je sdílené mezi všemi a ve skutečnosti charakteristickým znakem remodelace chromatinu závislé na ATP, je to, že všechny vedou ke změně dostupnosti DNA.
Studie zabývající se aktivací genů in vivo[53] a ještě úžasnější je přestavba in vitro[54] odhalili, že události remodelace chromatinu a vazba transkripčních faktorů jsou cyklické a periodické povahy. I když důsledky toho pro reakční mechanismus remodelace chromatinu nejsou známy, dynamická povaha systému mu umožňuje umožnit rychlejší reakci na vnější podněty. Nedávná studie naznačuje, že se pozice nukleosomů významně mění během vývoje myších embryonálních kmenových buněk a tyto změny souvisejí s vazbou vývojových transkripčních faktorů.[55]
Dynamická remodelace nukleosomu napříč genomem kvasinek
Studie z roku 2007 katalogizovaly polohy nukleosomů v kvasinkách a ukázaly, že nukleosomy jsou vyčerpány promotér regiony a počátky replikace.[56][57][58]Asi 80% kvasinkového genomu se zdá být pokryto nukleosomy[59] a vzor umístění nukleosomů jasně souvisí s oblastmi DNA, které regulují transkripce, oblasti, které jsou transkribovány a oblasti, které iniciují replikaci DNA.[60] V poslední době byla zkoumána nová studie dynamické změny v přemisťování nukleosomů během události globálního přepisování transkripce k objasnění účinků na přemístění nukleosomů během transkripčních změn v kvasinkách v celém genomu (Saccharomyces cerevisiae ).[61] Výsledky naznačují, že nukleosomy, které byly lokalizovány do promotorových oblastí, jsou nahrazeny v reakci na stres (jako tepelný šok ). Kromě toho odstranění nukleosomů obvykle odpovídalo transkripční aktivaci a nahrazení nukleosomů obvykle odpovídalo transkripční represi, pravděpodobně proto, že transkripční faktor vazebné stránky se staly více či méně přístupnými. Obecně pouze jeden nebo dva nukleosomy byly přemístěny na promotor, aby se provedly tyto transkripční změny. Avšak i v chromozomálních oblastech, které nebyly spojeny se změnami transkripce, bylo pozorováno přemístění nukleosomů, což naznačuje, že zakrytí a odkrytí transkripční DNA nemusí nutně vést k transkripční události. Po transkripci musí oblast rDNA chránit před jakýmkoli poškozením, což naznačuje, že proteiny HMGB hrají hlavní roli při ochraně oblasti bez nukleosomů.[62][63]
Sestavení nukleosomu in vitro

Lze sestavit nukleosomy in vitro buď použitím purifikovaných nativních nebo rekombinantních histonů.[64][65] Jedna standardní technika zavádění DNA kolem histonů zahrnuje použití soli dialýza. Reakce skládající se z histonových oktamerů a nahé šablony DNA může být inkubována společně při koncentraci soli 2 M. Postupným snižováním koncentrace soli se DNA ekvilibruje do polohy, kde je obalena kolem histonových oktamerů a tvoří nukleosomy. Za vhodných podmínek umožňuje tento rekonstituční proces experimentální mapování afinity polohování nukleosomů dané sekvence.[66]
Disulfidové zesítěné částice jádra nukleosomu
Nedávný pokrok ve výrobě částic jádra nukleosomu se zvýšenou stabilitou zahrnuje místně specifické disulfid křížové odkazy.[67] Do částice nukleosomu jádra mohou být zavedeny dvě různé příčné vazby. První z nich zesíťuje dvě kopie H2A prostřednictvím zavedeného cysteinu (N38C), což má za následek histonový oktamer který je stabilní proti ztrátě dimeru H2A / H2B během rekonstituce nukleosomu. Druhé zesítění může být zavedeno mezi H3 N-koncový histonový konec a konce nukleosomové DNA prostřednictvím začleněného konvertibilního nukleotidu.[68] Zesíťování DNA-histon oktamer stabilizuje jádrové částice nukleosomu proti disociaci DNA při velmi nízkých koncentracích částic a při zvýšených koncentracích solí.
Sestavení nukleosomu in vivo

Nukleosomy jsou základní balicí jednotkou DNA vytvořenou z histonových proteinů, kolem nichž je DNA stočena. Slouží jako kostra pro tvorbu chromatinové struktury vyššího řádu i pro vrstvu regulační kontroly genové exprese. Nukleosomy se rychle skládají na nově syntetizovanou DNA za replikační vidličkou.
H3 a H4
Histony H3 a H4 z rozložených starých nukleosomů jsou drženy v blízkosti a náhodně distribuovány na nově syntetizované DNA.[69] Jsou sestaveny komplexem chromatinového montážního faktoru 1 (CAF-1), který se skládá ze tří podjednotek (p150, p60 a p48).[70] Nově syntetizované H3 a H4 jsou sestaveny pomocí replikačního vazebného montážního faktoru (RCAF). RCAF obsahuje podjednotku Asf1, která se váže na nově syntetizované proteiny H3 a H4.[71] Staré proteiny H3 a H4 si zachovávají své chemické modifikace, které přispívají k přenosu epigenetického podpisu. Nově syntetizované proteiny H3 a H4 se v rámci procesu zrání chromatinu postupně acetylují na různých lysinových zbytcích.[72] Rovněž se předpokládá, že staré proteiny H3 a H4 v nových nukleosomech přijímají enzymy modifikující histon, které označují nové histony, což přispívá k epigenetické paměti.
H2A a H2B
Na rozdíl od staré H3 a H4 staré H2A a H2B histonové proteiny se uvolňují a degradují; proto jsou nově sestavené proteiny H2A a H2B inkorporovány do nových nukleosomů.[73] H2A a H2B jsou sestaveny do dimerů, které jsou poté naneseny na nukleosomy pomocí proteinu 1 nukleosomu (NAP-1), který také napomáhá klouzání nukleosomů.[74] Nukleosomy jsou také rozmístěny pomocí ATP-závislých komplexů remodelačních nukleosomů obsahujících enzymy jako Isw1 Ino80 a Chd1 a následně sestaveny do struktury vyššího řádu.[75][76]
Galerie
Krystalová struktura částice jádra nukleosomu (PDB: 1EQZ[27][28]) - různé pohledy zobrazující podrobnosti skládání a organizace histonů. Histony H2A, H2B, H3, H4 a DNA jsou barevné.
Viz také
Reference
- ^ Reece J, Campbell N (2006). Biologie. San Francisco: Benjamin Cummings. ISBN 978-0-8053-6624-2.
- ^ Alberts B (2002). Molekulární biologie buňky (4. vydání). New York: Garland Science. p. 207. ISBN 978-0-8153-4072-0.
- ^ Chromozomální DNA a její obal v chromatinovém vláknu.
- ^ Teif VB, Clarkson CT (2019). "Nucleosome Positioning". Encyklopedie bioinformatiky a výpočetní biologie. 2: 308–317. doi:10.1016 / B978-0-12-809633-8.20242-2. ISBN 9780128114322.
- ^ Olins AL, Olins DE (leden 1974). Msgstr "Sféroidní chromatinové jednotky (v tělech)". Věda. 183 (4122): 330–2. Bibcode:1974Sci ... 183..330O. doi:10.1126 / science.183.4122.330. PMID 4128918. S2CID 83480762.
- ^ McDonald D (prosinec 2005). „Milestone 9, (1973-1974) The nucleosome hypotéza: Alternativní strunová teorie“. Přírodní milníky: genový výraz. doi:10.1038 / nrm1798.
- ^ Kornberg RD (květen 1974). "Chromatinová struktura: opakující se jednotka histonů a DNA". Věda. 184 (4139): 868–71. Bibcode:1974Sci ... 184..868K. doi:10.1126 / science.184.4139.868. PMID 4825889.
- ^ Lorch Y, LaPointe JW, Kornberg RD (duben 1987). „Nukleosomy inhibují iniciaci transkripce, ale umožňují prodloužení řetězce s vytěsněním histonů“. Buňka. 49 (2): 203–10. doi:10.1016/0092-8674(87)90561-7. PMID 3568125. S2CID 21270171.
- ^ Han M, Grunstein M (1988). „Ztráta nukleosomů aktivuje in vivo promotory kvasinek“. Buňka. 55 (6): 1137–45. doi:10.1016/0092-8674(88)90258-9. PMID 2849508. S2CID 41520634.
- ^ A b U různých krystalů byly pozorovány hodnoty 146 a 147 párů bází
- ^ A b Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (září 1997). "Krystalová struktura částice jádra nukleosomu v rozlišení 2,8 A". Příroda. 389 (6648): 251–60. Bibcode:1997 Natur.389..251L. doi:10.1038/38444. PMID 9305837. S2CID 4328827.
- ^ Alberts B (2007). Molekulární biologie buňky (5. vydání). New York: Garland Science. p. 211. ISBN 978-0-8153-4106-2.
- ^ A b Bargaje R, Alam MP, Patowary A, Sarkar M, Ali T, Gupta S, Garg M, Singh M, Purkanti R, Scaria V, Sivasubbu S, Brahmachari V, Pillai B (říjen 2012). „Blízkost nukleosomu obsahujícího H2A.Z k počátečnímu místu transkripce ovlivňuje hladiny genové exprese v játrech a mozku savců“. Výzkum nukleových kyselin. 40 (18): 8965–78. doi:10.1093 / nar / gks665. PMC 3467062. PMID 22821566.
- ^ Zhou YB, Gerchman SE, Ramakrishnan V, Travers A, Muyldermans S (září 1998). "Poloha a orientace globulární domény linkeru histonu H5 na nukleosomu". Příroda. 395 (6700): 402–5. Bibcode:1998 Natur.395..402Z. doi:10.1038/26521. PMID 9759733. S2CID 204997317.
- ^ Thoma F, Koller T, Klug A (listopad 1979). „Zapojení histonu H1 do organizace nukleosomu a solí závislých nadstaveb chromatinu“. The Journal of Cell Biology. 83 (2 Pt 1): 403–27. doi:10.1083 / jcb.83.2.403. PMC 2111545. PMID 387806.
- ^ Clarke HJ (1992). "Jaderné a chromatinové složení savčích gamet a raných embryí". Biochemie a buněčná biologie. 70 (10–11): 856–66. doi:10.1139 / o92-134. PMID 1297351.
- ^ A b C d E F Felsenfeld G, Groudine M (leden 2003). „Ovládání dvojité šroubovice“. Příroda. 421 (6921): 448–53. Bibcode:2003 Natur.421..448F. doi:10.1038 / nature01411. PMID 12540921.
- ^ Struktura částice jádra nukleosomu při rozlišení 7 Á, Richmond, T., Finch, J.T., Rushton, B., Rhodes, D. a Klug, A. (1984) Nature 311, 532-37
- ^ Harp JM, Palmer EL, York MH, Gewiess A, Davis M, Bunick GJ (říjen 1995). „Preparativní separace částic jádra nukleosomu obsahující DNA s definovanou sekvencí ve více translačních fázích“. Elektroforéza. 16 (10): 1861–4. doi:10,1002 / elps.11501601305. PMID 8586054.
- ^ Palmer EL, Gewiess A, Harp JM, York MH, Bunick GJ (1995). "Velkovýroba fragmentů DNA palindromu". Analytická biochemie. 231 (1): 109–14. doi:10.1006 / abio.1995.1509. PMID 8678288.
- ^ Harp JM, Uberbacher EC, Roberson AE, Palmer EL, Gewiess A, Bunick GJ (1996). „Rentgenová difrakční analýza krystalů obsahujících dvojnásobné symetrické částice jádra nukleosomu“. Acta Crystallographica oddíl D. 52 (Pt 2): 283–8. doi:10.1107 / S0907444995009139. PMID 15299701.
- ^ Harp JM, Hanson BL, Timm DE, Bunick GJ (2000). "Asymetrie v jádrové částice nukleosomu při rozlišení 2,5 A". Acta Crystallographica oddíl D. 56 (Pt 12): 1513–34. doi:10.1107 / s0907444900011847. PMID 11092917.
- ^ Hanson BL, Alexander C, Harp JM, Bunick GJ (2004). "Příprava a krystalizace jádrové částice nukleosomu". Metody v enzymologii. 375: 44–62. doi:10.1016 / s0076-6879 (03) 75003-4. ISBN 9780121827793. PMID 14870658.
- ^ A b Chakravarthy S, Park YJ, Chodaparambil J, Edayathumangalam RS, Luger K (únor 2005). "Struktura a dynamické vlastnosti částic jádra nukleosomu". FEBS Dopisy. 579 (4): 895–8. doi:10.1016 / j.febslet.2004.11.030. PMID 15680970.
- ^ White CL, Suto RK, Luger K (září 2001). „Struktura jádrové části kvasinkového nukleosomu odhaluje zásadní změny v internukleozomových interakcích“. Časopis EMBO. 20 (18): 5207–18. doi:10.1093 / emboj / 20.18.5207. PMC 125637. PMID 11566884.
- ^ A b Stryer L (1995). Biochemie (čtvrté vydání). New York - Basingstoke: W. H. Freeman and Company. ISBN 978-0716720096.
- ^ A b Harp JM, Hanson BL, Timm DE, Bunick GJ (6. dubna 2000). „Rentgenová struktura částice jádra nukleosomu v rozlišení 2,5 A“. RCSB Proteinová datová banka (PDB). doi:10,2210 / pdb1eqz / pdb. ID PDB: 1EQZ. Citováno 8. října 2012. Citovat deník vyžaduje
| deník =(Pomoc) - ^ A b Harp JM, Hanson BL, Timm DE, Bunick GJ (prosinec 2000). "Asymetrie v jádrové částice nukleosomu při rozlišení 2,5 A". Acta Crystallographica oddíl D. 56 (Pt 12): 1513–34. doi:10.1107 / S0907444900011847. PMID 11092917. ID PDB: 1EQZ.
- ^ Alberts, Bruce. Základní buněčná biologie. 2. vyd. New York: Garland Science, 2009. Tisk.
- ^ Davey CA, Sargent DF, Luger K, Maeder AW, Richmond TJ (červen 2002). "Interakce zprostředkované rozpouštědly ve struktuře jádrové částice nukleosomu při rozlišení 1,9". Journal of Molecular Biology. 319 (5): 1097–113. doi:10.1016 / S0022-2836 (02) 00386-8. PMID 12079350.
- ^ Segal E, Fondufe-Mittendorf Y, Chen L, Thåström A, Field Y, Moore IK, Wang JP, Widom J (srpen 2006). „Genomický kód pro určování polohy nukleosomů“. Příroda. 442 (7104): 772–8. Bibcode:2006 Natur.442..772S. doi:10.1038 / nature04979. PMC 2623244. PMID 16862119.
- ^ Zheng C, Hayes JJ (duben 2003). "Struktury a interakce jádrových histonových koncových domén". Biopolymery. 68 (4): 539–46. doi:10,1002 / bip.10303. PMID 12666178.
- ^ Schalch T, Duda S, Sargent DF, Richmond TJ (červenec 2005). „Rentgenová struktura tetranukleosomu a její důsledky pro chromatinové vlákno“. Příroda. 436 (7047): 138–41. Bibcode:2005 Natur.436..138S. doi:10.1038 / nature03686. PMID 16001076. S2CID 4387396.
- ^ Robinson PJ, Fairall L, Huynh VA, Rhodes D (duben 2006). „EM měření definují rozměry chromatinového vlákna„ 30 nm “: důkaz kompaktní, vzájemně propojené struktury“. Sborník Národní akademie věd Spojených států amerických. 103 (17): 6506–11. Bibcode:2006PNAS..103.6506R. doi:10.1073 / pnas.0601212103. PMC 1436021. PMID 16617109.
- ^ Teif VB, Rippe K (září 2009). „Predikce pozic nukleosomů na DNA: kombinace preferencí vnitřní sekvence a aktivit remodelace“. Výzkum nukleových kyselin. 37 (17): 5641–55. doi:10.1093 / nar / gkp610. PMC 2761276. PMID 19625488.
- ^ Pennings S, Muyldermans S, Meersseman G, Wyns L (květen 1989). „Tvorba, stabilita a umístění histonů jádra nukleosomů znovu sestavených na ohnuté a jiné DNA odvozené od nukleosomů“. Journal of Molecular Biology. 207 (1): 183–92. doi:10.1016 / 0022-2836 (89) 90449-X. PMID 2738923.
- ^ Fu Y, Sinha M, Peterson CL, Weng Z (červenec 2008). Van Steensel B (ed.). „Izolátor vázající protein CTCF umisťuje 20 nukleosomů kolem svých vazebných míst napříč lidským genomem“. Genetika PLOS. 4 (7): e1000138. doi:10.1371 / journal.pgen.1000138. PMC 2453330. PMID 18654629.
- ^ A b Li G, Levitus M, Bustamante C, Widom J (leden 2005). "Rychlá spontánní dostupnost nukleosomální DNA". Přírodní strukturní a molekulární biologie. 12 (1): 46–53. doi:10.1038 / nsmb869. PMID 15580276. S2CID 14540078.
- ^ Li G, Widom J (srpen 2004). „Nukleosomy usnadňují jejich vlastní invazi“. Přírodní strukturní a molekulární biologie. 11 (8): 763–9. doi:10.1038 / nsmb801. PMID 15258568. S2CID 11299024.
- ^ Hodges C, Bintu L, Lubkowska L, Kashlev M, Bustamante C (červenec 2009). „Nukleosomální fluktuace určují dynamiku transkripce RNA polymerázy II“. Věda. 325 (5940): 626–8. Bibcode:2009Sci ... 325..626H. doi:10.1126 / science.1172926. PMC 2775800. PMID 19644123.
- ^ Yuan GC, Liu YJ, Dion MF, Slack MD, Wu LF, Altschuler SJ, Rando OJ (červenec 2005). "Identifikace pozic nukleosomů v S. cerevisiae v měřítku genomu". Věda. 309 (5734): 626–30. Bibcode:2005Sci ... 309..626Y. doi:10.1126 / science.1112178. PMID 15961632. S2CID 43625066.
- ^ A b Lai WK, Pugh BF (září 2017). „Porozumění dynamice nukleosomů a jejich vazbám na genovou expresi a replikaci DNA“. Nature Reviews Molecular Cell Biology. 18 (9): 548–562. doi:10.1038 / nrm.2017.47. PMC 5831138. PMID 28537572.
- ^ Allfrey VG, Faulkner R, Mirsky AE (květen 1964). „acetylace a methylace histonů a jejich možná role v regulaci syntézy RNA“. Sborník Národní akademie věd Spojených států amerických. 51 (5): 786–94. Bibcode:1964PNAS ... 51..786A. doi:10.1073 / pnas.51.5.786. PMC 300163. PMID 14172992.
- ^ Strahl BD, Allis CD (leden 2000). "Jazyk kovalentních modifikací histonu". Příroda. 403 (6765): 41–5. Bibcode:2000Natur.403 ... 41S. doi:10.1038/47412. PMID 10638745. S2CID 4418993.
- ^ Cosgrove MS, Boeke JD, Wolberger C (listopad 2004). "Regulovaná mobilita nukleosomů a histonový kód". Přírodní strukturní a molekulární biologie. 11 (11): 1037–43. doi:10.1038 / nsmb851. PMID 15523479. S2CID 34704745.
- ^ Ye J, Ai X, Eugeni EE, Zhang L, Carpenter LR, Jelinek MA, Freitas MA, Parthun MR (duben 2005). „Acetylace histonu H4 lysinu 91 a modifikace jádrové domény spojená se sestavením chromatinu“. Molekulární buňka. 18 (1): 123–30. doi:10.1016 / j.molcel.2005.02.031. PMC 2855496. PMID 15808514.
- ^ Fenley AT, Adams DA, Onufriev AV (září 2010). „Stav nabití globulárního histonového jádra řídí stabilitu nukleosomu“. Biofyzikální deník. 99 (5): 1577–85. Bibcode:2010BpJ .... 99.1577F. doi:10.1016 / j.bpj.2010.06.046. PMC 2931741. PMID 20816070.
- ^ Whitehouse I, Flaus A, Cairns BR, White MF, Workman JL, Owen-Hughes T (srpen 1999). "Nukleosomová mobilizace katalyzovaná kvasinkovým komplexem SWI / SNF". Příroda. 400 (6746): 784–7. Bibcode:1999 Natur.400..784W. doi:10.1038/23506. PMID 10466730. S2CID 2841873.
- ^ Kassabov SR, Zhang B, Persinger J, Bartoloměj B (únor 2003). "SWI / SNF rozbalí, sklouzne a znovu zabalí nukleosom". Molekulární buňka. 11 (2): 391–403. doi:10.1016 / S1097-2765 (03) 00039-X. PMID 12620227.
- ^ Bruno M, Flaus A, Stockdale C, Rencurel C, Ferreira H, Owen-Hughes T (prosinec 2003). „Výměna dimeru histonu H2A / H2B pomocí aktivit remodelace chromatinu závislých na ATP“. Molekulární buňka. 12 (6): 1599–606. doi:10.1016 / S1097-2765 (03) 00499-4. PMC 3428624. PMID 14690611.
- ^ Havas K, Flaus A, Phelan M, Kingston R, Wade PA, Lilley DM, Owen-Hughes T (prosinec 2000). "Generování superhelikální torze aktivitami remodelace chromatinu závislými na ATP". Buňka. 103 (7): 1133–42. doi:10.1016 / S0092-8674 (00) 00215-4. PMID 11163188. S2CID 7911590.
- ^ Mizuguchi G, Shen X, Landry J, Wu WH, Sen S, Wu C (leden 2004). „ATP řízená výměna varianty histonu H2AZ katalyzovaná komplexem remodelace chromatinu SWR1“. Věda. 303 (5656): 343–8. Bibcode:2004Sci ... 303..343M. doi:10.1126 / science.1090701. PMID 14645854. S2CID 9881829.
- ^ Métivier R, Penot G, Hübner MR, Reid G, Brand H, Kos M, Gannon F (prosinec 2003). „Estrogen receptor-alfa řídí usměrněný, cyklický a kombinatorický nábor kofaktorů na promotor přirozeného cíle“. Buňka. 115 (6): 751–63. doi:10.1016 / S0092-8674 (03) 00934-6. PMID 14675539. S2CID 145525.
- ^ Nagaich AK, Walker DA, Wolford R, Hager GL (duben 2004). "Rychlá periodická vazba a vytěsnění glukokortikoidního receptoru během remodelace chromatinu". Molekulární buňka. 14 (2): 163–74. doi:10.1016 / S1097-2765 (04) 00178-9. PMID 15099516.
- ^ Teif VB, Vainshtein Y, Caudron-Herger M, Mallm JP, Marth C, Höfer T, Rippe K (listopad 2012). "Pozice nukleosomů v celém genomu během vývoje embryonálních kmenových buněk". Přírodní strukturní a molekulární biologie. 19 (11): 1185–92. doi:10.1038 / nsmb.2419. PMID 23085715. S2CID 34509771.
- ^ Albert I, Mavrich TN, Tomsho LP, Qi J, Zanton SJ, Schuster SC, Pugh BF (březen 2007). "Translační a rotační nastavení nukleosomů H2A.Z napříč genomem Saccharomyces cerevisiae". Příroda. 446 (7135): 572–6. Bibcode:2007 Natur.446..572A. doi:10.1038 / nature05632. PMID 17392789. S2CID 4416890.
- ^ Li B, Carey M, Workman JL (únor 2007). "Úloha chromatinu během transkripce". Buňka. 128 (4): 707–19. doi:10.1016 / j.cell.2007.01.015. PMID 17320508. S2CID 1773333.
- ^ Whitehouse I, Rando OJ, Delrow J, Tsukiyama T (prosinec 2007). "Remodelace chromatinu na promotorech potlačuje transkripci antisense". Příroda. 450 (7172): 1031–5. Bibcode:2007 Natur.450.1031W. doi:10.1038 / nature06391. PMID 18075583. S2CID 4305576.
- ^ Lee W, Tillo D, Bray N, Morse RH, Davis RW, Hughes TR, Nislow C (October 2007). "A high-resolution atlas of nucleosome occupancy in yeast". Genetika přírody. 39 (10): 1235–44. doi:10.1038/ng2117. PMID 17873876. S2CID 12816925.
- ^ Eaton ML, Galani K, Kang S, Bell SP, MacAlpine DM (April 2010). "Conserved nucleosome positioning defines replication origins". Geny a vývoj. 24 (8): 748–53. doi:10.1101/gad.1913210. PMC 2854390. PMID 20351051.
- ^ Shivaswamy S, Bhinge A, Zhao Y, Jones S, Hirst M, Iyer VR (March 2008). "Dynamic remodeling of individual nucleosomes across a eukaryotic genome in response to transcriptional perturbation". PLOS Biology. 6 (3): e65. doi:10.1371/journal.pbio.0060065. PMC 2267817. PMID 18351804.
- ^ Murugesapillai D, McCauley MJ, Huo R, Nelson Holte MH, Stepanyants A, Maher LJ, Israeloff NE, Williams MC (srpen 2014). „Přemostění a smyčka DNA pomocí HMO1 poskytuje mechanismus pro stabilizaci chromatinu bez nukleosomů“. Výzkum nukleových kyselin. 42 (14): 8996–9004. doi:10.1093 / nar / gku635. PMC 4132745. PMID 25063301.
- ^ Murugesapillai D, McCauley MJ, Maher LJ, Williams MC (únor 2017). „Jednomolekulární studie architektonických proteinů ohýbajících DNA skupiny B s vysokou mobilitou“. Biofyzikální recenze. 9 (1): 17–40. doi:10.1007 / s12551-016-0236-4. PMC 5331113. PMID 28303166.
- ^ Hayes JJ, Lee KM (May 1997). "In vitro reconstitution and analysis of mononucleosomes containing defined DNAs and proteins". Metody. 12 (1): 2–9. doi:10.1006/meth.1997.0441. PMID 9169189.
- ^ Dyer PN, Edayathumangalam RS, White CL, Bao Y, Chakravarthy S, Muthurajan UM, Luger K (2004). "Reconstitution of nucleosome core particles from recombinant histones and DNA". Metody v enzymologii. 375: 23–44. doi:10.1016/s0076-6879(03)75002-2. ISBN 9780121827793. PMID 14870657.
- ^ Yenidunya A, Davey C, Clark D, Felsenfeld G, Allan J (April 1994). "Nucleosome positioning on chicken and human globin gene promoters in vitro. Novel mapping techniques". Journal of Molecular Biology. 237 (4): 401–14. doi:10.1006/jmbi.1994.1243. PMID 8151701.
- ^ Frouws TD, Barth PD, Richmond TJ (January 2018). "Site-Specific Disulfide Crosslinked Nucleosomes with Enhanced Stability". Journal of Molecular Biology. 430 (1): 45–57. doi:10.1016/j.jmb.2017.10.029. PMC 5757783. PMID 29113904.
- ^ Ferentz AE, Verdine GL (1994). "The Convertible Nucleoside Approach: Structural Engineering of Nucleic Acids by Disulfide Cross-Linking". In Eckstein F, Lilley DM (eds.). Nucleic Acids and Molecular Biology. Nucleic Acids and Molecular Biology. 8. pp. 14–40. doi:10.1007/978-3-642-78666-2_2. ISBN 978-3-642-78668-6.
- ^ Yamasu K, Senshu T (1990). "Conservative Segregation of Tetrametric Units of H3 and H4 Histones during Nucleosome Replication". Journal of Biochemistry. 170 (1): 15–20. doi:10.1093/oxfordjournals.jbchem.a122999. PMID 2332416.
- ^ Kaufman PD, Kobayashi R, Kessler N, Stillman B (June 1995). "The p150 and p60 subunits of chromatin assembly factor I: a molecular link between newly synthesized histones and DNA replication". Buňka. 81 (7): 1105–14. doi:10.1016/S0092-8674(05)80015-7. PMID 7600578. S2CID 13502921.
- ^ Tyler JK, Adams CR, Chen SR, Kobayashi R, Kamakaka RT, Kadonaga JT (December 1999). "The RCAF complex mediates chromatin assembly during DNA replication and repair". Příroda. 402 (6761): 555–60. Bibcode:1999Natur.402..555T. doi:10.1038/990147. PMID 10591219. S2CID 205097512.
- ^ Benson LJ, Gu Y, Yakovleva T, Tong K, Barrows C, Strack CL, Cook RG, Mizzen CA, Annunziato AT (April 2006). "Modifications of H3 and H4 during chromatin replication, nucleosome assembly, and histone exchange". The Journal of Biological Chemistry. 281 (14): 9287–96. doi:10.1074/jbc.M512956200. PMID 16464854.
- ^ Louters L, Chalkley R (June 1985). "Exchange of histones H1, H2A, and H2B in vivo". Biochemie. 24 (13): 3080–5. doi:10.1021/bi00334a002. PMID 4027229.
- ^ Park YJ, Chodaparambil JV, Bao Y, McBryant SJ, Luger K (January 2005). "Nucleosome assembly protein 1 exchanges histone H2A-H2B dimers and assists nucleosome sliding". The Journal of Biological Chemistry. 280 (3): 1817–25. doi:10.1074/jbc.M411347200. PMID 15516689.
- ^ Vincent JA, Kwong TJ, Tsukiyama T (May 2008). "ATP-dependent chromatin remodeling shapes the DNA replication landscape". Přírodní strukturní a molekulární biologie. 15 (5): 477–84. doi:10.1038/nsmb.1419. PMC 2678716. PMID 18408730.
- ^ Yadav T, Whitehouse I (April 2016). "Replication-Coupled Nucleosome Assembly and Positioning by ATP-Dependent Chromatin-Remodeling Enzymes". Zprávy buněk. 15 (4): 715–723. doi:10.1016/J.CELREP.2016.03.059. PMC 5063657. PMID 27149855.
externí odkazy
- MBInfo - What are nucleosomes
- Nucleosomes on the Richmond Lab website
- Proteopedia Nukleosomy
- Nucleosome at the PDB
- Dynamic Remodeling of Individual Nucleosomes Across a Eukaryotic Genome in Response to Transcriptional Perturbation
- Nucleosome positioning data and tools online (annotated list, constantly updated)
- Histone protein structure
- HistoneDB 2.0 - Database of histones and variants na NCBI