Katalytický intron skupiny I - Group I catalytic intron
| Katalytický intron skupiny I | |
|---|---|
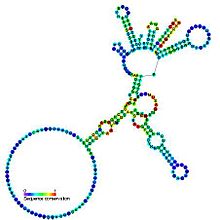 Předpovězeno sekundární struktura a zachování sekvence skupiny I katalytického intronu | |
| Identifikátory | |
| Symbol | Intron_gpI |
| Rfam | RF00028 |
| Další údaje | |
| RNA typ | Intron |
| Domény | Eukaryota; Bakterie; Viry |
| JÍT | GO termín musí začínat GO: |
| TAK | SO: 0000587 |
| PDB struktur | PDBe |
Introny skupiny I jsou velké samosestřihování ribozymy. Ony katalyzovat jejich vlastní excize z mRNA, tRNA a rRNA prekurzory v široké škále organismů.[1][2][3] Jádro sekundární struktura sestává z devíti spárovaných oblastí (P1-P9).[4] Skládají se v podstatě na dvě domén - doména P4-P6 (vytvořená skládáním šroubovice P5, P4, P6 a P6a) a doména P3-P9 (vytvořená ze šroubovice P8, P3, P7 a P9).[2] Značka sekundární struktury pro tuto rodinu představuje pouze toto konzervované jádro. Skupina I introny často mají dlouhou otevřené čtecí rámce vloženo smyčkové regiony.
Katalýza
Sestřih z introny skupiny I. je zpracován dvěma sekvenčními transesterifikace reakce.[3] The exogenní guanosin nebo guanosin nukleotid (exoG) nejprve zakotví na aktivním vazebném místě G umístěném v P7 a jeho 3'-OH je zarovnán tak, aby napadl fosfodiesterová vazba na 5 'spojovacím místě umístěném v P1, což vede k volné 3'-OH skupině na proti směru toku exon a exoG je připojen k 5 'konci intronu. Potom terminál G (omega G) intronu zamění exoG a obsadí G-vazebné místo pro organizaci druhé reakce přenosu esteru: 3'-OH skupina upstream exonu v P1 je zarovnána tak, aby zaútočila na 3 'spoj místo v P10, vedoucí k ligace sousedních upstream a downstream exonů a uvolnění katalytického intronu.
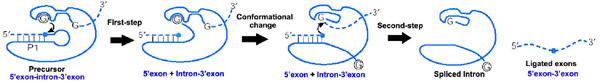
Mechanismus dvou kovů a iontů pozorovaný v proteinu polymerázy a fosfatázy bylo navrženo k použití ve skupině I a introny skupiny II zpracovat reakce přenosu fosforylu,[5] což bylo jednoznačně prokázáno strukturou Azoarcus skupina I intron v roce 2006.[6]


Skládání intronů
Od počátku 90. let začali vědci studovat, jak skupina I Intron dosahuje své přirozené struktury in vitro a některé mechanismy RNA skládací byly dosud oceněny.[10] Je dohodnuto, že terciární struktura je složen po vytvoření sekundární struktury. Během skládání jsou molekuly RNA rychle osídleny do různých skládacích meziproduktů, meziprodukty obsahující nativní interakce jsou dále skládány do nativní struktury rychlou cestou skládání, zatímco ty obsahující nepůvodní interakce jsou zachyceny v metastabilní nebo stabilní nepůvodní konformace a proces převodu na nativní strukturu probíhá velmi pomalu. Je evidentní, že introny skupiny I lišící se v sadě periferních prvků vykazují různé potenciály při vstupu do rychlé skládací dráhy. Mezitím je kooperativní montáž terciární struktury důležitá pro skládání nativní struktury. Složení intronů skupiny I in vitro se nicméně setkává s oběma termodynamické a kinetický výzvy. Několik proteinů vázajících RNA a chaperony Bylo prokázáno, že podporují skládání intronů skupiny I in vitro a v bakteriích stabilizací nativních meziproduktů a destabilizací nepůvodních struktur.
Distribuce, fylogeneze a mobilita
Introny skupiny I jsou distribuovány v bakteriích, nižší eukaryoty a vyšší rostliny. Zdá se však, že jejich výskyt v bakteriích je sporadičtější než v nízkoeukaryotických buňkách a převládají u vyšších rostlin. The geny ta skupina, kterou Iintronové přerušují, se významně liší: přerušují rRNA, mRNA a tRNA geny v bakteriálních genomech, stejně jako v mitochondriální a chloroplast genomy nižších eukaryot, ale napadají pouze rRNA geny v jaderný genom nižších eukaryot. U vyšších rostlin se zdá, že tyto introny jsou omezeny na několik genů pro tRNA a mRNA chloroplastů a mitochondrií.
Introny skupiny I se také nacházejí vložené do genů široké škály bakteriofágy z Grampozitivní bakterie.[11] Nicméně, jejich distribuce ve fágu Gramnegativní bakterie je omezen hlavně na T4, T-sudé a T7-like bakteriofágy.[11][12][13][14]
Teorie intron-early a intron-late našly důkazy při vysvětlování původu intronů skupiny I. Některé introny skupiny I kódují naváděcí endonukleáza (HEG), který katalyzuje intronovou mobilitu. Navrhuje se, aby HEG přesunuly intron z jednoho místa do druhého, z jednoho organismu do druhého, a tak odpovídaly za široké šíření sobeckých intronů skupiny I. Dosud nebyla identifikována žádná biologická role pro introny skupiny I, kromě toho, že se spojily z předchůdce, aby se zabránilo smrti hostitele, kterým žijí. Bylo zjištěno, že malý počet intronů skupiny I kóduje třídu proteinů nazývaných maturázy, které usnadňují spojování intronů.
Viz také
- Intron
- Skupina I Intron sekvence a struktura databáze
- Spojte web
- Jaderné introny
- Intron skupiny II
- Intron skupiny III
- Twintron
- LtrA
- Cyklický riboswitch di-GMP-II kde je příklad riboswitche působícího společně se intronem skupiny I k regulaci exprese genu
Reference
- ^ Nielsen H, Johansen SD (2009). „Introny skupiny I: Pohyb v nových směrech“. RNA Biol. 6 (4): 375–83. doi:10,4161 / rna.6.4.9334. PMID 19667762. Citováno 2010-07-15.
- ^ A b Cate JH, Gooding AR, Podell E a kol. (Září 1996). "Krystalová struktura ribozymové domény skupiny I: principy balení RNA". Věda. 273 (5282): 1678–85. doi:10.1126 / science.273.5282.1678. PMID 8781224.
- ^ A b Čech TR (1990). „Sesazování intronů skupiny I“. Annu. Biochem. 59: 543–68. doi:10.1146 / annurev.bi.59.070190.002551. PMID 2197983.
- ^ Woodson SA (červen 2005). "Struktura a montáž intronů skupiny I". Curr. Opin. Struct. Biol. 15 (3): 324–30. doi:10.1016 / j.sbi.2005.05.007. PMID 15922592.
- ^ Steitz, TA; Steitz JA (1993). „Obecný mechanismus dvou kovů a iontů pro katalytickou RNA“. Proc Natl Acad Sci USA. 90 (14): 6498–6502. doi:10.1073 / pnas.90.14.6498. PMC 46959. PMID 8341661.
- ^ Stahley, MR; Strobel SA (2006). „RNA sestřih: krystalová struktura intronu skupiny I odhaluje základ výběru místa sestřihu a katalýzy kovovými ionty“. Curr Opin Struct Biol. 16 (3): 319–326. doi:10.1016 / j.sbi.2006.04.005. PMID 16697179.
- ^ Golden BL, Gooding AR, Podell ER, Cech TR (1998). „Preorganizované aktivní místo v krystalové struktuře ribozymu Tetrahymena“. Věda. 282 (5387): 259–64. doi:10.1126 / science.282.5387.259. PMID 9841391.
- ^ Golden BL, Kim H, Chase E (2005). "Krystalová struktura komplexu ribozymu a produktu fága Twort skupiny I". Nat Struct Mol Biol. 12 (1): 82–9. doi:10.1038 / nsmb868. PMID 15580277.
- ^ Guo F, Gooding AR, Cech TR (2004). "Struktura Tetrahymena ribozymu: základní trojitý sendvič a kovový ion v aktivním místě". Mol Cell. 16 (3): 351–62. doi:10.1016 / j.molcel.2004.10.003. PMID 15525509.
- ^ Brion P, Westhof E (1997). "Hierarchie a dynamika skládání RNA". Annu Rev Biophys Biomol Struct. 26: 113–37. doi:10.1146 / annurev.biophys.26.1.113. PMID 9241415.
- ^ A b Edgell DR, Belfort M, Shub DA (říjen 2000). „Překážky intronové promiskuity u bakterií“. J. Bacteriol. 182 (19): 5281–9. doi:10.1128 / jb.182.19.5281-5289.2000. PMC 110968. PMID 10986228.
- ^ Sandegren L, Sjöberg BM (květen 2004). „Distribuce, sekvenční homologie a navádění intronů skupiny I mezi bakteriofágy podobné T: důkaz nedávného přenosu starých intronů“. J. Biol. Chem. 279 (21): 22218–27. doi:10,1074 / jbc.M400929200. PMID 15026408.
- ^ Bonocora RP, Shub DA (prosinec 2004). „Samosestřihovací skupina I intron v DNA polymerázových genech bakteriofágů podobných T7“. J. Bacteriol. 186 (23): 8153–5. doi:10.1128 / JB.186.23.8153-8155.2004. PMC 529087. PMID 15547290.
- ^ Lee CN, Lin JW, Weng SF, Tseng YH (prosinec 2009). „Genomická charakterizace intronového fágu podobného T7 phiL7 z Xanthomonas campestris“. Appl. Environ. Microbiol. 75 (24): 7828–37. doi:10.1128 / AEM.01214-09. PMC 2794104. PMID 19854925.
Další čtení
- Chauhan, S; Caliskan G; Briber RM; Perez-Salas U; Rangan P; Thirumalai D; Woodson SA (2005). „Terciární interakce RNA zprostředkovávají nativní kolaps ribozymu bakteriální skupiny I.“. J Mol Biol. 353 (5): 1199–1209. doi:10.1016 / j.jmb.2005.09.015. PMID 16214167.
- Haugen, P; Simon DM; Bhattacharya D (2005). "Přirozená historie intronů skupiny I". Trendy v genetice. 21 (2): 111–119. doi:10.1016 / j.tig.2004.12.007. PMID 15661357.
- Rangan, P; Masquida, B; Westhof E; Woodson SA (2003). "Sestavení jádrových šroubovic a rychlé terciární skládání malé bakteriální skupiny I ribozymu". Proc Natl Acad Sci USA. 100 (4): 1574–1579. doi:10.1073 / pnas.0337743100. PMC 149874. PMID 12574513.
- Schroeder, R; Barta A; Semrad K (2004). "Strategie pro skládání a montáž RNA". Nat Rev Mol Cell Biol. 5 (11): 908–919. doi:10.1038 / nrm1497. PMID 15520810.
- Thirumalai, D; Lee N; Woodson SA; Klimov D (2001). "Rané události ve skládání RNA". Annu Rev Phys Chem. 52: 751–762. doi:10,1146 / annurev.physchem. 52.1.751. PMID 11326079.
- Treiber, DK; Williamson JR (1999). "Vystavení kinetických pasti při skládání RNA". Curr Opin Struct Biol. 9 (3): 339–345. doi:10.1016 / S0959-440X (99) 80045-1. PMID 10361090.
- Xiao, M; Leibowitz MJ; Zhang Y (2003). „Společné skládání ribozymu Candida do katalyticky aktivní struktury za rychlým zhutněním RNA“. Nucleic Acids Res. 31 (14): 3901–3908. doi:10.1093 / nar / gkg455. PMC 165970. PMID 12853605.
