Heterogenita nádoru - Tumour heterogeneity
Heterogenita nádoru popisuje pozorování odlišné nádorové buňky může ukázat odlišně morfologické a fenotypový profily, včetně buněčné morfologie, genové exprese, metabolismu, motility, proliferace a metastatického potenciálu.[1] K tomuto jevu dochází jak mezi nádory (inter-tumorová heterogenita), tak uvnitř tumorů (intra-tumor heterogenita). Minimální úroveň intra-nádorové heterogenity je jednoduchým důsledkem nedokonalosti replikace DNA: kdykoli se buňka (normální nebo rakovinová) rozdělí, pár mutace jsou získány[2]—Vedoucí k rozmanité populaci rakovinných buněk.[3] The heterogenita rakovinných buněk přináší významné výzvy při navrhování účinných strategií léčby. Výzkum porozumění a charakterizace heterogenity však může umožnit lepší pochopení příčin a progrese nemoci. To má zase potenciál řídit tvorbu rafinovanějších léčebných strategií, které obsahují znalosti o heterogenitě, aby se dosáhlo vyšší účinnosti.[4]
Heterogenita nádoru byla pozorována u leukémie,[5] prsa,[6] prostata,[7][8][9] dvojtečka,[10][11][12] mozek,[13] jícen,[14] hlava a krk,[15] měchýř[16] a gynekologické karcinomy,[17] liposarkom,[18] a mnohočetný myelom.[19]
Modely heterogenity
K vysvětlení heterogenity nádorových buněk se používají dva modely. Tohle jsou rakovinová kmenová buňka model a klonální evoluce Modelka. Modely se navzájem nevylučují a předpokládá se, že oba přispívají k různorodosti heterogenity v různých typech nádorů.[20]
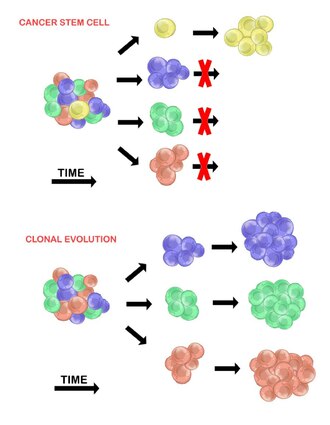
Rakovinové kmenové buňky
Model rakovinných kmenových buněk tvrdí, že v populaci nádorových buněk existuje pouze malá podmnožina buněk tumourigenic (schopný tvořit nádory). Tyto buňky se nazývají rakovinové kmenové buňky (CSC), a jsou poznamenány schopností jak sebeobnovy, tak odlišit se od jiných než tumourigenních potomků. Model CSC předpokládá, že heterogenita pozorovaná mezi nádorovými buňkami je výsledkem rozdílů v kmenových buňkách, ze kterých pocházejí. Variabilita kmenových buněk je často způsobena epigenetický změny, ale mohou být také výsledkem klonálního vývoje populace CSC, kde je to výhodné genetické mutace se mohou hromadit v CSC a jejich potomcích (viz níže).[20]
Důkaz modelu rakovinných kmenových buněk byl prokázán u mnoha typů nádorů včetně leukémie,[21][22] glioblastom,[23] rakovina prsu,[24] a rakovina prostaty.[25]
O existenci CSC se však stále diskutuje. Jedním z důvodů je to, že je obtížné reprodukovat markery pro CSC napříč několika nádory. Dále se využívají metody pro stanovení tumourigenního potenciálu xenograft modely. Tyto metody trpí inherentními omezeními, jako je potřeba kontrolovat imunitní odpověď u transplantovaného zvířete a významný rozdíl v podmínkách prostředí od místa primárního nádoru po místo xenograftu (např. absence požadovaných exogenních molekul nebo kofaktorů).[26] To způsobilo určité pochybnosti o přesnosti výsledků CSC a závěrech o tom, které buňky mají tumorogenní potenciál.
Klonální evoluce
Model klonální evoluce poprvé navrhl v roce 1976 Peter Nowell.[27] V tomto modelu nádory vznikají z jedné mutované buňky a akumulují další mutace, jak postupuje. Tyto změny vedou k dalším subpopulacím a každá z těchto subpopulací má schopnost se dále dělit a mutovat. Tato heterogenita může vést k subklonům, které mají evoluční výhoda nad ostatními v rámci prostředí nádoru a tyto subklony se mohou v průběhu času stát dominantním v nádoru.[28][29] Když byl tento model navržen, umožňoval porozumět růstu nádoru, selhání léčby a agresi nádoru, ke které dochází během přirozeného procesu tvorby nádoru.[28]
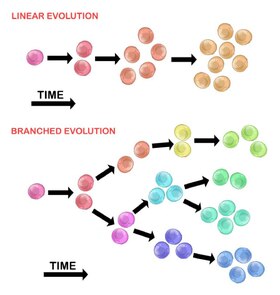
K vývoji počáteční nádorové buňky může dojít dvěma způsoby:
Lineární expanze
Sekvenčně seřazené mutace se hromadí v genech řidičů, geny potlačující nádory, a Oprava DNA enzymy, což vede k klonální expanzi nádorových buněk. Je méně pravděpodobné, že lineární expanze bude odrážet koncový bod maligního nádoru[30] protože akumulace mutací je u heterogenních nádorů stochastická.
Rozvětvená expanze
Expanze do několika subklonálních populací probíhá prostřednictvím mechanismu rozdělení.[28] Tato metoda je více spojena s heterogenitou nádoru než s lineární expanzí. Získávání mutací je náhodné v důsledku zvýšení genomická nestabilita s každou další generací. Dlouhodobá mutační akumulace může poskytnout a selektivní výhoda během určitých stádií progrese nádoru. The mikroprostředí nádoru může také přispívat k expanzi nádoru, protože je schopen měnit selektivní tlaky, kterým jsou nádorové buňky vystaveny.[30]
Druhy a příčiny heterogenity
Mezi nádorovými buňkami bylo pozorováno více typů heterogenity vyplývajících z genetické i negenetické variability.[31]
Genetická heterogenita
Genetická heterogenita je společným rysem nádorových genomů a může vzniknout z různých zdrojů. Některé druhy rakoviny jsou zahájeny, když exogenní faktory zavádějí mutace, jako je ultrafialové záření (rakovina kůže) a tabák (rakovina plic). Častějším zdrojem je genomová nestabilita, která často vzniká při narušení klíčových regulačních cest v buňkách. Některé příklady zahrnují postižené Oprava DNA mechanismy, které mohou vést ke zvýšeným chybám replikace a defektům v mitóza stroje, které umožňují rozsáhlý zisk nebo ztrátu celku chromozomy.[32] Dále je možné, že genetická variabilita bude dále zvýšena některými terapiemi rakoviny (např. léčba temozolomid a další chemoterapie léky).[33][34]
Heterogenita mutačního tumoru se týká variací frekvence mutací v různých genech a vzorcích a lze ji prozkoumat MutSig. Etiologie mutačních procesů se může značně lišit mezi vzorky nádorů stejného nebo různých typů rakoviny a může se projevovat v různých kontextově závislých mutačních profilech. Může to být prozkoumáno COSMIC mutační podpisy nebo MutaGene.
Jiná heterogenita
Nádorové buňky mohou také vykazovat heterogenitu mezi svými profily exprese. To je často způsobeno podkladem epigenetický Změny.[31]Byly detekovány variace signatur exprese v různých oblastech vzorků tumoru u jedince. Vědci prokázali, že konvergentní mutace ovlivňují H3K36 methyltransferáza SETD2 a histon H3K4 demethyláza KDM5C vznikly v prostorově oddělených částech nádoru. Podobně, MTOR, gen kódující buněčnou regulaci kináza Ukázalo se, že je konstitutivně aktivní, čímž zvyšuje S6 fosforylace. To je aktivní fosforylace může sloužit jako biomarker u karcinomu čirých buněk.[30]
Mechanochemické heterogenita je charakteristickým znakem života eukaryotický buňky. Má dopad na epigenetický genová regulace. Heterogenní dynamické mechanochemické procesy regulují vzájemné vztahy ve skupině buněčných povrchů přilnavost.[35] Vývoj a šíření nádoru je doprovázen změnami v heterogenitě chaotická dynamika mechanochemického interakčního procesu ve skupinových buňkách, včetně buněk v nádoru, a je hierarchický pro hostitele pacientů s rakovinou.[36] Biologické jevy mechanochemické heterogenity mohou být použity pro diferenciální rakovina žaludku diagnostika proti pacientům s zánět z žaludeční sliznice[37] a pro zvýšení antimetastatické aktivity dendritické buňky na základě vakcíny když je mechanicky heterogenizován mikročástice pro jejich načtení se použije nádorových buněk.[38] Existuje také možný metodický přístup založený na simultánních ultrazvukových zobrazovacích diagnostických technikách a terapii, pokud jde o mechanochemický účinek na nanobubliny konglomeráty s léky v nádoru.
Mikroprostředí nádoru
Heterogenitu mezi nádorovými buňkami lze dále zvýšit kvůli heterogenitě v mikroprostředí nádoru. Regionální rozdíly v nádoru (např. dostupnost kyslíku) působí na nádorové buňky různé selektivní tlaky, což vede k širšímu spektru dominantních subklonů v různých prostorových oblastech nádoru. Vliv mikroprostředí na klonální dominanci je také pravděpodobným důvodem heterogenity mezi primárním a metastazující tumory pozorované u mnoha pacientů, stejně jako inter-tumorová heterogenita pozorovaná u pacientů se stejným typem tumoru.[39]
Důsledky a výzvy
Odolnost vůči léčbě
Heterogenní nádory mohou vykazovat různé citlivosti na cytotoxické léky mezi různými klonálními populacemi. To se připisuje klonálním interakcím, které mohou inhibovat nebo měnit terapeutickou účinnost, což představuje výzvu pro úspěšné terapie u heterogenních nádorů (a jejich heterogenních metastáz).[1]
Podávání léků v heterogenních nádorech málokdy zabije všechny nádorové buňky. Počáteční populace heterogenních nádorů může úzké místo tak, že přežije jen málo buněk rezistentních na léky (pokud existují). To umožňuje populacím rezistentních nádorů replikovat a růst nového nádoru prostřednictvím mechanismu evolučního rozvětvení (viz výše). Výsledný repopulovaný nádor je heterogenní a rezistentní k použité počáteční lékové terapii. Repopulovaný nádor se může také vrátit agresivnějším způsobem.
Správa cytotoxické léky často vede k počátečnímu zmenšení nádoru. To představuje destrukci počátečních neresistentních subklonálních populací v heterogenním nádoru, přičemž zůstávají pouze rezistentní klony. Tyto rezistentní klony nyní obsahují selektivní výhodu a mohou se replikovat za účelem repopulace nádoru. K replikaci pravděpodobně dojde prostřednictvím větvící se evoluce, což přispěje k heterogenitě nádoru. Repopulovaný nádor se může zdát agresivnější. To je přičítáno selektivní výhodě nádorových buněk rezistentní na léčivo.

Objev biomarkerů
Kvůli genetickým rozdílům uvnitř a mezi nádory biomarkery které mohou předvídat odpověď na léčbu nebo prognózu, nemusí být široce použitelné. Bylo však navrženo, že samotná úroveň heterogenity může být použita jako biomarker, protože u heterogennějších nádorů je pravděpodobnější, že budou obsahovat subklony rezistentní na léčbu.[31] Další výzkum týkající se vývoje biomarkerů, které zohledňují heterogenitu, stále probíhá.
Modelové systémy
Současné modelové systémy obvykle postrádají heterogenitu pozorovanou u lidských rakovin.[40] Abychom mohli přesně studovat heterogenitu nádoru, musíme vyvinout přesnější preklinické modely. Jeden takový model, xenograft tumoru odvozený od pacienta, prokázal vynikající užitečnost při zachování heterogenity nádoru a zároveň umožňuje podrobné studium řidičů klonální kondice.[41] Ani tento model však nedokáže zachytit úplnou složitost rakoviny.
Současné strategie
Zatímco problém identifikace, charakterizace a léčby heterogenity nádorů stále probíhá v aktivním výzkumu, byly navrženy některé účinné strategie, včetně experimentálních i výpočetních řešení.
Experimentální
- Cílený přístup: analyzovat konkrétní genetiku místo nebo soubor loci. K tomu může dojít detekcí alelické nerovnováhy (DNA tumoru je srovnávána s DNA zárodečné linie), amplifikací chromozomálních oblastí (RYBA ) a / nebo sekvenování specifických genů. Tato metoda se používá ke sledování vývoje konkrétní sledované mutace nebo k potvrzení mutace, kterou mohou vědci předpokládat v nádoru.[1]
- Výhoda: Umožňuje analýzu konkrétních genů (tj. řidičské geny, nádorové supresory). Proces je jednoduchý s přímou interpretací výsledků. RYBA a imunofluorescence umožňuje zaměření na podtypy nádorových buněk.[1]
- Nevýhoda: Omezené analýze budou chybět další důležité mutace a vzorce klonální expanze. Alelickou nerovnováhu může být obtížné ověřit pomocí mikrosatelitních markerů, což vyžaduje ověření nezávislou technikou (tj. RYBA). RYBA vyžaduje velké množství buněk a je náročné na pracovní sílu.[1]
- Celý genomový přístup: analýza celého genomu ve vzorcích nádorů. To lze provést prostřednictvím karyotypizace nebo komparativní genomová hybridizace (CGH) k detekci chromozomálních abnormalit. Sekvenování nádorových biopsií je stále běžnější.[1]
- Výhoda: Při identifikaci variant se nespoléhá na předchozí znalosti. karyotypizace identifikuje oblasti velkých chromozomálních abnormalit. CGH poskytuje objektivní pokrytí a umožňuje detekovat alelickou nerovnováhu malého rozsahu (Pole SNP). Sekvenování identifikuje všechny varianty, které přispívají k heterogenitě nádoru.[1]
- Nevýhoda: Je obtížné určit funkční dopad variant (tj. neutrální nebo patogenní). Omezené rozlišení. Karyotypizace kultivovaných buněk může být ovlivněno směrem k preferenčnímu růstu vybraných subpopulací nádorových buněk. Omezené rozlišení v obou metodách.[1] Přístup celého genomu může generovat velké množství dat a může být obtížné jej interpretovat.
- Strategie vzorkování více regionů: obecně vyžaduje několik pooperačních vzorků nádoru ze samostatných oblastí mikrodisekovaného nádoru. Je důležité vyhnout se kontaminaci nezhoubných buněk, abyste zajistili přesnou reprezentaci genové exprese a genetického složení pouze v nádorových buňkách. Analýza nádorové DNA v prostorově oddělených oblastech umožňuje konstrukci trojrozměrného evolučního modelu heterogenity nádoru.[1] Multiregionální vzorkování se často používá v kombinaci s přístupem celého genomu k vytvoření tohoto modelu rozšíření 3D heterogenity.
- Podélný odběr vzorků: prostřednictvím progrese nádoru nebo progrese léčby bylo v některých případech využito získávání vzorků nádoru během několika časových bodů. To bylo navrženo jako spolehlivá metoda pro sledování klonální evoluce.[34][42][43] Tato technika se však v praxi ukazuje jako náročná, protože vyžaduje periodickou invazivní biopsii. Nový výzkum využití cirkulující bezbuněčné nádorové DNA v krvi může poskytnout neinvazivní způsob identifikace biomarkerů během léčby.[44] Podélný odběr vzorků použitý v kombinaci s přístupem celého genomu umožní identifikaci akumulovaných mutací nádorových buněk v čase. To může zase identifikovat klíčové mutace řidiče (viditelné v počátečních vzorcích nádoru).
- Adaptivní terapie lze použít k prevenci dalšího růstu nádoru úpravou dávky léčiva a načasování podávání léčiva na základě reakce tumoru. Předpokládá se, že tato strategie brání rezistentním variantám dominovat v nádoru. Je však zapotřebí dalšího výzkumu ohledně jeho použitelnosti.[45]
Sekvenování
- Hromadné sekvenování nádorů lze použít, kde je DNA extrahována ze směsi nádorových buněk a analyzována najednou. Přítomnost heterogenních populací nádorů (subklony) přináší další výzvy, jako například:
- Neschopnost detekovat mutace ve vzácných subklonech. Protože se tyto mutace vyskytnou s nízkou frekvencí ve sdruženém vzorku, mohou být nerozeznatelné od šumu pozadí. Aktivně se však vyvíjí mnoho volajících variant, které jsou speciálně navrženy pro údaje o rakovině a jejichž cílem je identifikovat vzácné varianty přítomné v menších subklonálních populacích.[46][47][48][49] Tito typicky využívají spárovanou normální DNA jako prostředek k rozlišení true somatická variace z variace zárodečné linie a chyba sekvenování pozadí.
- Neschopnost určit, které subklony obsahují každou mutaci. Vzhledem k tomu, že jsou data shromážděna, není jasné, které mutace se vyskytují společně a ze kterých populací pocházejí. Vyvíjejí se nové nástroje, které se pokouší vyřešit klonální strukturu pomocí frekvence alel pro pozorované mutace.[50]
- Sekvenování jednotlivých buněk je nová technika, která je cenná pro hodnocení heterogenity nádorů, protože může charakterizovat jednotlivé nádorové buňky. To znamená, že celý mutační profil několika odlišných buněk lze určit bez dvojznačnosti. Zatímco se současnou technologií je obtížné vyhodnotit dostatečně velký počet jednotlivých buněk k získání statistické síly, jednobuněčná data o nádoru mají několik výhod, včetně:
- Schopnost postavit a fylogenetický strom ukazující vývoj populací nádorů. Pomocí sekvencí celého genomu nebo SNP Na základě pseudosekvencí z jednotlivých buněk lze odhadnout vývoj subklonu. To umožňuje identifikaci populací, které přetrvávaly v průběhu času, a může zúžit seznam mutací, které potenciálně udělují specifickou subklonu růstovou výhodu nebo rezistenci vůči léčbě.[51] Algoritmy pro odvození fylogeneze nádoru z jednobuněčných dat sekvenování DNA zahrnují SCITE,[52] OncoNEM,[53] SiFit,[54] SiCloneFit,[55] PhISCS,[56] a PhISCS-BnB.[57]
- Sekvenování sekcí lze provést na více částech jednoho solidního nádoru a odchylky ve frekvencích mutací napříč řezy lze analyzovat, aby se odvodila klonální struktura. Mezi výhody tohoto přístupu oproti jednoduchému sekvenování patří větší statistická síla a dostupnost přesnějších informací o prostorovém umístění vzorků. Ten lze použít k odvození frekvence klonů v sekcích a poskytnutí vhledu o tom, jak se nádor vyvíjí ve vesmíru. K odvození genotypů klonů a fylogenetických stromů, které modelují vývoj nádoru v čase, bylo vyvinuto několik výpočetních metod[58][59][60] včetně Clomial,[61] klonovat HD,[62] PhyloWGS,[63] PyClone,[64] Cloe,[65] phyC,[66] Baldachýn,[67] TargetClone, ddClone,[68] PASTRI,[69] GLClone,[70] Vlastnost,[71] WSCUnmix,[72] B-SCITE.,[73] ThetA,[74] SIFA,[75] Sclust,[76] SeqClone,[77] KALDER,[78] BAMSE,[79], Meltos[80], SubMARine[81]a RNDCLONE.[82]
Viz také
Reference
- ^ A b C d E F G h i Marusyk, A; Polyak, K (2010). „Heterogenita nádorů: příčiny a důsledky“. Biochimica et Biophysica Acta (BBA) - Recenze na rakovinu. 1805 (1): 105–117. doi:10.1016 / j.bbcan.2009.11.002. PMC 2814927. PMID 19931353.
- ^ Vogelstein, Bert; Papadopoulos, N .; Velculescu, V.E .; Zhou, S .; Diaz, L.A .; Kinzler, K.W. (2013). „Krajiny genomu rakoviny“. Věda. 373 (6127): 1546–1556. Bibcode:2013Sci ... 339.1546V. doi:10.1126 / science.1235122. PMC 3749880. PMID 23539594.
- ^ Heppner, G.A. (1984). „Heterogenita nádorů“. Výzkum rakoviny. 44 (6): 2259–2265. PMID 6372991.
- ^ Reiter, Johannes G; Makohon-Moore, Alvin P; Gerold, Jeffrey M; Heyde, Alexander; Attiyeh, Marc A; Kohutek, Zachary A; Tokheim, Collin J; Brown, Alexia; DeBlasio, Rayne; Niyazov, Juliana; Zucker, Amanda; Karchin, Rachel; Kinzler, Kenneth W; Iacobuzio-Donahue, Christine A; Vogelstein, Bert; Nowak, Martin A (2018). „Minimální heterogenita funkčního řidičského genu mezi neléčenými metastázami“. Věda. 361 (6406): 1033–1037. doi:10.1126 / science.aat7171. PMC 6329287. PMID 30190408.
- ^ Campbell, P. J .; Pleasance, E. D .; Stephens, P. J .; Dicks, E; Rance, R; Sbohem, já; Follows, G. A .; Green, A. R .; Futreal, P. A .; Stratton, M. R. (2008). „Subklonální fylogenetické struktury u rakoviny odhaleny ultrahlubokým sekvenováním“. Sborník Národní akademie věd. 105 (35): 13081–13086. Bibcode:2008PNAS..10513081C. doi:10.1073 / pnas.0801523105. PMC 2529122. PMID 18723673.
- ^ Shipitsin, M; Campbell, L. L .; Argani, P; Weremowicz, S; Bloushtain-Qimron, N; Yao, J; Nikolskaya, T; Serebryiskaya, T; Beroukhim, R; Hučení; Halushka, M. K .; Sukumar, S; Parker, L. M .; Anderson, K. S .; Harris, L. N .; Garber, J. E.; Richardson, A. L .; Schnitt, S. J .; Nikolsky, Y; Gelman, R. S .; Polyak, K (2007). "Molekulární definice heterogenity nádoru prsu". Rakovinová buňka. 11 (3): 259–273. doi:10.1016 / j.ccr.2007.01.013. PMID 17349583.
- ^ MacIntosh, C. A .; Stower, M; Reid, N; Maitland, N. J. (1998). „Přesná mikrodisekce lidských karcinomů prostaty odhaluje genotypovou heterogenitu“. Výzkum rakoviny. 58 (1): 23–28. PMID 9426051.
- ^ Alvarado, C; Beitel, L. K .; Sircar, K; Aprikian, A; Trifiro, M; Gottlieb, B (2005). „Somatický mozaikismus a rakovina: Mikro-genetické vyšetření role genu pro androgenní receptor v rakovině prostaty“. Výzkum rakoviny. 65 (18): 8514–8518. doi:10.1158 / 0008-5472.CAN-05-0399. PMID 16166332.
- ^ Konishi, N; Hiasa, Y; Matsuda, H; Tao, M; Tsuzuki, T; Hayashi, I; Kitahori, Y; Shiraishi, T; Yatani, R; Shimazaki, J (1995). "Intratumorová buněčná heterogenita a změny v ras onkogenu a p53 tumor supresorovém genu u lidského karcinomu prostaty". American Journal of Pathology. 147 (4): 1112–1122. PMC 1871010. PMID 7573356.
- ^ González-García, I; Solé, R. V .; Costa, J (2002). „Dynamika metapopulace a prostorová heterogenita u rakoviny“. Sborník Národní akademie věd. 99 (20): 13085–13089. Bibcode:2002PNAS ... 9913085G. doi:10.1073 / pnas.202139299. PMC 130590. PMID 12351679.
- ^ Samowitz, W. S .; Slattery, M. L. (1999). "Regionální reprodukovatelnost nestability mikrosatelitů u sporadického kolorektálního karcinomu". Geny, chromozomy a rakovina. 26 (2): 106–114. doi:10.1002 / (SICI) 1098-2264 (199910) 26: 2 <106 :: AID-GCC2> 3.0.CO; 2-F. PMID 10469448.
- ^ Giaretti, W; Monako, R; Pujic, N; Rapallo, A; Nigro, S; Geido, E (1996). "Intratumorová heterogenita mutací K-ras2 v kolorektálních adenokarcinomech: asociace se stupněm aneuploidie DNA". American Journal of Pathology. 149 (1): 237–245. PMC 1865212. PMID 8686748.
- ^ Heppner, G. H. (1984). "Heterogenita nádoru". Výzkum rakoviny. 44 (6): 2259–2265. PMID 6372991.
- ^ Maley, C. C .; Galipeau, P. C .; Finley, J. C .; Wongsurawat, V. J .; Li, X; Sanchez, C. A .; Paulson, T. G .; Blount, P.L .; Risques, R. A .; Rabinovitch, P. S .; Reid, B. J. (2006). „Genetická klonální diverzita předpovídá progresi do adenokarcinomu jícnu“. Genetika přírody. 38 (4): 468–473. doi:10.1038 / ng1768. PMID 16565718.
- ^ Califano, J; Van Der Riet, P; Westra, W; Nawroz, H; Clayman, G; Piantadosi, S; Corio, R; Lee, D; Greenberg, B; Koch, W; Sidransky, D (1996). "Model genetické progrese pro rakovinu hlavy a krku: důsledky pro rakovinu v terénu". Výzkum rakoviny. 56 (11): 2488–2492. PMID 8653682.
- ^ Sauter, G; Moch, H; Gasser, T. C .; Mihatsch, M. J .; Waldman, F. M. (1995). "Heterogenita chromozomu 17 a počet kopií genů erbB-2 u primární a metastatické rakoviny močového měchýře". Cytometrie. 21 (1): 40–46. doi:10,1002 / cyto.990210109. PMID 8529469.
- ^ Fujii, H; Yoshida, M; Gong, Z. X .; Matsumoto, T; Hamano, Y; Fukunaga, M; Hruban, R. H .; Gabrielson, E; Shirai, T (2000). „Častá genetická heterogenita v klonální evoluci gynekologického karcinosarkomu a její vliv na fenotypovou rozmanitost“. Výzkum rakoviny. 60 (1): 114–120. PMID 10646862.
- ^ Horvai, A.E .; Devries, S; Roy, R; O'Donnell, R. J .; Waldman, F (2009). „Podobnost v genetických změnách mezi spárovanými dobře diferencovanými a dediferencovanými složkami dediferencovaného liposarkomu“. Moderní patologie. 22 (11): 1477–1488. doi:10.1038 / modpathol.2009.119. PMID 19734852.
- ^ Pantou, D; Rizou, H; Tsarouha, H; Pouli, A; Papanastasiou, K; Stamatellou, M; Trangas, T; Pandis, N; Bardi, G (2005). "Cytogenetické projevy heterogenity mnohočetného myelomu". Geny, chromozomy a rakovina. 42 (1): 44–57. doi:10.1002 / gcc.20114. PMID 15495197.
- ^ A b Shackleton, M; Quintana, E; Fearon, E. R .; Morrison, S. J. (2009). „Heterogenita u rakoviny: rakovinové kmenové buňky versus klonální evoluce“. Buňka. 138 (5): 822–829. doi:10.1016 / j.cell.2009.08.017. PMID 19737509.
- ^ Lapidot, T; Sirard, C; Vormoor, J; Murdoch, B; Hoang, T; Caceres-Cortes, J; Minden, M; Paterson, B; Caligiuri, M. A .; Dick, J. E. (1994). "Buňka iniciující lidskou akutní myeloidní leukémii po transplantaci do SCID myší". Příroda. 367 (6464): 645–648. Bibcode:1994 Natur.367..645L. doi:10.1038 / 367645a0. PMID 7509044.
- ^ Wang, J. C .; Lapidot, T; Cashman, J. D .; Doedens, M; Addy, L; Sutherland, D. R .; Nayar, R; Laraya, P; Minden, M; Keating, A; Eaves, A. C .; Eaves, C. J .; Dick, J. E. (1998). „Vysoká úroveň štěpu NOD / SCID myší primitivními normálními a leukemickými hematopoetickými buňkami od pacientů s chronickou myeloidní leukémií v chronické fázi“. Krev. 91 (7): 2406–2414. doi:10,1182 / krev. V91.7.2406. PMID 9516140.
- ^ Singh, S. K .; Hawkins, C; Clarke, I.D .; Squire, J. A .; Bayani, J; Skrýt, T; Henkelman, R. M .; Cusimano, M. D .; Dirks, P. B. (2004). "Identifikace buněk iniciujících lidský mozkový nádor". Příroda. 432 (7015): 396–401. Bibcode:2004 Natur.432..396S. doi:10.1038 / nature03128. PMID 15549107.
- ^ Al-Hajj, M; Wicha, M. S .; Benito-Hernandez, A; Morrison, S. J .; Clarke, M. F. (2003). „Prospektivní identifikace tumorigenních buněk rakoviny prsu“. Sborník Národní akademie věd. 100 (7): 3983–3988. Bibcode:2003PNAS..100.3983A. doi:10.1073 / pnas.0530291100. PMC 153034. PMID 12629218.
- ^ Maitland, N.J .; Collins, A. T. (2008). „Kmenové buňky rakoviny prostaty: nový cíl terapie“. Journal of Clinical Oncology. 26 (17): 2862–2870. doi:10.1200 / JCO.2007.15.1472. PMID 18539965.
- ^ Meacham, C.E .; Morrison, S. J. (2013). „Heterogenita nádorů a plasticita rakovinných buněk“. Příroda. 501 (7467): 328–337. Bibcode:2013Natur.501..328M. doi:10.1038 / příroda12624. PMC 4521623. PMID 24048065.
- ^ Nowell, P. C. (1976). „Klonální vývoj populací nádorových buněk“. Věda. 194 (4260): 23–28. Bibcode:1976Sci ... 194 ... 23N. doi:10.1126 / science.959840. PMID 959840.
- ^ A b C Swanton, C (2012). „Různorodost intratumorů: Evoluce v prostoru a čase“. Výzkum rakoviny. 72 (19): 4875–4882. doi:10.1158 / 0008-5472.CAN-12-2217. PMC 3712191. PMID 23002210.
- ^ Merlo, L. M. F .; Pepper, J. W .; Reid, B. J .; Maley, C. C. (2006). „Rakovina jako evoluční a ekologický proces“. Nature Reviews Cancer. 6 (12): 924–935. doi:10.1038 / nrc2013. PMID 17109012.
- ^ A b C Gerlinger, M; Rowan, A. J .; Horswell, S; Larkin, J; Endesfelder, D; Gronroos, E; Martinez, P; Matthews, N; Stewart, A; Tarpey, P; Varela, já; Phillimore, B; Begum, S; McDonald, N. Q .; Butler, A; Jones, D; Raine, K; Latimer, C; Santos, C. R .; Nohadani, M; Eklund, A. C .; Spencer-Dene, B; Clark, G; Pickering, L; Razítko, G; Gore, M; Szallasi, Z; Dolů, J; Futreal, P. A .; Swanton, C (2012). „Heterogenita intratumorů a rozvětvená evoluce odhalená multiregionovým sekvenováním“. New England Journal of Medicine. 366 (10): 883–892. doi:10.1056 / NEJMoa1113205. PMC 4878653. PMID 22397650.
- ^ A b C Marusyk, A; Almendro, V; Polyak, K (2012). „Intra-tumorová heterogenita: Vypadající sklo pro rakovinu?“. Nature Reviews Cancer. 12 (5): 323–334. doi:10.1038 / nrc3261. PMID 22513401.
- ^ Burrell, R. A .; McGranahan, N; Bartek, J; Swanton, C (2013). „Příčiny a důsledky genetické heterogenity ve vývoji rakoviny“. Příroda. 501 (7467): 338–345. Bibcode:2013Natur.501..338B. doi:10.1038 / příroda12625. PMID 24048066.
- ^ Johnson, B. E .; Mazor, T; Hong, C; Barnes, M; Aihara, K; McLean, C. Y .; Fouse, S. D .; Yamamoto, S; Ueda, H; Tatsuno, K; Asthana, S; Jalbert, L. E.; Nelson, S. J .; Bollen, A. W .; Gustafson, W. C .; Charron, E; Weiss, W. A .; Smirnov, I. V .; Song, J. S .; Olshen, A. B .; Cha, S; Zhao, Y; Moore, R. A .; Mungall, A. J .; Jones, S. J .; Hirst, M; Marra, M. A .; Saito, N; Aburatani, H; Mukasa, A (2014). „Mutační analýza odhaluje původ a terapii řízený vývoj rekurentního gliomu“. Věda. 343 (6167): 189–193. Bibcode:2014Sci ... 343..189J. doi:10.1126 / science.1239947. PMC 3998672. PMID 24336570.
- ^ A b Ding, L; Ley, T. J .; Larson, D. E.; Miller, C. A .; Koboldt, D.C .; Welch, J. S .; Ritchey, J. K .; Young, M. A .; Lamprecht, T; McLellan, M. D .; McMichael, J. F .; Wallis, J. W .; Lu, C; Shen, D; Harris, C. C .; Dooling, D. J .; Fulton, R. S .; Fulton, L. L .; Chen, K; Schmidt, H; Kalicki-Veizer, J; Magrini, V. J .; Cook, L; McGrath, S. D .; Vickery, T. L .; Wendl, M. C .; Heath, S; Watson, M. A .; Link, D. C .; Tomasson, M. H. (2012). „Klonální evoluce u relabující akutní myeloidní leukémie odhalená sekvenováním celého genomu“. Příroda. 481 (7382): 506–510. Bibcode:2012Natur.481..506D. doi:10.1038 / příroda10738. PMC 3267864. PMID 22237025.
- ^ G.M.Edelman (1989). „Topobiologie“. J. Scientific American. 260 (5): 76–88. doi:10.1038 / scientificamerican0589-76. PMID 2717916.
- ^ V.E. Orel; N.N Dzyatkovskaya; M.I. Danko; A.V. Romanov; Y.I. Mel'nik; Y.A. Grinevich; S.V. Martynenko (2004). "Prostorový a mechanoemisní chaos mechanicky deformovaných nádorových buněk". J. Journal of Mechanics in Medicine and Biology. 4 (1): 31–45. doi:10.1142 / s0219519404000886.
- ^ V.E. Orel; A.V. Romanov; N.N. Dzyatkovskaya; Yu.I. Mel’nik (2002). "Zařízení a algoritmus pro odhad mechanoemissonového chaosu v krvi pacientů s rakovinou žaludku". J. Lékařské inženýrství a fyzika. 24 (5): 365–371. doi:10.1016 / s1350-4533 (02) 00022-x. PMID 12052364.
- ^ N. Khranovskaya; V. Orel; Y. Grinevich; O. Alekseenko; A. Romanov; O. Skachkova; N.Dzyatkovskaya; A. Burlaka; S.Lukin (2012). „Mechanická heterogenizace buněk Lewisova karcinomu plic může zlepšit antimetastatický účinek dendritických buněk“. J. Journal of Mechanics in Medicine and Biology. 3 (12): 22. doi:10.1142 / S0219519411004757.
- ^ Junttila, M. R.; De Sauvage, F. J. (2013). „Vliv heterogenity mikroprostředí nádoru na terapeutickou odpověď“. Příroda. 501 (7467): 346–354. Bibcode:2013Natur.501..346J. doi:10.1038 / příroda12626. PMID 24048067.
- ^ Auman, James Todd; McLeod, Howard L. (01.01.2010). "Buněčné linie rakoviny tlustého střeva a konečníku postrádají molekulární heterogenitu klinických kolorektálních nádorů". Klinický kolorektální karcinom. 9 (1): 40–47. doi:10.3816 / ccc.2010.n.005. PMID 20100687.
- ^ Cassidy, John W .; Caldas, Carlos; Bruna, Alejandra (01.08.2015). „Zachování heterogenity tumoru u xenograftů tumoru odvozených od pacienta“. Výzkum rakoviny. 75 (15): 2963–2968. doi:10.1158 / 0008-5472.CAN-15-0727. ISSN 0008-5472. PMC 4539570. PMID 26180079.
- ^ Bai H, Harmancı AS, Erson-Omay AZ, Li J, Coșkun S, Simon M, et al. (Listopad 2015). "Integrovaná genomová charakterizace maligní progrese gliomu s mutací IDH1". Genetika přírody. 48 (1): 59–66. doi:10,1038 / ng.3457. PMC 4829945. PMID 26618343.
- ^ Bedard, P.L .; Hansen, A. R .; Ratain, M. J .; Siu, L. L. (2013). „Heterogenita nádorů na klinice“. Příroda. 501 (7467): 355–364. Bibcode:2013Natur.501..355B. doi:10.1038 / příroda12627. PMC 5224525. PMID 24048068.
- ^ Dawson, S. J.; Tsui, D. W .; Murtaza, M; Biggs, H; Rueda, O. M .; Chin, S.F .; Dunning, M. J .; Gale, D; Forshew, T; Mahler-Araujo, B; Rajan, S; Humphray, S; Becq, J; Halsall, D; Wallis, M; Bentley, D; Caldas, C; Rosenfeld, N (2013). "Analýza cirkulující nádorové DNA pro monitorování metastatického karcinomu prsu". New England Journal of Medicine. 368 (13): 1199–1209. doi:10.1056 / NEJMoa1213261. PMID 23484797.
- ^ Gatenby, R. A .; Silva, A. S .; Gillies, R. J .; Frieden, B. R. (2009). „Adaptivní terapie“. Výzkum rakoviny. 69 (11): 4894–4903. doi:10.1158 / 0008-5472.CAN-08-3658. PMC 3728826. PMID 19487300.
- ^ Cibulskis, K; Lawrence, M. S .; Carter, S.L .; Sivachenko, A; Jaffe, D; Sougnez, C; Gabriel, S; Meyerson, M; Lander, E. S .; Getz, G (2013). „Citlivá detekce somatických bodových mutací v nečistých a heterogenních vzorcích rakoviny“. Přírodní biotechnologie. 31 (3): 213–219. doi:10.1038 / nbt.2514. PMC 3833702. PMID 23396013.
- ^ Koboldt, D.C .; Zhang, Q; Larson, D. E.; Shen, D; McLellan, M. D .; Lin, L; Miller, C. A .; Mardis, E. R .; Ding, L; Wilson, R. K. (2012). „Var Skenovat 2: Objev somatické mutace a změny počtu kopií u rakoviny sekvenováním exome ". Výzkum genomu. 22 (3): 568–576. doi:10.1101 / gr.129684.111. PMC 3290792. PMID 22300766.
- ^ Saunders, C. T .; Wong, W. S .; Swamy, S; Becq, J; Murray, L. J .; Cheetham, R. K. (2012). "Strelka: Přesné somatické volání malé varianty od sekvenovaných párů nádor-normální vzorek". Bioinformatika. 28 (14): 1811–1817. doi:10.1093 / bioinformatika / bts271. PMID 22581179.
- ^ Carter, S.L .; Cibulskis, K; Helman, E; McKenna, A; Shen, H; Zack, T; Laird, P. W .; Onofrio, R. C .; Winckler, W; Weir, B. A .; Beroukhim, R; Pellman, D; Levine, D. A .; Lander, E. S .; Meyerson, M; Getz, G (2012). „Absolutní kvantifikace somatických změn DNA u lidské rakoviny“ (PDF). Přírodní biotechnologie. 30 (5): 413–421. doi:10.1038 / nbt.2203. PMC 4383288. PMID 22544022.
- ^ Shah, S. P .; Roth, A; Goya, R; Oloumi, A; Ha, G; Zhao, Y; Turashvili, G; Ding, J; Tse, K; Haffari, G; Bashashati, A; Prentice, L. M .; Khattra, J; Burleigh, A; Yap, D; Bernard, V; McPherson, A; Shumansky, K; Crisan, A; Giuliany, R; Heravi-Moussavi, A; Rosner, J; Lai, D; Birol, I; Varhol, R; Tam, A; Dhalla, N; Zeng, T; Ma, K; Chan, S. K. (2012). „Spektrum klonální a mutační evoluce primárních triple-negativních karcinomů prsu“. Příroda. 486 (7403): 395–399. Bibcode:2012Natur.486..395S. doi:10.1038 / příroda10933. PMC 3863681. PMID 22495314.
- ^ Navin, N; Kendall, J; Troge, J; Andrews, P; Rodgers, L; McIndoo, J; Cook, K; Štěpánský, A; Levy, D; Esposito, D; Muthuswamy, L; Krasnitz, A; McCombie, W. R .; Hicks, J; Wigler, M (2011). „Evoluce nádoru odvozená od sekvenování podle jedné buňky“. Příroda. 472 (7341): 90–94. Bibcode:2011Natur.472 ... 90N. doi:10.1038 / nature09807. PMC 4504184. PMID 21399628.
- ^ Jahn, Katharina (2016). "Odvození stromu pro data z jedné buňky". Genome Biology. 17: 86. doi:10.1186 / s13059-016-0936-x. PMC 4858868. PMID 27149953.
- ^ Ross, Edith (2016). „OncoNEM: odvození vývoje nádoru z dat sekvenování jednotlivých buněk“. Genome Biology. 17: 69. doi:10.1186 / s13059-016-0929-9. PMC 4832472. PMID 27083415.
- ^ Zafar, Hamim (2017). „SiFit: odvození nádorových stromů z jednobuněčných sekvenčních dat podle modelů konečných míst“. Genome Biology. 18 (1): 178. doi:10.1186 / s13059-017-1311-2. PMC 5606061. PMID 28927434.
- ^ Zafar, Hamim (2019). „SiCloneFit: Bayesiánský závěr struktury populace, genotypu a fylogeneze nádorových klonů z jednobuněčných dat sekvenování genomu“. Výzkum genomu. 29 (11): 1847–1859. doi:10.1101 / gr.243121.118. PMC 6836738. PMID 31628257.
- ^ Malikic, Salem; Rashidi Mehrabadi, Farid (2019). „PhISCS: kombinatorický přístup k nedokonalé rekonstrukci fylogeneze nádoru pomocí integrovaného použití jednobuněčných a hromadných sekvenčních dat“. Výzkum genomu. 29 (11): 1860–1877. doi:10,1101 / gr.234435.118. PMID 31628256.
- ^ Sadeqi Azer, Erfan; Rashidi Mehrabadi, Farid (2020). „PhISCS-BnB: rychlý algoritmus větvení a vázání pro dokonalý problém rekonstrukce fylogeneze nádoru“. Bioinformatika. 36 (Supplement_1): i169 – i176. doi:10.1093 / bioinformatika / btaa464. PMID 32657358.
- ^ Kuipers, Jack (2017). „Pokroky v porozumění evoluci nádoru prostřednictvím jednobuněčného sekvenování“. Biochimica et Biophysica Acta (BBA) - Recenze na rakovinu. 1867 (2): 127–138. doi:10.1016 / j.bbcan.2017.02.001. PMC 5813714. PMID 28193548.
- ^ Schwartz, Russell (13. února 2017). „Vývoj fylogenetiky nádorů: principy a praxe“. Genetika hodnocení přírody. 18 (4): 213–229. doi:10.1038 / nrg.2016.170. PMC 5886015. PMID 28190876.
- ^ Farahani, Hossein; de Souza, Camila P. E .; Billings, Raewyn; Yap, Damian; Shumansky, Karey; Wan, Adrian; Lai, Daniel; Mes-Masson, Anne-Marie; Aparicio, Samuel; P. Shah, Sohrab (18. října 2017). „Inženýrské směsi buněčných linií in vitro a důkladné vyhodnocení výpočetních metod pro klonální rozklad a podélnou dynamiku rakoviny“. Vědecké zprávy. 7 (1): 13467. Bibcode:2017NatSR ... 713467F. doi:10.1038 / s41598-017-13338-8. PMC 5647443. PMID 29044127.
- ^ Zare, Habil (2014). „Odvození klonální kompozice z více částí rakoviny prsu“. PLOS výpočetní biologie. 10 (7): e1003703. Bibcode:2014PLSCB..10E3703Z. doi:10.1371 / journal.pcbi.1003703. PMC 4091710. PMID 25010360.
- ^ Fischer, Andrej (2014). „Rekonstrukce klonálního složení ve vysokém rozlišení u rakoviny“. Zprávy buněk. 7 (5): 1740–1752. doi:10.1016 / j.celrep.2014.04.055. PMC 4062932. PMID 24882004.
- ^ Deshwar, Amit (2015). „Monitorování progrese chronické lymfocytární leukémie sekvenováním celého genomu odhaluje heterogenní vzorce evoluce klonů“. Genome Biology. 16: 35. doi:10.1186 / s13059-015-0602-8. PMC 4359439. PMID 25786235.
- ^ Roth, Andrew (2014). „PyClone: statistická inference struktury klonální populace u rakoviny“. Přírodní metody. 11 (4): 396–398. doi:10.1038 / nmeth.2883. PMC 4864026. PMID 24633410.
- ^ Marass, Francesco (2015). "Fylogenetický latentní rysový model pro klonální dekonvoluci". Annals of Applied Statistics. 10 (4): 2377–2404. arXiv:1604.01715. doi:10.1214 / 16-AOAS986.
- ^ Matsui, Yusuke (2016). "phyC: Shlukování evolučních stromů rakoviny". PLOS výpočetní biologie. 13 (5): e1005509. Bibcode:2017PLSCB..13E5509M. bioRxiv 10.1101/069302. doi:10.1371 / journal.pcbi.1005509. PMC 5432190. PMID 28459850.
- ^ Jiang, Yuchao; Qiu, Yu; Minn, Andy J .; Zhang, Nancy R. (29. srpna 2016). „Posouzení heterogenity intratumoru a sledování podélné a prostorové klonální evoluční historie sekvenováním nové generace“. Sborník Národní akademie věd. 113 (37): E5528–37. doi:10.1073 / pnas.1522203113. PMC 5027458. PMID 27573852.
- ^ Salehi, Sohrab (2017). „ddClone: společný statistický závěr klonálních populací z údajů o sekvenování jednotlivých buněk a hromadných nádorů“. Genome Biology. 18 (1): 44. doi:10.1186 / s13059-017-1169-3. PMC 5333399. PMID 28249593.
- ^ Satas, Gryte (2017). „Odvození fylogeneze nádoru pomocí vzorkování důležitosti omezené stromem“. Bioinformatika. 33 (14): i152 – i160. doi:10.1093 / bioinformatika / btx270. PMC 5870673. PMID 28882002.
- ^ Geng, Yu (2017). "Identifikace heterogenních vzorců alelické nerovnováhy na germlinových variantách k odvození klonální architektury". Mezinárodní konference o inteligentních počítačích. Přednášky z informatiky. 10362: 286–297. doi:10.1007/978-3-319-63312-1_26. ISBN 978-3-319-63311-4.
- ^ Ramazzotti, Daniele; Graudenzi, Alex; Sano, Luca De; Antoniotti, Marco; Caravagna, Giulio (4. září 2017). "Učení mutačních grafů evoluce jednotlivých nádorů z dat sekvenování více vzorků". bioRxiv 10.1101/132183.
- ^ Roman, Theodore; Xie, Lu; Schwartz, Russell; Raphael, Benjamin J. (23. října 2017). „Automatická dekonvoluce strukturovaných směsí z genomických dat heterogenních nádorů“. PLOS výpočetní biologie. 13 (10): e1005815. arXiv:1604.02487. Bibcode:2017PLSCB..13E5815R. doi:10.1371 / journal.pcbi.1005815. PMC 5695636. PMID 29059177.
- ^ Malikic, Salem (2017). „Integrativní odvození vývoje subklonálního nádoru z jednobuněčných a objemových sekvenčních dat“. bioRxiv 10.1101/234914.
- ^ Oesper, Layla; Mahmoody, Ahmad; Raphael, Benjamin J. (29. července 2013). „THetA: odvození intra-nádorové heterogenity z vysoce výkonných dat sekvenování DNA“. Genome Biology. 14 (7): R80. doi:10.1186 / gb-2013-14-7-r80. ISSN 1474-760X. PMC 4054893. PMID 23895164.
- ^ Zeng, By (2018). "Identifikace subklonů nádorů na základě fylogeneze pomocí Bayesianského modelu přidělení funkcí". arXiv:1803.06393 [stat.AP ].
- ^ Cun, Yupeng; Yang, Tsun-Po; Achter, Viktor; Lang, Ulrich; Peifer, Martin (24. května 2018). "Analýza počtu kopií a odvození subklonálních populací v genomech rakoviny pomocí Sclustu". Přírodní protokoly. 13 (6): 1488–1501. doi:10.1038 / nprot.2018.033. ISSN 1754-2189. PMID 29844525.
- ^ Wang, Xiaodong; Ogundijo, Oyetunji E. (01.12.2019). „SeqClone: sekvenční odvození nádorových subklonů na základě Monte Carla“. BMC bioinformatika. 20 (1): 6. doi:10,1186 / s12859-018-2562-r. ISSN 1471-2105. PMC 6320595. PMID 30611189.
- ^ Raphael, Benjamin J .; Satas, Gryte; Myers, Matthew A. (22. ledna 2019). „Odvození vývoje nádoru z podélných vzorků“. bioRxiv: 526814. doi:10.1101/526814.
- ^ Toosi, Hosein; Moeini, Ali; Hajirasouliha, Iman (6. června 2019). „BAMSE: Výběr Bayesovského modelu pro odvození fylogeneze nádoru mezi více vzorky“. BMC bioinformatika. 20 (11): 282. doi:10.1186 / s12859-019-2824-3. ISSN 1471-2105. PMC 6551234. PMID 31167637.
- ^ Ricketts, Camir; Seidman, Daniel; Popic, Victoria; Hormozdiari, Fereydoun; Batzoglou, Serafim; Hajirasouliha, Iman (4. října 2019). „Meltos: Rekonstrukce fylogeneze více vzorků tumoru pro strukturní varianty“. Bioinformatika. 36 (4): 1082–1090. doi:10.1093 / bioinformatika / btz737. PMID 31584621.
- ^ Sundermann, Linda. "Reconstructing tumor evolutionary histories and clone trees in polynomial-time with SubMARine" (PDF). bioRxiv. Citováno 22. června 2020.
- ^ Zhou, Tianjian. "RNDCLONE: TUMOR SUBCLONE RECONSTRUCTION BASED ON INTEGRATING DNA AND RNA SEQUENCE DATA" (PDF). Citováno 23. srpna 2020.
