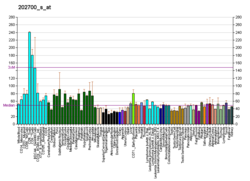
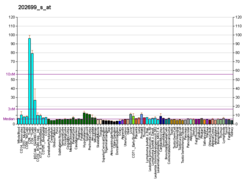
TMEM63A - TMEM63A
Transmembránový protein 63A je protein že u lidí je kódován TMEM63A gen.[5][6][7] Zralý lidský protein je přibližně 92,1 kilodaltonů (kDa), s relativně vysokou ochranou hmoty v ortologech.[8] Protein obsahuje jedenáct transmembránové domény a je vložen do membrány lysozom.[9][10] Analýza BioGPS pro TMEM63A u lidí ukazuje, že gen je všudypřítomně exprimován s nejvyššími úrovněmi exprese nalezenými v T-buňky a dendritické buňky.[11]
Gen
Přehled
TMEM63A se nachází na negativním řetězci DNA chromozom 1 na místě 1q42.12, klenout se základní páry 226 033 237 až 22 6 070 069.[7] Aliasy zahrnují KIAA0489 a KIAA0792. Produkt lidského genu je 4 469 základní pár mRNA s 25 předpokládanými exony.[12] Existuje 9 předpokládaných sestřihových izoforem genu, z nichž tři kódují proteiny. Promotér analýza byla provedena pomocí El Dorado[13] skrz Genomatix stránka softwaru. Předpokládaná oblast promotoru zahrnuje 971 základní páry, od 226 070 920 do 226 069 950 na záporném řetězci chromozom 1.
Genové sousedství
TMEM63A se nachází v sousedství EPHX1 gen na pozitivním smyslovém řetězci DNA na chromozom 1, stejně jako LEVIČÁK 1 gen na negativním sense řetězci.[7] Další geny ve stejné oblasti chromozom 1 zahrnout SRP9 a LEVIČÁK 3 na kladném řetězci a MIR 6741 a PYCR2 na záporném řetězci.
Výraz
TMEM63 je všudypřítomně exprimován v lidském těle na různých úrovních a vyskytuje se s nejvyšší relativní prevalencí v CD 8+ T buňky a CD 4+ T buňky.[11][14] Mírné relativní úrovně exprese jsou také pozorovány v celém mozku, zejména v mozku týlní lalok, temenní lalok, a slinivka břišní.[14] Analýza TMEM63A Exprese u myší pomocí BioGPS odhalila více variabilních vzorců exprese, přičemž nejvyšší exprese byla pozorována v žaludek a tlusté střevo.[11] Pomocí programu El Dorado z Genomatix, transkripční faktor regulace, která zjistila, že „TMEM63A“ je vysoce regulován E2F buněčný cyklus regulační orgány a EGR1, faktor považovaný za a gen potlačující nádor s výrazem v mozek.[13] The 3 ‘UTR se předpokládá, že bude vázán regulačním prvkem miR-9 / 9ab.[15]
Protein
Vlastnosti a vlastnosti
Zralá forma lidského proteinu TMEM63A má 807 aminokyselina zbytky s izoelektrický bod ze dne 6.925.[8] To je v ortologech poměrně konzervativní. A VÝBUCH srovnání ukázalo, že protein obsahuje tři domény: RSN1_TM a dvě domény neznámé funkce (DUF4463 a DUF221).[16] Předpokládá se účast RSN1_TM Golgi váček doprava a exocytóza. DUF4463 je cytosolický a vzdáleně homologní k Proteiny vázající RNA. Tuto doménu lze použít k určení orientace proteinu v membrána, s N-konec bílkoviny v lysozom a C-konec nachází se v cytosol.
Posttranslační modifikace byla stanovena experimentálně i pomocí bioinformatické analýza. Existují dvě pravděpodobná místa glykosylace na proteinu: N38 a N450.[17] Ty byly předpovězeny pomocí programu NetNGlyc z EXPASY a aminokyselinová sekvence TMEM63A, stejně jako odvozená orientace proteinu v membráně.[18] Existují tři pravděpodobné stránky fosforylace na proteinu: S85, S98 a S735, které byly předpovězeny pomocí programu NetPhos.[19]
Protein má tři izoformy. Zralý protein se označuje jako izoforma CRA. Další dvě izoformy jsou X1 a X2, což je 630 aminokyselinových zbytků, respektive 468 aminokyselinových zbytků. Isoform X1 chybí N-konec zralého proteinu, zatímco izoformě 2 chybí C-konec.[8]
Interakce
Pomocí textových informací se předpokládá, že TMEM63A potenciálně interaguje s dalšími šesti proteiny: EEF1D,[20] FAM163B, CPNE9, TMEM90A, STAC2, HEATR3, a WDR67.[21]
Funkce
Funkce TMEM63A není známa, ačkoli jedna studie zjistila, že se nacházela v oblasti pravděpodobně regulované mir-200a, spojený s epiteliální homeostázou.[22] Jiný zjistil, že je v kvantitativním znakovém lokusu spojeném s haloperidol -indukovaný katalepsie.[23]
Evoluční historie
Paralogy
TMEM63A má dva paralogy: TMEM63B, který je umístěn na 6p21.1, a TMEM63C, který je umístěn na C14orf171.[24] Zarovnání mezi nimi ukazuje, že TMEM63C je více příbuzný TMEM63B než TMEM63A.[8] Srovnání BLAST ukázalo homologii TMEM63A a TMEM63B k proteinům stejně vzdáleným jako rostliny, zatímco TMEM63C byl homologní pouze tak vzdáleně jako v drosophila.[16] To naznačuje, že se TMEM63C pravděpodobně lišila od dvou na začátku roku bezobratlých.
Ortologický prostor
TMEM63A má velký ortologický prostor s homology přítomnými v organismech vzdálených příbuzných jako rostliny.
| Rod a druh | Běžné jméno | Třída | Přistoupení | Procento identity |
|---|---|---|---|---|
| Otolemur garnettii | Bush dítě | Mammalia | XP_003791028.1 | 91% |
| Vicugna pacos | Alpaka | Mammalia | XP_006198896.1 | 92% |
| Mus musculus | Myš | Mammalia | NP_659043.1 | 90% |
| Trichechus manatus latirostris | Západoindický kapustňák | Mammalia | XP_004375949.1 | 89% |
| Canis lupus familiaris | Pes | Mammalia | NP_001274088.1 | 89% |
| Myotis davidii | Netopýr ušatý | Mammalia | XP_006761379.1 | 80% |
| Pelodiscus sinensis | Čínská softshellová želva | Sauropsida | XP_006118107.1 | 71% |
| Alligator sinensis | Čínský aligátor | Reptilia | XP_006016630.1 | 70% |
| Ficedula albicollis | Límeček černohlavý | Aves | XP_005043078.1 | 69% |
| Gallus gallus | Červená džungle | Aves | XP_419384.3 | 68% |
| Xenopus tropicalis | Západní drápá žába | Obojživelníci | NP_001072343.1 | 65% |
| Ictalurus punctatus | Kanál sumec | Actinopterygii | AHH42519.1 | 54% |
| Culex quinquefasciatus | Jižní dům komár | Insecta | XP_001861445.1 | 34% |
| Clonorchis sinensis | Čínská jaterní náhoda | Trematoda | GAA53916.1 | 23% |
| Oryza sativa | Asijská rýže | Liliopsida | NP_001065504.1 | 20% |
Reference
- ^ A b C GRCh38: Vydání souboru 89: ENSG00000196187 - Ensembl, Květen 2017
- ^ A b C GRCm38: Vydání souboru 89: ENSMUSG00000026519 - Ensembl, Květen 2017
- ^ „Human PubMed Reference:“. Národní centrum pro biotechnologické informace, Americká národní lékařská knihovna.
- ^ „Myš PubMed Reference:“. Národní centrum pro biotechnologické informace, Americká národní lékařská knihovna.
- ^ Nagase T, Ishikawa K, Suyama M, Kikuno R, Miyajima N, Tanaka A, Kotani H, Nomura N, Ohara O (duben 1999). „Predikce kódujících sekvencí neidentifikovaných lidských genů. XI. Kompletní sekvence 100 nových klonů cDNA z mozku, které kódují velké proteiny in vitro“. DNA Res. 5 (5): 277–86. doi:10.1093 / dnares / 5.5.277. PMID 9872452.
- ^ Seki N, Ohira M, Nagase T, Ishikawa K, Miyajima N, Nakajima D, Nomura N, Ohara O (únor 1998). "Charakterizace cDNA klonů v knihovnách cDNA s frakcionací podle velikosti z lidského mozku". DNA Res. 4 (5): 345–9. doi:10.1093 / dnares / 4.5.345. PMID 9455484.
- ^ A b C „Entrez Gene: TMEM63A transmembránový protein 63A“.
- ^ A b C d "Analýza TMEM63A". Pracovní stůl biologie. Superpočítačové centrum v San Diegu - Kalifornská univerzita v San Diegu. Citováno 8. května 2014.[trvalý mrtvý odkaz ]
- ^ Schroder BA, Wrocklage C, Hasilik A, Saftig P (19. října 2010). "Proteom lysozomů". Proteomika. 10 (22): 4053–4076. doi:10.1002 / pmic.201000196. PMID 20957757.
- ^ Schroder BA, Wrocklage C, Pan C, Jager R, Kosters B, Schafer H, Elsasser HP, Mann M, Hasilik A (28. srpna 2007). "Integrované a asociované lysozomální membránové proteiny". Provoz. 8 (12): 1676–1686. doi:10.1111 / j.1600-0854.2007.00643.x. PMID 17897319.
- ^ A b C „BioGPS: TMEM63A“. Citováno 12. května 2014.
- ^ "Soubor: TMEM63A". Citováno 8. května 2014.
- ^ A b „El Dorado“. Genomatix. Citováno 17. dubna 2014.
- ^ A b „GDS596 / 214833_at / TMEM63A“. NCBI.
- ^ „TargetScanHuman 6.2“. Whitehead Institute for Biomedical Research. Citováno 23. dubna 2014.
- ^ A b Marchler-Bauer A a kol. (2011). „CDD: Databáze chráněných domén pro funkční anotaci proteinů“. Nucleic Acids Res. 39 (D): 225–229. doi:10.1093 / nar / gkq1189. PMC 3013737. PMID 21109532.
- ^ „O94886 (TM63A_HUMAN)“. UniProtKB. Citováno 5. května 2014.
- ^ Gupta R, Jung E, Brunak S (2004). "Predikce N-glykosylačních míst v lidských proteinech". Citovat deník vyžaduje
| deník =(Pomoc) - ^ Blorn N, Gammeltoft S, Brunak S (1999). "Predikce sekvencí a struktur na fosforylačních místech eukaryotických proteinů". Journal of Molecular Biology. 294 (5): 1351–1362. doi:10.1006 / jmbi.1999.3310. PMID 10600390.
- ^ „Genové karty“. Weizmann Institute of Science. Citováno 16. května 2014.
- ^ "Řetězcová databáze". Citováno 16. května 2014.
- ^ Bonnet E, Tatari M, Joshi A a kol. (2010). „Inference sítě modulů z datové sady genové exprese rakoviny identifikuje regulované moduly microRNA“. PLOS ONE. 5 (4): e10162. doi:10.1371 / journal.pone.0010162. PMC 2854686. PMID 20418949.
- ^ Hofstetter JR, Hitzemann RJ, Belknap JK, Walter NA, McWeeney SK, Mayeda AR (2008). „Charakterizace lokusu kvantitativních znaků pro haloperidolem indukovanou katalepsii na distálním myším chromozomu 1“. Geny, mozek a chování. 7 (2): 214–223. doi:10.1111 / j.1601-183x.2007.00340.x. PMID 17696997.
- ^ Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ (říjen 1990). Msgstr "Základní vyhledávací nástroj pro místní zarovnání". J. Mol. Biol. 215 (3): 403–10. doi:10.1016 / S0022-2836 (05) 80360-2. PMID 2231712.
Další čtení
- Gregory SG, Barlow KF, McLay KE a kol. (2006). „Sekvence DNA a biologická anotace lidského chromozomu 1“. Příroda. 441 (7091): 315–21. doi:10.1038 / nature04727. PMID 16710414.
- Gerhard DS, Wagner L, Feingold EA a kol. (2004). „Stav, kvalita a rozšíření projektu cDNA NIH v plné délce: Mammalian Gene Collection (MGC)“. Genome Res. 14 (10B): 2121–7. doi:10,1101 / gr. 2596504. PMC 528928. PMID 15489334.
- Ota T, Suzuki Y, Nishikawa T a kol. (2004). „Kompletní sekvenování a charakterizace 21 243 lidských cDNA plné délky“. Nat. Genet. 36 (1): 40–5. doi:10.1038 / ng1285. PMID 14702039.
- Strausberg RL, Feingold EA, Grouse LH a kol. (2003). „Generování a počáteční analýza více než 15 000 lidských a myších cDNA sekvencí plné délky“. Proc. Natl. Acad. Sci. USA. 99 (26): 16899–903. doi:10.1073 / pnas.242603899. PMC 139241. PMID 12477932.
- Suzuki Y, Yoshitomo-Nakagawa K, Maruyama K a kol. (1997). "Konstrukce a charakterizace knihovny cDNA obohacené o celou délku a 5'-end". Gen. 200 (1–2): 149–56. doi:10.1016 / S0378-1119 (97) 00411-3. PMID 9373149.
- Maruyama K, Sugano S (1994). „Oligo-capping: jednoduchá metoda k nahrazení struktury cap eukaryotických mRNA oligoribonukleotidy“. Gen. 138 (1–2): 171–4. doi:10.1016/0378-1119(94)90802-8. PMID 8125298.