Úpravy genomu mimo cíl - Off-target genome editing
Úpravy genomu mimo cíl se týká nespecifických a nezamýšlených genetických modifikací, které mohou vzniknout použitím vytvořená nukleáza technologie jako: klastrované, pravidelně rozložené, krátké palindromické opakování (CRISPR )-Cas9, transkripční aktivátorové efektorové nukleázy (TALEN ), meganukleázy, a nukleázy zinkového prstu (ZFN).[1] Tyto nástroje používají k navázání předem určené sekvence různé mechanismy DNA („Cíl“), které štěpí (nebo „stříhají“) a vytvářejí dvouvláknový chromozomální zlom (DSB), který vyvolává Mechanismy opravy DNA (nehomologní spojování konců (NHEJ ) a homologní rekombinace (HR )) a vede k místně specifickým úpravám.[2] Pokud se tyto komplexy neváží na cíl, často výsledkem homologních sekvencí a / nebo tolerance nesouladu, budou štěpit mimo cíl DSB a způsobovat nespecifické genetické modifikace.[3][4][5] Konkrétně se mimocílové efekty skládají z nezamýšlených bodové mutace,[6] vypuštění,[7][8] vložení[5] inverze,[5] a translokace.[9][8]
Designové nukleázové systémy, jako je CRISPR-cas9, se stávají stále populárnějšími výzkumnými nástroji díky své jednoduchosti, škálovatelnosti a cenové dostupnosti.[10][11] S tímto je řečeno, mimocílové genetické modifikace jsou časté a mohou změnit funkci jinak neporušených genů. Několik studií, které používaly časná činidla CRISPR-cas9, zjistilo, že více než 50% mutací indukovaných endonukleázou naváděných RNA se na cíli nevyskytovalo.[3][7] The Cas9 naváděcí RNA (gRNA) rozpoznává cílovou sekvenci DNA o 20 bp, kterou se váže a štěpí za účelem „úpravy“ sekvence DNA. Vazba cílové sekvence však může tolerovat neshody až několika párů bází, což znamená, že často existují tisíce možných vazebných míst, která představují několik experimentálních a bezpečnostních problémů.[12][3] V oblasti výzkumu mohou účinky mimo cíl zmást proměnné v biologických studiích, což vede k potenciálně zavádějícím a nereprodukovatelným výsledkům.[2] V klinické sféře obklopují hlavní obavy narušení životně důležitých kódujících oblastí, což vede k genotoxickým účinkům, jako je rakovina.[13] V souladu s tím zlepšení specifičnosti[14][15] nástrojů pro úpravu genomu a detekce[9][16] dopadů mimo cíl rychle postupují v oblastech výzkumu. Takový výzkum zahrnuje vývoj návrhářských nukleáz[17] a objev,[18] výpočetní predikční programy a databáze,[19][20] a vysoce výkonné sekvenování[9][16] snížit a předvídat mutační výskyt. Mnoho návrhářských nukleázových nástrojů je stále v relativním plenkách a jako jejich molekulární vlastnosti a in vivo chování bude lépe pochopeno, bude čím dál přesnější a předvídatelnější.
Mechanismy
Systém CRISPR-Cas9 funguje jako adaptivní imunitní systém v bakteriích a archaeách.[21] Když virus infikuje bakterie, tento systém začleňuje segmenty virové DNA do bakteriálního genomu. Při druhé invazi transkripty z těchto sekvencí směrují aktivitu nukleázy na její komplementární sekvenci v napadajícím viru tak, aby ji zničila.[22][23][24]
Aby bylo možné tuto metodu extrapolovat na eukaryota, aby bylo možné vyvinout metodu úpravy genu, je nutný protein Cas9, rozpoznávací sekvence RNA a transaktivující RNA. Fúze jak rozpoznávací sekvenční specificity CRISPR RNA (crRNA), tak transaktivační RNA (tracrRNA) se běžně používá v experimentech a nazývá se jedna vodicí RNA (sgRNA).[25] Plní obě funkce: prvních 20 nukleotidů sgRNA je komplementárních s cílovou sekvencí DNA (funkce cr), zatímco následující nukleotidy jsou součástí protospacer sousední motiv (PAM; funkce tracr).[26][27][28]
Vazba nukleázy mimo cílení pochází z částečné, ale dostatečné shody s cílovou sekvencí. Mechanismy vazby mimo cíl lze seskupit do dvou hlavních forem: tolerance nesouladu základny a nesoulad boule.[29]
Tolerance nesouladu základny
I když se předpokládá, že specificita Cas9 je řízena 20nt sgRNA a PAM, off-target mutace stále převládají a mohly by nastat s až 3 až 5 párů bazických párů (z 20) mezi sgRNA a cílovou sekvencí DNA.[25][30] Kromě toho by sekundární struktury sgRNA mohly také ovlivnit štěpení míst v cílové a mimo cílové oblasti. Jak již bylo zmíněno výše, sgRNA se skládá ze sekvence (~ 20nt), která je komplementární k cílovým sekvencím, a poté následuje sekvence PAM, která aktivuje endonukleáza aktivita. I když se ukázalo, že 10–12 nt přilehlých k PAM (nazývaných „sekvence semen“) stačilo pro specificitu Cas9, Wu et al. ukázal, že v katalyticky mrtvém Cas9 je pro specifičnost vyžadováno pouze 1-5 párů bazí sekvence semen.[31] To bylo později prokázáno i jinými studiemi. Vazba na protein Cas9 je dále ovlivněna řadou mechanismů:
- Sekvence semen určuje frekvenci semen plus PAM v genomu a řídí efektivní koncentraci komplexu Cas9 sgRNA.
- Semena bohatá na uracil mají pravděpodobně nízkou hladinu sgRNA a zvyšují specificitu, protože více uracilů v sekvenci může zavést ukončení sgRNA transkripce.[31][32]
- Nesoulady na 5 'konci crRNA jsou snášenlivější, protože důležité místo by sousedilo s PAM maticí. Jednoduché a dvojité nesoulady jsou také tolerovány na základě toho, jak je umístit.
- V nedávné studii Ren et al. pozorovali souvislost mezi účinností mutageneze a obsahem GC v sgRNA. K dobré úpravě je zapotřebí alespoň 4–6 bp sousedících s PAM.[33]
- Při výběru gRNA je guanin upřednostňován před cytosinem jako první bází semene sousedícího s PAM, cytosin jako první v 5 'a adenin uprostřed sekvence. Tento design je založen na stabilitě spojené s tvorbou G čtyřlůžkové pokoje.[31][32][34]
- A Čip byla provedena Kim et al. předvádění, že přidání čištěného Cas9 spolu s sgRNA způsobilo nízké cílové účinky, což znamená, že tyto účinky způsobuje více faktorů.[35]
Je důležité si uvědomit, že methylace DNA CpG míst snižuje účinnost vazby Cas9 a dalších faktorů v buňkách. Proto existuje epigenetický odkaz, který bude více prozkoumán pro budoucnost editace epigenomu.[36]
Variace v PAM sekvenci mohou také ovlivnit aktivitu sgRNA, což zase ovlivní samotnou sgRNA. V běžně používaných systémech Cas9 je motivem PAM 5 „NGG 3“, kde N představuje kterýkoli ze čtyř nukleotidů DNA. Požadavek na PAM sekvenci může způsobit problémy se specificitou, protože některé oblasti budou mít k dispozici cílovou sekvenci pro provedení požadované genetické modifikace. Zpráva uvádí, že 99,96% míst, o nichž se dříve předpokládalo, že jsou jedinečnými cíli Cas9 v lidských exonech, může mít potenciální účinky mimo cíl obsahující NAG nebo NGG PAM a nesoulad jedné báze v sekvenci semen.[37]
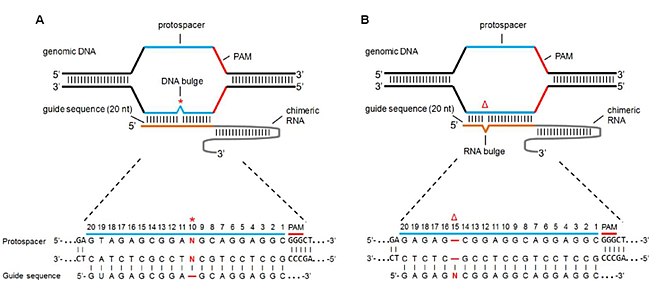
Nesoulad boule
Jak mimo cílová místa s chybějícími bázemi (nebo delecemi), tak mimo cílová místa s dalšími bázemi (nebo inzercemi) nazývanými RNA boule a DNA boule, mají vliv na specificitu Cas9 a aktivitu štěpení. Lin et al. napodobil tyto boule přidáním a odstraněním bází ze sekvence sgRNA tak, že by delece báze v sgRNA poskytla vyboulení RNA a vložení báze vedlo k vyboulení DNA.[7] Studiem mutačních rychlostí pomocí NHEJ dospěli k následujícím výsledkům:
- V případě vydutí čisté DNA byly mutace dobře tolerovány (tj. Stále ještě převládala aktivita štěpení Cas9). Oblasti tolerance boule zahrnovaly sedm bází z PAM a 5 'a 3' konce sekvence semen. To mělo za následek podobné nebo mírně vyšší (v některých případech) mutace ve srovnání s nulovými boulemi.
- V případě výčnělků čistě RNA byla indukována vyšší aktivita Cas9 na mnoha pozicích ve srovnání s výčnělky DNA. Tato charakteristika byla přičítána skutečnosti, že RNA je pružnější než DNA, a má tedy menší vazebný trest s vydutím RNA, což má za následek vyšší toleranci a vyšší cílové mutace.[38]
- Vyšší obsah GC v sekvenci sgRNA vedl k vyšší toleranci, a tedy k vyšší rychlosti mimo cílovou mutaci.
- Výdechy 2bp-5bp byly nápadně tolerantnější a vyvolávaly mutace než jediná 2bp boule.
Vylepšení


Metody ke zvýšení specificity
Široce používané Streptococcus pyogenes Nukleáza Cas9 (SpCas9) je účinná, ale indukuje nežádoucí mimotelové mutace při vysokých frekvencích. Bylo popsáno několik inženýrských a screeningových metod ve snaze snížit off-target mutace v celém genomu, včetně mutace nukleázy, protospacer sousední motiv (PAM) změna sekvence, průvodce RNA (gRNA) zkrácení a objev nových nukleáz.[39] Například v roce 2013 Fu et al. uvedli, že zkrácením gRNA z délky <20 bp na 17 nebo 18 bp se cílová specificita nukleázy zvýšila až 5 000krát a případy nesouladu nad 3 bázemi se vyskytly zřídka, pokud k nim vůbec došlo.[14]
Cas9 nickázy
Nukleázu spCas9 lze také mutovat různými způsoby, aby se zlepšila specificita a kontrola. Nukleázové domény mohou být mutovány nezávisle na sobě na tzv Cas9 nickázy. Tyto nukleázy mají jednu aktivní a neaktivní doménu nukleáz, což má za následek vznik komplexu, který provádí jednořetězcové štěpení.[4] Cas9 nickázy mohou být použity v tandemu (známé jako spárované nickázy), které provádějí dva jednořetězcové „řezy“ na alternativních vláknech.[4] Při použití této strategie musí oba Cas9 nickasy společně lokalizovat, vázat a štěpit svůj cíl, což drasticky snižuje pravděpodobnost off-target indels.[4] Také DSB štěpené spárovanými nickázami mají namísto tupých konců dlouhé převisy, které poskytují vylepšenou kontrolu cílených inzercí.
Fok1-dCas9 a dimerizační nukleázy
Tak jako monomerní nukleázy často zahrnují vysokou úroveň účinků mimo cíl, dimerizace je atraktivní strategie. V dimerním systému se musí obě nukleázy vázat na své jednotlivé cíle nebo „poloviční místa“ a poté interagovat a dimerizovat, aby zahájily štěpení, což výrazně snižuje pravděpodobnost účinků mimo cíl. Metoda, která zahrnuje spolehlivost závislou na dimerizaci FokI byly vyvinuty nukleázové domény používané v ZFN a TALEN s jednoduchostí CRISPR-cas9.[17] FokI nukleáza byla původně nalezena v Flavobacterium okeanokoites, a bude štěpit pouze DNA s aktivací dimerizace. V zásadě vědci spojili tuto nukleázu s komplexem CRISPR s neaktivní nukleázou Cas9 (Fok1-dCas9).[17] GRNA směruje komplex CRISPR do cílového místa, ale „štěpení“ je provedeno dimerizovaným Fok1. Odhaduje se, že strategie Fok1-dCas9 snižuje detekovatelné účinky mimo cíl 10 000krát, což je efektivní pro aplikace vyžadující vysoce přesnou a specifickou úpravu genomu.[17][40]
Mutace nukleázy
Kromě cíle gRNA vyžaduje Cas9 vazbu na specifickou 2-6 nukleotidovou sekvenci PAM. V běžně používaných systémech SpCas9 je motivem PAM 5 „NGG 3“, kde N představuje kterýkoli ze čtyř nukleotidů DNA. Požadavek sekvence PAM může způsobit omezení specificity, protože některé oblasti nebudou mít k dispozici cílovou sekvenci pro provedení požadované genetické modifikace. Sekvenci PAM lze upravit na nekanonické motivy NAG a NGA, které nejen zlepšují specificitu, ale také snižují účinky mimo cíl.[41] A Mutant D1135E Zdá se, že mění specificitu PAM. Mutant D1135E snižuje účinky mimo cíl a zvyšuje specificitu SpCas9.[41] Další varianta, SpCas9-HF1, také vede k příznivému zlepšení specificity Cas9.[42] Bylo identifikováno několik kombinací substitucí, které vytvářejí nespecifické kontakty DNA (N497A, R661A, Q695A a Q926A).[42] Čtyřnásobná substituce těchto zbytků (později pojmenovaná SpCas9-HF1) má extrémně nízké úrovně mimo cílových účinků, jak je detekováno GUIDE-seq experimenty.[42] Varianty jako SpCas9-HF1 a D1135E a další podobné lze kombinovat, testovat a snadno přidávat ke stávajícím vektorům SpCas9, aby se snížila rychlost mutací mimo cíl. Mnoho z výše uvedených inženýrských strategií lze navíc kombinovat a vytvořit tak stále robustnější a spolehlivější nástroje pro úpravu nukleáz vedené RNA. Směrovaná evoluce může být také použita ke snížení aktivity nukleáz na konkrétní cílové sekvence, což vede k variantám, jako je SpartaCas (obsahující mutace D23A, T67L, Y128V a D1251G ve srovnání s divokým typem SpCas9).[43]
CRISPRi a CRISPRa
CRISPR interference (CRISPRi ) a aktivace CRISPR (CRISPRa ) byly také vyvinuty.[44] Tyto systémy mohou přesně změnit transkripci genů na úrovni DNA, aniž by způsobily nevratné genetické změny.[44] Navíc přímým působením na DNA jsou obecně specifičtější a předvídatelnější ve srovnání s RNAi.[45] Ačkoli CRISPRi / a nemůže nahradit editaci genomu ve všech experimentech, mohou v některých případech působit jako účinná alternativa. CRISPRi a CRISPRa používají deaktivovaný enzym Cas9 (dCas9), který nemůže štěpit DNA, ale může dodávat transkripční aktivátory a represory k modulaci požadované genové exprese s vysokou přesností.[44] V současné době jsou mimo cílové účinky CRISPRi minimální a vykazují sníženou odezvu a citlivost na neshody jedné báze.[44] Důležité je, že pokud se nevyhnutelně vyskytnou nespecifické účinky, jsou reverzibilní, časově závislé a méně škodlivé než úpravy DNA, což z nich činí efektivní alternativy, které mohou omezit mimosmyslovou zátěž, kdykoli je to možné. CRISPR-cas13b pomocí systému CRISPR-Cas typu IV (na rozdíl od běžně používaného typu II) lze cílit a upravovat specifické sekvence RNA.[46] Taková platforma pro editaci RNA má schopnost specificky editovat mRNA, a tedy translaci proteinu, aniž by se změnila DNA. Představuje slibnou technologii, která, pokud bude úspěšná, sníží zátěž nevratných off-target mutací.
Detekce
I když je možné provést pečlivá opatření, abychom se vyhnuli mutacím mimo cíl, ai když se to podaří, je třeba provést potvrzovací obrazovku, aby bylo možné prověřit nezamýšlené mutace. V současné době existuje spousta neobjektivních a nezaujatých metod pro takovou obrazovku a pouze dvě in vitro metody. Všechny tyto položky jsou uvedeny níže:
Cílené sekvenování, exome a celé genomy
V případě normálního cíleného sekvenování předpjatý přístup přinese výsledky pouze pro zamýšlenou oblast zachycení, což znemožňuje hledání, protože na obrazovce se neobjeví žádné neočekávané mutace. I když je to snadné a levné, po přidání více cílových stránek se stává časově náročným a nákladným. Exome sekvenování využívá zachycení exomu k získání oblastí kódujících proteiny genomu. Je nezaujatý, nicméně nepřinese cílové mutace v nekódující oblasti genomu. V případě sekvenování celého genomu, je celý genom vyšetřován na mimo cílové mutace. V současné době je tato metoda nákladná a stejně jako sekvenování exome vyžaduje celý genom k provedení závěrů referenční genom.[47]
ŽEHNAT

ŽEHNAT je nejjednodušší způsob detekce a kvantifikace off-target mutací skríninkem na DSB v genomu. Tato metoda se spoléhá na přímou in situ přeruší označování obohacení na streptavidin. Vyvinut v roce 2013,[48] BLESS se provádí ligací konců DSB s biotinem, tj. Biotinylací. Poté následuje separace / sběr uvedených ligovaných konců pomocí streptavidinu. Spojená sekvence se přidá k biotinylovaným sekvencím a tato finální směs se poté sekvenuje, čímž se získá poloha mutace mimo cíl. Být nestranný v přírodě, BLESS poskytuje informace o místě mutace v genomu, spíše než o bílkovinách zapojených nebo spojených s DSB. BLESS však dokáže detekovat pouze mutace v době experimentu, nikoli ty, které byly vytvořeny dříve a byly opraveny.[49]
LAM-HTGTS
Zprostředkováno lineární zesílení - vysoce propustné sekvenční sekvenování genomu nebo LAM-HTGTS je metoda vyvinutá ke sledování translokačních událostí způsobených spojením mezi DSB.[50] Tato technika, vyvinutá pro detekci mutací mimo cíl z TALEN a CRISPR-Cas9, je založena na opravě DNA koncem spojení v DSB. Jakmile je nukleáza přidána, pokračuje v produkci on- a off-target mutací. Spolu s tím existuje sekvence návnady, která se také štěpí. Pokud se tedy na jiném chromozomu než na chromozomu sekvence návnady vyskytne další DSB, oba se spojí, což vede k translokaci. Protože je návnadová sekvence známá, je tato translokovaná sekvence amplifikována pomocí primerů. V případě, že nedochází k translokaci, existuje restrikční místo, ve kterém se štěpí, aby se zabránilo amplifikaci pouze sekvence návnady. Amplifikovaná DNA se poté sekvenuje, aby se studovaly velké genomové přesmyky způsobené mutacemi mimo cíl. Jednou nevýhodou je, že se spoléhá na současnou přítomnost návnady a dalšího DSB.
GUIDE-Seq
Dalším přístupem k nalezení mimocílových mutací v důsledku aktivity nukleáz je GUIDE-Seq metoda. GUIDE-seq nebo Genome Wide Nestranná identifikace DSB povolená sekvenováním je založena na zabudování dvouřetězcových oligodeoxynukleotidů (dsODN) do DSB prostřednictvím NHEJ. Po jeho zesílení následuje sekvenování. Protože k sekvenci dsODN budou použity dva primery, budou amplifikovány oblasti lemující DSB spolu s DSB. To umožňuje mapování mutace mimo cíl. Tato technika byla použita k identifikaci všech dříve známých off-target stránek i nových s frekvencemi tak nízkými jako 0,03%. Stejně jako BLESS však může GUIDE-seq detekovat pouze DSB přítomné v době studia.
Digenome-Seq
Jeden ze současných in vitro Metody, Digenome-Seq využívá vlastnost Cas9 štěpit genom, aby získal nestranný profil celého genomu. V této metodě je Cas9 přidán do gDNA a následné účinky jsou studovány pomocí vysoce výkonného sekvenování. Vzhledem k tomu, že fragmenty jsou tvořeny v důsledku stejné nukleázy, mohou být konce těchto fragmentů mapovány zarovnány. Dvě velké výhody spočívají v tom, že jej lze použít ke studiu až 10 gRNA najednou a dokáže identifikovat cíle s frekvencí pouhých 0,01%.[9] Hlavní výhodou však je, že tato metoda je in vitro tj. DSB zavedené Cas9 nebudou zpracovány strojem pro opravu DNA (na rozdíl od BLESS a GUIDE-seq) a budou tedy zahrnovat všechny možné mimo cílové mutanty. Může to však také vést k velkému počtu falešných poplachů.[51]
KRUH SEKV
Nejnovější přírůstek do in vitro metod detekce mutací mimo cíl je CIRCLE-seq. Licencováno společností Beacon genomics (spolu s GUIDE-seq),[52] CIRCLE-seq si klade za cíl odstranit nevýhody Digenome-seq, jako je potřeba velké velikosti vzorku a hloubky čtení (~ 400 milionů čtení) a vysoké pozadí, které ztěžuje identifikaci nízkofrekvenčních štěpných událostí.[53] Přijímá a restrikční enzym nezávislá strategie pro vytvoření a výběr konverze náhodně stříhané DNA. Při štěpení vytvoří cílová DNA kmenovou smyčku, do které lze přidat adaptéry pro sekvenování. I když se to ukázalo jako možné, druhá možnost přinesla dvojnásobně vysoký rozdíl v detekci /. Ve druhém případě je sekvence štěpena pomocí Cas9 a když je znovu štěpena na polovičním místě, je k dispozici kruhový řez (což je důvod pro název CIRCLE-seq). Téměř všechny weby identifikované cirkularizací obsahují jak lineární detekovaná místa, tak i novější, což naznačuje, že CIRCLE-seq mezi předpovědi nezaujímá a získává také silné nízkofrekvenční přerušení. Dále pomáhá sekvenovat místo přerušení z obou stran štěpení ve srovnání s jinými metodami, které mají pouze jednu stranu pro čtení.
Čárově kódované knihovny cílů
Rovněž mohou být napadeny nukleázy, jako je Cas9 in vitro randomizovanými knihovnami cílů.[54] Ligace adaptéru pro kvantifikaci štěpených a neštěpených členů knihovny umožňuje objektivní měření specifičnosti profilu nukleázy. Měření štěpení čárově kódovaných knihoven cílů (BLT) pomocí SpCas9 ukázalo, že profily specificity byly specifické pro průvodce a závisí na naváděcí sekvenci i na samotné nukleáze. Objektivní profily specificity založené na každém konkrétním komplexu Cas9-gRNA lze poté použít k vytvoření prediktivních modelů specifických pro průvodce pro in vitro výstřih.
Význam
Genová terapie
Aby technologie úpravy genů učinily skok k bezpečnému a rozšířenému použití na klinice, je třeba zastarat míru modifikace mimo cíl. Bezpečnost léčby genovou terapií je nanejvýš důležitá, zejména během klinických studií, kdy modifikace mimo cíl mohou blokovat další vývoj kandidátského produktu.[55] Snad nejznámějším příkladem moderní genové terapie je terapie CAR-T, která se používá k léčbě B-buněčný lymfom. Aby se omezila rychlost štěpení mimo cíl, používá terapie vysoce specifický a jemně vyladěný TALEN, u kterého se prokázalo, že má malou nebo žádnou interakci pozadí mimo cíl.[55] VOZÍK imunoterapie je ex vivo postup, což znamená, že imunitní buňky pacienta (v tomto případě T-buňky ) jsou extrahovány a upravovány pomocí návrhářských nukleáz.[55] Zatímco vývoj systému TALEN je nákladný a časově náročný, výzkumné a technické úpravy drasticky omezily jejich míru interakce mimo cíl. Pacienti, kteří jsou léčeni, jsou však stále často sledováni a budou po dobu příštích 15 let, aby bylo možné analyzovat účinky mimo cíl a imunogenní odpovědi a vzít je v úvahu, když se do klinického hodnocení dostanou nové genové terapie.[56]
Testy autologních pomocných T buněk CCR5 ZFN
Fáze I / II klinické hodnocení zahrnovalo 12 pacientů se syndromem získané imunodeficience (AIDS), aby otestovali bezpečnost a účinnost podávání ZFN-modifikovaných autologních pomocných T buněk.[57] Prostřednictvím cílených delecí vlastní ZFN deaktivuje chemokinový receptor C-C 5 (CCR5 ) gen, který kóduje koreceptor, který používá virus HIV k vstupu do buňky.[58] V důsledku vysokého stupně sekvenční homologie mezi C-C chemokinovými receptory se tento ZFN také štěpí CCR2, což vedlo k delecím ∼15kb mimo cíl a genomickým přeskupením.[58][59] Dopady těchto CCR2 úpravy stále nejsou známy a doposud nebyly hlášeny žádné vedlejší účinky. Nicméně, CCR2 je známo, že má mnoho kritických rolí v nervových a metabolických systémech.[60][61]
Genové disky
Inženýrské genové disky využívající CRISPR-cas9 jsou v současné době testovány a byly navrženy jako strategie k eliminaci invazivních druhů a vektorů chorob. Genetickou modifikací organismu k expresi endogenní sekvenčně specifické endonukleázy lze cíl (například gen plodnosti) štěpit na opačném chromozomu.[62] DSB v cíli vede k homologní opravě, která účinně činí organismus homozygotní pro požadovanou cílovou sekvenci. Tato strategie, známá jako naváděcí pohon, může potlačit populaci ovlivněním kritického genu nebo indukcí recesivní sterility. Pokud by však byl takový systém vypuštěn do volné přírody, systém CRISPR-cas9 by zůstal fungovat neomezeně dlouho. S každou další generací by mutace mimo cíl byly stále pravděpodobnější a účinky těchto mutací na druh by byly stochastické. Off-target mutace by mohly deaktivovat supresivní vlastnosti genového pohonu při zachování exprese endonukleázy. V takové situaci by existovalo zvýšené riziko toku genů mezi cílovým druhem a jinými druhy, což by pravděpodobně vedlo k nežádoucím výsledkům.[63]
Kontroverze
Zvýšené používání editace genomu a jeho případný překlad směrem ke klinickému použití vyvolalo kontroverze kolem skutečné zátěže technologií mimo cíl.
Schaefer et al. 2017
30. května 2017 byl v Nature Methods publikován dvoustránkový korespondenční článek, který uváděl neobvykle vysoký počet mimo cíl SNV a indely po sekvenování myší, které byly dříve zapojeny do in vivo experiment s opravou genů.[64] Předchozí experiment, dokončený stejnou skupinou, úspěšně obnovil vizi slepého kmene myší (1. kolo) opravou mutace Y347X v Pde6b gen pomocí systému CRISPR-cas9.[65] Po dokončení experimentu byly dvě geneticky opravené myši sekvenovány v celém genomu a porovnány s kontrolními a známými genomy myšího kmene. Bylo objeveno více než 1 600 SNV a 128 indelů, z nichž 1 397 SNV a 117 indelů bylo sdíleno mezi dvěma upravenými myšmi, což naznačuje, že účinky mimo cíl nebyly náhodné. Algoritmy pokoušející se předpovědět umístění těchto mimotelových mutací selhaly u drtivé většiny lokusů. Pro srovnání, studie sekvenování celé exome z roku 2016 našla 19 SNV a 3 indely u 5 upravených myší, zatímco Schaefer et al. našel 115 exonických SNV a 9 indelů pouze u 2 upravených myší.[66] Mnoho odborníků s papírem nesouhlasilo a kritizovali jej prostřednictvím článků v časopisech[66] a sociální média, což naznačuje, že v původním článku byla použita neobvyklá léčba CRISPR a velikost vzorku byla příliš významná (n = 2). Nature Methods vydal dvě redakční poznámky na papíře.[67] Stále se však zjistí, že sazby mimo cíl jsou častější in vivo ve srovnání s experimenty na buněčné kultuře a jsou považovány za zvláště běžné u lidí.[3][7]
Reference
- ^ Costa JR, Bejcek BE, McGee JE, Fogel AI, Brimacombe KR, Ketteler R (2004). Sittampalam GS, Coussens NP, Brimacombe K, Grossman A, Arkin M, Auld D a kol. (eds.). Manuál pro stanovení testu. Bethesda (MD): Eli Lilly & Company a National Center for Advancing Translational Sciences. PMID 29165977.
- ^ A b Cong L, Zhang F (2015). „Genome engineering using CRISPR-Cas9 system“. Metody v molekulární biologii. 1239: 197–217. doi:10.1007/978-1-4939-1862-1_10. hdl:1721.1/102943. ISBN 978-1-4939-1861-4. PMC 3969860. PMID 25408407.
- ^ A b C d Fu Y, Foden JA, Khayter C, Maeder ML, Reyon D, Joung JK, Sander JD (září 2013). „Vysokofrekvenční mimotelová mutageneze vyvolaná nukleázami CRISPR-Cas v lidských buňkách“. Přírodní biotechnologie. 31 (9): 822–6. doi:10,1038 / nbt.2623. PMC 3773023. PMID 23792628.
- ^ A b C d Cho SW, Kim S, Kim Y, Kweon J, Kim HS, Bae S, Kim JS (leden 2014). „Analýza mimocílových účinků CRISPR / Cas-odvozených RNA naváděných endonukleáz a nickáz“. Výzkum genomu. 24 (1): 132–41. doi:10,1101 / gr.162339.113. PMC 3875854. PMID 24253446.
- ^ A b C Veres A, Gosis BS, Ding Q, Collins R, Ragavendran A, Brand H, Erdin S, Cowan CA, Talkowski ME, Musunuru K (červenec 2014). „Nízký výskyt mutací mimo cíl u jednotlivých klonů lidských kmenových buněk cílených na CRISPR-Cas9 a TALEN detekovaných sekvenováním celého genomu“. Buňková kmenová buňka. 15 (1): 27–30. doi:10.1016 / j.stem.2014.04.020. PMC 4082799. PMID 24996167.
- ^ Wang H, Yang H, Shivalila CS, Dawlaty MM, Cheng AW, Zhang F, Jaenisch R (květen 2013). „Jednostupňová generace myší nesoucích mutace ve více genech metodou genomu zprostředkovanou CRISPR / Cas“. Buňka. 153 (4): 910–8. doi:10.1016 / j.cell.2013.04.025. PMC 3969854. PMID 23643243.
- ^ A b C d Lin Y, Cradick TJ, Brown MT, Deshmukh H, Ranjan P, Sarode N, Wile BM, Vertino PM, Stewart FJ, Bao G (červen 2014). „Systémy CRISPR / Cas9 mají aktivitu mimo cíl s inzercemi nebo delecemi mezi cílovou sekvencí DNA a sekvencemi naváděcí RNA“. Výzkum nukleových kyselin. 42 (11): 7473–85. doi:10.1093 / nar / gku402. PMC 4066799. PMID 24838573.
- ^ A b Cradick TJ, Fine EJ, Antico CJ, Bao G (listopad 2013). „Systémy CRISPR / Cas9 zaměřené na geny β-globin a CCR5 mají podstatnou aktivitu mimo cíl“. Výzkum nukleových kyselin. 41 (20): 9584–92. doi:10.1093 / nar / gkt714. PMC 3814385. PMID 23939622.
- ^ A b C d Kim D, Bae S, Park J, Kim E, Kim S, Yu HR, Hwang J, Kim JI, Kim JS (březen 2015). „Digenome-seq: profilování účinků CRISPR-Cas9 mimo cíl v lidských buňkách v celém genomu“. Přírodní metody. 12 (3): 237–43, 1 s po 243. doi:10.1038 / nmeth.3284. PMID 25664545.
- ^ Mahfouz MM, Piatek A, Stewart CN (říjen 2014). „Genomové inženýrství prostřednictvím systémů TALEN a CRISPR / Cas9: výzvy a perspektivy“. Plant Biotechnology Journal. 12 (8): 1006–14. doi:10.1111 / pbi.12256. PMID 25250853.
- ^ Pennisi E (srpen 2013). "CRISPR šílenství". Věda. 341 (6148): 833–6. doi:10.1126 / science.341.6148.833. PMID 23970676.
- ^ Hsu PD, Lander ES, Zhang F (červen 2014). „Vývoj a aplikace CRISPR-Cas9 pro genomové inženýrství“. Buňka. 157 (6): 1262–78. doi:10.1016 / j.cell.2014.05.010. PMC 4343198. PMID 24906146.
- ^ Eid A, Mahfouz MM (říjen 2016). „Úpravy genomu: cesta CRISPR / Cas9 od lavice po kliniku“. Experimentální a molekulární medicína. 48 (10): e265. doi:10.1038 / emm.2016.111. PMC 5099421. PMID 27741224.
- ^ A b Fu Y, Sander JD, Reyon D, Cascio VM, Joung JK (březen 2014). „Zlepšení specificity nukleázy CRISPR-Cas pomocí zkrácených vodicích RNA“. Přírodní biotechnologie. 32 (3): 279–284. doi:10,1038 / nbt.2808. PMC 3988262. PMID 24463574.
- ^ Standage-Beier K, Zhang Q, Wang X (listopad 2015). „Cílené rozsáhlé delece bakteriálních genů pomocí CRISPR-Nickáz“. ACS Syntetická biologie. 4 (11): 1217–25. doi:10.1021 / acssynbio.5b00132. PMC 4655420. PMID 26451892.
- ^ A b Tsai SQ, Nguyen NT, Malagon-Lopez J, Topkar VV, Aryee MJ, Joung JK (červen 2017). „CIRCLE-seq: vysoce citlivý in vitro screening pro genomovou CRISPR-Cas9 nukleázu mimo cíle“. Přírodní metody. 14 (6): 607–614. doi:10,1038 / nmeth.4278. PMC 5924695. PMID 28459458.
- ^ A b C d Tsai SQ, Wyvekens N, Khayter C, Foden JA, Thapar V, Reyon D, Goodwin MJ, Aryee MJ, Joung JK (červen 2014). „Dimerní CRISPR RNA naváděné FokI nukleázy pro vysoce specifickou editaci genomu“. Přírodní biotechnologie. 32 (6): 569–76. doi:10.1038 / nbt.2908. PMC 4090141. PMID 24770325.
- ^ Ungerer J, Pakrasi HB (prosinec 2016). „Cpf1 je všestranný nástroj pro úpravy genomu CRISPR napříč různými druhy sinic“. Vědecké zprávy. 6: 39681. doi:10.1038 / srep39681. PMC 5175191. PMID 28000776.
- ^ Singh R, Kuscu C, Quinlan A, Qi Y, Adli M (říjen 2015). „Informace o vazbě Cas9-chromatinu umožňují přesnější predikci CRISPR mimo cíl“. Výzkum nukleových kyselin. 43 (18): e118. doi:10.1093 / nar / gkv575. PMC 4605288. PMID 26032770.
- ^ Tsai SQ, Zheng Z, Nguyen NT, Liebers M, Topkar VV, Thapar V, Wyvekens N, Khayter C, Iafrate AJ, Le LP, Aryee MJ, Joung JK (únor 2015). „GUIDE-seq umožňuje genomové profilování štěpení mimo cíl nukleázami CRISPR-Cas“. Přírodní biotechnologie. 33 (2): 187–197. doi:10.1038 / nbt.3117. PMC 4320685. PMID 25513782.
- ^ Horvath P, Barrangou R (leden 2010). „CRISPR / Cas, imunitní systém bakterií a archea“. Věda. 327 (5962): 167–70. doi:10.1126 / science.1179555. PMID 20056882.
- ^ Gasiunas G, Barrangou R, Horvath P, Siksnys V (září 2012). „Ribonukleoproteinový komplex Cas9-crRNA zprostředkovává specifické štěpení DNA pro adaptivní imunitu bakterií“. Sborník Národní akademie věd Spojených států amerických. 109 (39): E2579-86. doi:10.1073 / pnas.1208507109. PMC 3465414. PMID 22949671.
- ^ Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E (srpen 2012). "Programovatelná duální RNA vedená endonukleáza DNA v adaptivní bakteriální imunitě". Věda. 337 (6096): 816–21. doi:10.1126 / science.1225829. PMC 6286148. PMID 22745249.
- ^ Zhang XH, Tee LY, Wang XG, Huang QS, Yang SH (listopad 2015). „Efekty mimo cíl v genomovém inženýrství zprostředkovaném CRISPR / Cas9“. Molekulární terapie. Nukleové kyseliny. 4: e264. doi:10.1038 / mtna.2015.37. PMC 4877446. PMID 26575098.
- ^ A b Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA, Zhang F (únor 2013). „Multiplexní genomové inženýrství využívající systémy CRISPR / Cas“. Věda. 339 (6121): 819–23. doi:10.1126 / science.1231143. PMC 3795411. PMID 23287718.
- ^ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Almendros C (březen 2009). "Krátké sekvence motivů určují cíle prokaryotického obranného systému CRISPR". Mikrobiologie. 155 (Pt 3): 733–40. doi:10.1099 / mic.0.023960-0. PMID 19246744.
- ^ Sternberg SH, Redding S, Jinek M, Greene EC, Doudna JA (březen 2014). "Výslech DNA pomocí CRISPR RNA naváděné endonukleázy Cas9". Příroda. 507 (7490): 62–7. doi:10.1038 / příroda13011. PMC 4106473. PMID 24476820.
- ^ Anders C, Niewoehner O, Duerst A, Jinek M (září 2014). „Strukturní základ rozpoznávání cílové DNA závislé na PAM pomocí endonukleázy Cas9“. Příroda. 513 (7519): 569–73. doi:10.1038 / příroda13579. PMC 4176945. PMID 25079318.
- ^ Lin Y, Cradick TJ, Brown MT, Deshmukh H, Ranjan P, Sarode N, Wile BM, Vertino PM, Stewart FJ, Bao G (červen 2014). „Systémy CRISPR / Cas9 mají aktivitu mimo cíl s inzercemi nebo delecemi mezi cílovou sekvencí DNA a sekvencemi naváděcí RNA“. Výzkum nukleových kyselin. 42 (11): 7473–85. doi:10.1093 / nar / gku402. PMC 4066799. PMID 24838573.
- ^ Hsu PD, Scott DA, Weinstein JA, Ran FA, Konermann S, Agarwala V, Li Y, Fine EJ, Wu X, Shalem O, Cradick TJ, Marraffini LA, Bao G, Zhang F (září 2013). „DNA zaměřená na specificitu nukleáz Cas9 vedených RNA“. Přírodní biotechnologie. 31 (9): 827–32. doi:10,1038 / nbt.2647. PMC 3969858. PMID 23873081.
- ^ A b C Wu X, Scott DA, Kriz AJ, Chiu AC, Hsu PD, Dadon DB, Cheng AW, Trevino AE, Konermann S, Chen S, Jaenisch R, Zhang F, Sharp PA (červenec 2014). „Vazba endonukleázy CR9PR CR9PR v buňkách savců na celý genom“. Přírodní biotechnologie. 32 (7): 670–6. doi:10,1038 / nbt.2889. PMC 4145672. PMID 24752079.
- ^ A b Wang T, Wei JJ, Sabatini DM, Lander ES (leden 2014). „Genetické obrazovky v lidských buňkách pomocí systému CRISPR-Cas9“. Věda. 343 (6166): 80–4. doi:10.1126 / science.1246981. PMC 3972032. PMID 24336569.
- ^ Ren X, Yang Z, Xu J, Sun J, Mao D, Hu Y, Yang SJ, Qiao HH, Wang X, Hu Q, Deng P, Liu LP, Ji JY, Li JB, Ni JQ (listopad 2014). „Vylepšená specificita a účinnost systému CRISPR / Cas9 s optimalizovanými parametry sgRNA v Drosophile“. Zprávy buněk. 9 (3): 1151–62. doi:10.1016 / j.celrep.2014.09.044. PMC 4250831. PMID 25437567.
- ^ Moreno-Mateos MA, Vejnar CE, Beaudoin JD, Fernandez JP, Mis EK, Khokha MK, Giraldez AJ (říjen 2015). „CRISPRscan: návrh vysoce účinných sgRNA pro cílení CRISPR-Cas9 in vivo“. Přírodní metody. 12 (10): 982–8. doi:10.1038 / nmeth.3543. PMC 4589495. PMID 26322839.
- ^ Kim S, Kim D, Cho SW, Kim J, Kim JS (červen 2014). „Vysoce účinná editace genomu vedená RNA v lidských buňkách prostřednictvím dodávky purifikovaných ribonukleoproteinů Cas9“. Výzkum genomu. 24 (6): 1012–9. doi:10,1101 / gr. 171322.113. PMC 4032847. PMID 24696461.
- ^ Kuscu C, Arslan S, Singh R, Thorpe J, Adli M (červenec 2014). „Analýza v rámci celého genomu odhaluje charakteristiky mimo cílových míst vázaných endonukleázou Cas9“. Přírodní biotechnologie. 32 (7): 677–83. doi:10,1038 / nbt.2916. PMID 24837660.
- ^ Mali P, Aach J, Stranges PB, Esvelt KM, Moosburner M, Kosuri S, Yang L, Church GM (září 2013). „Transkripční aktivátory CAS9 pro screening cílové specificity a spárované nickázy pro kooperativní genomové inženýrství“. Přírodní biotechnologie. 31 (9): 833–8. doi:10,1038 / nbt.2675. PMC 3818127. PMID 23907171.
- ^ Alberts B., Johnson A., Lewis J., Raff M., Roberts K., Walter P. (2007). Molekulární biologie buňky. New York: Garland Science.CS1 maint: více jmen: seznam autorů (odkaz)
- ^ Kadam US, Shelake RM, Chavhan RL, Suprasanna P (2018). „Obavy týkající se„ mimo cíl “aktivity endonukleáz upravujících genom“. Fyziologie a biochemie mravenců. 131: 22–30. doi:10.1016 / j.plaphy.2018.03.027. PMID 29653762.
- ^ Wyvekens N, Topkar VV, Khayter C, Joung JK, Tsai SQ (červenec 2015). "Dimeric CRISPR RNA-Guided FokI-dCas9 Nucleases Directed by Truncated gRNAs for Highly Specific Genome Editing". Lidská genová terapie. 26 (7): 425–31. doi:10.1089/hum.2015.084. PMC 4509490. PMID 26068112.
- ^ A b Kleinstiver BP, Prew MS, Tsai SQ, Topkar VV, Nguyen NT, Zheng Z, Gonzales AP, Li Z, Peterson RT, Yeh JR, Aryee MJ, Joung JK (July 2015). "Engineered CRISPR-Cas9 nucleases with altered PAM specificities". Příroda. 523 (7561): 481–5. doi:10.1038/nature14592. PMC 4540238. PMID 26098369.
- ^ A b C Kleinstiver BP, Pattanayak V, Prew MS, Tsai SQ, Nguyen NT, Zheng Z, Joung JK (January 2016). "High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects". Příroda. 529 (7587): 490–5. doi:10.1038/nature16526. PMC 4851738. PMID 26735016.
- ^ Cerchione, Derek; Loveluck, Katherine; Tillotson, Eric L .; Harbinski, Fred; DaSilva, Jen; Kelley, Chase P .; Keston-Smith, Elise; Fernandez, Cecilia A .; Myer, Vic E .; Jayaram, Hariharan; Steinberg, Barrett E. (16. dubna 2020). „SMOOT knihovny a fágem indukovaná řízená evoluce Cas9 k inženýrství snížené aktivity mimo cíl“. PLOS ONE. 15 (4): e0231716. doi:10.1371 / journal.pone.0231716.
- ^ A b C d Gilbert LA, Horlbeck MA, Adamson B, Villalta JE, Chen Y, Whitehead EH, Guimaraes C, Panning B, Ploegh HL, Bassik MC, Qi LS, Kampmann M, Weissman JS (October 2014). "Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation". Buňka. 159 (3): 647–61. doi:10.1016/j.cell.2014.09.029. PMC 4253859. PMID 25307932.
- ^ Larson MH, Gilbert LA, Wang X, Lim WA, Weissman JS, Qi LS (November 2013). "CRISPR interference (CRISPRi) for sequence-specific control of gene expression". Přírodní protokoly. 8 (11): 2180–96. doi:10.1038/nprot.2013.132. PMC 3922765. PMID 24136345.
- ^ Cox DB, Gootenberg JS, Abudayyeh OO, Franklin B, Kellner MJ, Joung J, Zhang F (November 2017). "RNA editing with CRISPR-Cas13". Věda. 358 (6366): 1019–1027. doi:10.1126/science.aaq0180. PMC 5793859. PMID 29070703.
- ^ Zischewski J, Fischer R, Bortesi L (2017). "Detection of on-target and off-target mutations generated by CRISPR/Cas9 and other sequence-specific nucleases". Biotechnologické pokroky. 35 (1): 95–104. doi:10.1016/j.biotechadv.2016.12.003. PMID 28011075.
- ^ Crosetto N, Mitra A, Silva MJ, Bienko M, Dojer N, Wang Q, Karaca E, Chiarle R, Skrzypczak M, Ginalski K, Pasero P, Rowicka M, Dikic I (duben 2013). „Mapování dvouřetězcových zlomů DNA s nukleotidovým rozlišením sekvenováním příští generace“. Přírodní metody. 10 (4): 361–5. doi:10.1038 / nmeth.2408. PMC 3651036. PMID 23503052.
- ^ Tsai SQ, Joung JK (May 2016). "Defining and improving the genome-wide specificities of CRISPR-Cas9 nucleases". Genetika hodnocení přírody. 17 (5): 300–12. doi:10.1038/nrg.2016.28. PMC 7225572. PMID 27087594.
- ^ Mallin H, Hestericová M, Reuter R, Ward TR (May 2016). "Library design and screening protocol for artificial metalloenzymes based on the biotin-streptavidin technology". Přírodní protokoly. 11 (5): 835–52. doi:10.1038/nprot.2016.019. PMID 27031496.
- ^ Fu BX, St Onge RP, Fire AZ, Smith JD (June 2016). "Distinct patterns of Cas9 mismatch tolerance in vitro and in vivo". Výzkum nukleových kyselin. 44 (11): 5365–77. doi:10.1093/nar/gkw417. PMC 4914125. PMID 27198218.
- ^ "CIRCLE-Seq Press Release". beacongenomics.com. Citováno 2018-03-01.
- ^ Tsai SQ, Nguyen NT, Malagon-Lopez J, Topkar VV, Aryee MJ, Joung JK (June 2017). "CIRCLE-seq: a highly sensitive in vitro screen for genome-wide CRISPR-Cas9 nuclease off-targets". Přírodní metody. 14 (6): 607–614. doi:10.1038/nmeth.4278. PMC 5924695. PMID 28459458.
- ^ Huston, Nicholas C.; Tycko, Josh; Tillotson, Eric L .; Wilson, Christopher J.; Myer, Vic E .; Jayaram, Hariharan; Steinberg, Barrett E. (1 June 2019). "Identification of Guide-Intrinsic Determinants of Cas9 Specificity". CRISPR Journal. 2 (3): 172–185. doi:10.1089/crispr.2019.0009. ISSN 2573-1599.
- ^ A b C Gautron AS, Juillerat A, Guyot V, Filhol JM, Dessez E, Duclert A, Duchateau P, Poirot L (December 2017). "Fine and Predictable Tuning of TALEN Gene Editing Targeting for Improved T Cell Adoptive Immunotherapy". Molekulární terapie. Nukleové kyseliny. 9: 312–321. doi:10.1016/j.omtn.2017.10.005. PMC 5684446. PMID 29246309.
- ^ Levine BL, Miskin J, Wonnacott K, Keir C (March 2017). "Global Manufacturing of CAR T Cell Therapy". Molekulární terapie. Metody a klinický vývoj. 4: 92–101. doi:10.1016/j.omtm.2016.12.006. PMC 5363291. PMID 28344995.
- ^ Tebas P, Stein D, Tang WW, Frank I, Wang SQ, Lee G, Spratt SK, Surosky RT, Giedlin MA, Nichol G, Holmes MC, Gregory PD, Ando DG, Kalos M, Collman RG, Binder-Scholl G, Plesa G, Hwang WT, Levine BL, June CH (March 2014). „Genová editace CCR5 v autologních CD4 T buňkách osob infikovaných HIV“. The New England Journal of Medicine. 370 (10): 901–10. doi:10.1056 / NEJMoa1300662. PMC 4084652. PMID 24597865.
- ^ A b Lee HJ, Kim E, Kim JS (leden 2010). „Cílené chromozomální delece v lidských buňkách pomocí nukleáz se zinkovým prstem“. Výzkum genomu. 20 (1): 81–9. doi:10.1101 / gr.099747.109. PMC 2798833. PMID 19952142.
- ^ Lee HJ, Kweon J, Kim E, Kim S, Kim JS (March 2012). "Targeted chromosomal duplications and inversions in the human genome using zinc finger nucleases". Výzkum genomu. 22 (3): 539–48. doi:10.1101/gr.129635.111. PMC 3290789. PMID 22183967.
- ^ Boring L, Gosling J, Cleary M, Charo IF (August 1998). "Decreased lesion formation in CCR2-/- mice reveals a role for chemokines in the initiation of atherosclerosis". Příroda. 394 (6696): 894–7. doi:10.1038/29788. PMID 9732872.
- ^ El Khoury J, Toft M, Hickman SE, Means TK, Terada K, Geula C, Luster AD (April 2007). "Ccr2 deficiency impairs microglial accumulation and accelerates progression of Alzheimer-like disease". Přírodní medicína. 13 (4): 432–8. doi:10.1038/nm1555. PMID 17351623.
- ^ Champer J, Buchman A, Akbari OS (March 2016). "Cheating evolution: engineering gene drives to manipulate the fate of wild populations". Genetika hodnocení přírody. 17 (3): 146–59. doi:10.1038/nrg.2015.34. PMID 26875679.
- ^ Webber BL, Raghu S, Edwards OR (August 2015). "Opinion: Is CRISPR-based gene drive a biocontrol silver bullet or global conservation threat?". Sborník Národní akademie věd Spojených států amerických. 112 (34): 10565–7. doi:10.1073/pnas.1514258112. PMC 4553820. PMID 26272924.
- ^ Schaefer KA, Wu WH, Colgan DF, Tsang SH, Bassuk AG, Mahajan VB (May 2017). "Unexpected mutations after CRISPR-Cas9 editing in vivo". Přírodní metody. 14 (6): 547–548. doi:10.1038/nmeth.4293. PMC 5796662. PMID 28557981.
- ^ Wu WH, Tsai YT, Justus S, Lee TT, Zhang L, Lin CS, Bassuk AG, Mahajan VB, Tsang SH (August 2016). "CRISPR Repair Reveals Causative Mutation in a Preclinical Model of Retinitis Pigmentosa". Molekulární terapie. 24 (8): 1388–94. doi:10.1038/mt.2016.107. PMC 5023380. PMID 27203441.
- ^ A b Wilson CJ, Fennell T, Bothmer A, Maeder ML, Reyon D, Cotta-Ramusino C, Fernandez CA, Marco E, Barrera LA (2017-07-10). "The experimental design and data interpretation in 'Unexpected mutations after CRISPR Cas9 editing in vivo' by Schaefer et al. are insufficient to support the conclusions drawn by the authors". bioRxiv: 153338. doi:10.1101/153338.
- ^ Nakajima K, Kazuno AA, Kelsoe J, Nakanishi M, Takumi T, Kato T (October 2016). "Exome sequencing in the knockin mice generated using the CRISPR/Cas system". Vědecké zprávy. 6: 34703. doi:10.1038/srep34703. PMC 5048150. PMID 27698470.
