Dlouho rozptýlený jaderný prvek - Long interspersed nuclear element
| Dlouho rozptýlený jaderný prvek | |
|---|---|
 Krystalová struktura cílící endonukleázy lidského LINE-1 retrotransposonu | |
| Identifikátory | |
| Symbol | ČÁRA |
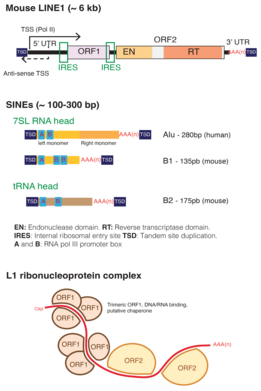
Dlouho rozptýlené jaderné prvky (ŘÁDKY)[1] (také známý jako dlouhé proložené nukleotidové prvky[2] nebo dlouho proložené prvky[3]) jsou skupinou non-LTR (dlouhé opakování terminálu ) retrotranspozony které jsou rozšířené v genomu mnoha lidí eukaryoty.[4][5] Tvoří přibližně 21,1% lidský genom.[6][7][8] LINE tvoří rodinu transpozice, kde každá LINE je asi 7 000 základní páry dlouho. ŘÁDKY jsou přepsal do mRNA a přeloženo do protein který funguje jako reverzní transkriptáza. Reverzní transkriptáza vytváří DNA kopii LINE RNA, kterou lze integrovat do genomu na novém místě.
Jedinou hojnou linií u lidí je LINE1. Lidský genom obsahuje odhadem 100 000 zkrácených a 4 000 prvků LINE-1 v plné délce.[9] Kvůli hromadění náhodných mutací se sekvence mnoha LINE degenerovala do té míry, že již nejsou přepsány nebo přeloženy. K datu inzerce transpozonu v genomu lze použít srovnání sekvencí DNA LINE.
Historie objevů
První popis sekvence odvozené od LINE přibližně 6,4 kb publikoval J. Adams et al. v roce 1980.[10]
Typy
Na základě strukturních rysů a fylogeneze jeho klíčového enzymu, reverzní transkriptáza (RT), ŘÁDKY jsou seskupeny do pěti hlavních skupin zvaných L1, RTE, R2, I a Jockey, které lze rozdělit na nejméně 28 kladů.[11](obr. 1)
V rostlinných genomech byly dosud hlášeny pouze LINE kladu L1 a RTE.[12][13][14] Zatímco prvky L1 se diverzifikují do několika subclades, LINE typu RTE jsou vysoce konzervované, často tvoří jednu rodinu.[15][16]
U hub byly identifikovány prvky podobné Tad, L1, CRE, Deceiver a Inkcap,[17] s prvky podobnými Tadu, které se objevují výlučně v genomech hub.[18]
Všechny LINE kódují alespoň jeden protein ORF2, který obsahuje doménu RT a endonukleázu (EN), buď N-terminál OPICE nebo C-terminál RLE nebo zřídka obojí. A ribonukleáza H doména je občas přítomna. Kromě evolučních starověkých superrodin R2 a RTE kódují LINE obvykle další protein s názvem ORF1, který může obsahovat Gag-koleno, a RRM podobný L1 (InterPro: IPR035300 ), a / nebo esteráza. LINE prvky jsou ve srovnání s relativně vzácné LTR-retrotranspozony v rostlinách, houbách nebo hmyzu, ale jsou dominantní u obratlovců a zejména u savců, kde představují přibližně 20% genomu.[11](obr. 1)
Prvek L1
The LINE-1 / L1 -element je jedním z prvků, které jsou v lidském genomu stále aktivní dodnes. Vyskytuje se ve všech savci[19] až na megabaty.[20]
Další prvky
Zbytky prvků L2 a L3 se nacházejí v lidském genomu.[8] Odhaduje se, že prvky L2 a L3 byly aktivní před ~ 200–300 miliony let. Na rozdíl od prvků L1 chybí prvky L2 doprovodné duplikace cílového místa.[21] Prvky L2 (a L3) jsou ve stejné skupině jako clade CR1, Jockey.[22]
Incidence
V člověku
V prvním návrhu lidského genomu byl zlomek LINE prvků lidského genomu uveden jako 21% a počet jejich kopií jako 850 000. Z nich, L1 Prvky L2 a L3 tvořily 516 000, 315 000 a 37 000 kopií. Neautonomní SINUS prvky, na kterých závisí L1 prvky pro jejich množení tvoří 13% lidského genomu a mají počet kopií přibližně 1,5 milionu.[8] Pravděpodobně pocházeli z rodiny RTE LINE.[23] Nedávné odhady ukazují, že typický lidský genom obsahuje v průměru 100 elementů L1 s potenciálem mobilizace, avšak existuje značné množství variací a někteří jedinci mohou obsahovat větší počet aktivních prvků L1, což činí tyto jedince náchylnějšími k mutagenezi indukované L1.[24]
Zvýšené L1 počty kopií byly také nalezeny v mozcích lidí se schizofrenií, což naznačuje, že prvky LINE mohou hrát roli v některých neuronových onemocněních.[25]

Propagace
LINE prvky se šíří takzvaným mechanismem cíleného reverzního transkripce (TPRT), který byl poprvé popsán pro R2 prvek z bource morušového Bombyx mori.
ORF2 (a ORF1, pokud jsou přítomny) proteiny se primárně sdružují v cis s jejich kódováním mRNA, tvořící a ribonukleoprotein (RNP) komplex, pravděpodobně složený ze dvou ORF2 a neznámého počtu ORF1 trimerů.[26] Komplex je transportován zpět do jádro, kde doména endonukleázy ORF2 otevírá DNA (u TTAAAA hexanukleotidových motivů u savců[27]). Skupina 3'OH je tedy uvolněna pro reverzní transkriptázu k primární reverzní transkripci transkriptu LINE RNA. Po reverzní transkripci je cílové vlákno štěpeno a nově vytvořeno cDNA je integrován[28]
Nové inzerce vytvářejí krátké TSD a většina nových inzertů je silně zkrácena na 5’ (průměrná velikost inzertu u lidí 900pb) a často invertována (Szak et al., 2002). Protože chybí jejich 5’UTR, většina nových vložek je nefunkčních.
Regulace LINE aktivity
Ukázalo se, že hostitelské buňky regulují aktivitu retrotranspozice L1, například prostřednictvím epigenetického umlčování. RNA interference (RNAi) mechanismus malé interferující RNA odvozený od L1 sekvence mohou způsobit potlačení L1 retrotranspozice.[29]
V rostlinných genomech může epigenetická modifikace LINE vést ke změnám exprese blízkých genů a dokonce k fenotypovým změnám: V genomu palmového oleje je methylace LINE typu Karma základem somaklonální „plášťové“ varianty této rostliny, která je zodpovědná za ztráta výnosu.[30]
Člověk APOBEC3C bylo hlášeno zprostředkované omezení prvků LINE-1 a je to kvůli interakci mezi A3C s ORF1p, která ovlivňuje aktivitu reverzní transkriptázy.[31]
Souvislost s chorobami
Historickým příkladem choroby způsobené L1 je hemofilie A, která je způsobena inzerční mutageneze.[32] Existuje téměř 100 příkladů známých onemocnění způsobených vložením retroelementu, včetně některých typů rakoviny a neurologických poruch.[33] Korelace mezi L1 mobilizace a onkogeneze byla hlášena u rakoviny epiteliálních buněk (karcinom ).[34] Hypomethylace LINES je spojena s nestabilitou chromozomů a změnou genové exprese[35] a nachází se v různých typech rakovinných buněk v různých typech tkání.[36][35] Hypomethylace specifické L1 lokalizované v genu MET onco je spojena s tumorogenezí rakoviny močového měchýře,[37] Porucha spánku na směny[38] je spojena se zvýšeným rizikem rakoviny, protože se snižuje noční expozice melatonin, hormon, u kterého bylo prokázáno, že snižuje indukci L1 nestabilita genomu.[39]
Reference
- ^ Ewing AD, Kazazian HH (červen 2011). „Resekvenování celého genomu umožňuje detekci mnoha vzácných inzertních alel LINE-1 u lidí“. Výzkum genomu. 21 (6): 985–90. doi:10,1101 / gr. 114777,110. PMC 3106331. PMID 20980553.
- ^ Huang X, Su G, Wang Z, Shangguan S, Cui X, Zhu J a kol. (Březen 2014). „Hypomethylace dlouho rozptýleného nukleotidového prvku-1 v periferních mononukleárních buňkách juvenilních pacientů se systémovým lupus erythematodes v Číně“. International Journal of Reumatic Diseases. 17 (3): 280–90. doi:10.1111 / 1756-185X.12239. PMID 24330152.
- ^ Rodić N, Burns KH (březen 2013). „Long interspersed element-1 (LINE-1): cestující nebo řidič v lidských novotvarech?“. Genetika PLOS. 9 (3): e1003402. doi:10.1371 / journal.pgen.1003402. PMC 3610623. PMID 23555307.
- ^ Singer MF (březen 1982). "SINE a LINE: vysoce opakované krátké a dlouhé rozptýlené sekvence v savčích genomech". Buňka. 28 (3): 433–4. doi:10.1016/0092-8674(82)90194-5. PMID 6280868.
- ^ Jurka, J. (1998). "Opakování v genomové DNA: Těžba a význam". Aktuální názor na strukturní biologii. 8 (3): 333–337. doi:10.1016 / S0959-440X (98) 80067-5. PMID 9666329.
- ^ Lindblad-Toh K, Wade CM, Mikkelsen TS, Karlsson EK, Jaffe DB, Kamal M a kol. (Prosinec 2005). "Sekvence genomu, komparativní analýza a haplotypová struktura domácího psa". Příroda. 438 (7069): 803–19. Bibcode:2005 Natur.438..803L. doi:10.1038 / příroda04338. PMID 16341006.
- ^ Schumann GG, Gogvadze EV, Osanai-Futahashi M, Kuroki A, Münk C, Fujiwara H a kol. (01.01.2010). Unikátní funkce opakujících se transkriptomů. International Review of Cell and Molecular Biology. 285. str. 115–88. doi:10.1016 / B978-0-12-381047-2.00003-7. ISBN 9780123810472. PMID 21035099.
- ^ A b C Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J a kol. (Únor 2001). „Počáteční sekvenování a analýza lidského genomu“ (PDF). Příroda. 409 (6822): 860–921. Bibcode:2001 Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Sheen FM, Sherry ST, Risch GM, Robichaux M, Nasidze I, Stoneking M a kol. (Říjen 2000). „Čtení mezi ČÁRAMI: lidská genomická variace vyvolaná retrotranspozicí LINE-1“. Výzkum genomu. 10 (10): 1496–508. doi:10,1101 / gr.149400. PMC 310943. PMID 11042149.
- ^ Adams JW, Kaufman RE, Kretschmer PJ, Harrison M, Nienhuis AW (prosinec 1980). „Rodina dlouho opakovaných sekvencí DNA, z nichž jedna kopie je vedle lidského genu beta-globinu“. Výzkum nukleových kyselin. 8 (24): 6113–28. doi:10.1093 / nar / 8.24.6113. PMC 328076. PMID 6258162.
- ^ A b Kapitonov VV, Tempel S, Jurka J (prosinec 2009). „Jednoduchá a rychlá klasifikace retrotranspozonů jiných než LTR na základě fylogeneze jejich proteinových sekvencí domény RT“. Gen. 448 (2): 207–13. doi:10.1016 / j.gene.2009.07.019. PMC 2829327. PMID 19651192.
- ^ Heitkam T, Schmidt T (září 2009). „BNR - rodina LINE z Beta vulgaris - obsahuje doménu RRM v otevřeném čtecím rámci 1 a definuje subkladu L1 přítomnou v různých rostlinných genomech“. The Plant Journal. 59 (6): 872–82. doi:10.1111 / j.1365-313x.2009.03923.x. PMID 19473321.
- ^ Zupunski V, Gubensek F, Kordis D (říjen 2001). „Evoluční dynamika a evoluční historie v RTE kladu retrotranspozonů jiných než LTR“. Molekulární biologie a evoluce. 18 (10): 1849–63. doi:10.1093 / oxfordjournals.molbev.a003727. PMID 11557792.
- ^ Komatsu M, Shimamoto K, Kyozuka J (srpen 2003). „Dvoustupňová regulace a kontinuální retrotranspozice rýže typu LINE retrotransposon Karma“. Rostlinná buňka. 15 (8): 1934–44. doi:10.1105 / tpc.011809. PMC 167180. PMID 12897263.
- ^ Heitkam T, Holtgräwe D, Dohm JC, Minoche AE, Himmelbauer H, Weisshaar B a kol. (Srpen 2014). „Profilování značně diverzifikovaných rostlinných linií odhaluje odlišné subclady specifické pro rostliny“. The Plant Journal. 79 (3): 385–97. doi:10.1111 / tpj.12565. PMID 24862340.
- ^ Smyshlyaev G, Voigt F, Blinov A, Barabas O, Novikova O (prosinec 2013). „Akvizice H-domény ribonukleázy podobné archaei rostlinami L1 retrotransposony podporuje modulární evoluci“. Sborník Národní akademie věd Spojených států amerických. 110 (50): 20140–5. Bibcode:2013PNAS..11020140S. doi:10.1073 / pnas.1310958110. PMC 3864347. PMID 24277848.
- ^ Novikova O, Fet V, Blinov A (únor 2009). "Retrotranspozony jiné než LTR v houbách". Funkční a integrační genomika. 9 (1): 27–42. doi:10.1007 / s10142-008-0093-8. PMID 18677522.
- ^ Malik HS, Burke WD, Eickbush TH (červen 1999). „Věk a vývoj retrotransponovatelných prvků jiných než LTR“. Molekulární biologie a evoluce. 16 (6): 793–805. doi:10.1093 / oxfordjournals.molbev.a026164. PMID 10368957.
- ^ Warren WC, Hillier LW, Marshall Graves JA, Birney E, Ponting CP Grützner F a kol. (Květen 2008). „Analýza genomu ptakopysků odhaluje jedinečné podpisy evoluce“. Příroda. 453 (7192): 175–83. Bibcode:2008 Natur.453..175 W.. doi:10.1038 / nature06936. PMC 2803040. PMID 18464734.
- ^ Smith JD, Gregory TR (červen 2009). „Velikosti genomu megabatů (Chiroptera: Pteropodidae) jsou pozoruhodně omezené“. Biologické dopisy. 5 (3): 347–51. doi:10.1098 / rsbl.2009.0016. PMC 2679926. PMID 19324635.
- ^ Kapitonov VV, Pavlicek A, Jurka J (01.01.2006). Antologie lidské repetitivní DNA. Wiley-VCH Verlag GmbH & Co. KGaA. doi:10.1002 / 3527600906.mcb.200300166. ISBN 9783527600908.
- ^ Lovsin N, Gubensek F, Kordi D (prosinec 2001). „Evoluční dynamika v nové kladu L2 retrotranspozonů jiných než LTR v Deuterostomii“. Molekulární biologie a evoluce. 18 (12): 2213–24. doi:10.1093 / oxfordjournals.molbev.a003768. PMID 11719571.
- ^ Malik, HS; Eickbush, TH (září 1998). „Třída RTE retrotranspozonů jiných než LTR je široce distribuována u zvířat a je původem mnoha SINE“. Molekulární biologie a evoluce. 15 (9): 1123–34. doi:10.1093 / oxfordjournals.molbev.a026020. PMID 9729877.
- ^ Streva VA, Jordan VE, Linker S, Hedges DJ, Batzer MA, Deininger PL (březen 2015). „Sekvenování, identifikace a mapování aktivovaných prvků L1 (SIMPLE) odhaluje významné rozdíly v prvcích L1 v plné délce mezi jednotlivci“. BMC Genomics. 16 (220): 220. doi:10.1186 / s12864-015-1374-r. PMC 4381410. PMID 25887476.
- ^ Bundo M, Toyoshima M, Okada Y, Akamatsu W, Ueda J, Nemoto-Miyauchi T a kol. (Leden 2014). „Zvýšená retrotranspozice l1 v neuronovém genomu u schizofrenie“. Neuron. 81 (2): 306–13. doi:10.1016 / j.neuron.2013.10.053. PMID 24389010.
- ^ Babushok DV, Ostertag EM, Courtney CE, Choi JM, Kazazian HH (únor 2006). „Integrace L1 v modelu transgenní myši“. Výzkum genomu. 16 (2): 240–50. doi:10,1101 / gr. 4571606. PMC 1361720. PMID 16365384.
- ^ Jurka J (březen 1997). „Sekvenční vzorce naznačují enzymatické zapojení do integrace savčích retroposonů“. Sborník Národní akademie věd Spojených států amerických. 94 (5): 1872–7. Bibcode:1997PNAS ... 94.1872J. doi:10.1073 / pnas.94.5.1872. PMC 20010. PMID 9050872.
- ^ Luan DD, Korman MH, Jakubczak JL, Eickbush TH (únor 1993). „Reverzní transkripce R2Bm RNA je aktivována nickem v chromozomálním cílovém místě: mechanismus pro retrotranspozici bez LTR“. Buňka. 72 (4): 595–605. doi:10.1016/0092-8674(93)90078-5. PMID 7679954.
- ^ Yang N, Kazazian HH (září 2006). „L1 retrotranspozice je potlačena endogenně kódovanými malými interferujícími RNA v lidských kultivovaných buňkách“. Přírodní strukturní a molekulární biologie. 13 (9): 763–71. doi:10.1038 / nsmb1141. PMID 16936727.
- ^ Ong-Abdullah M, Ordway JM, Jiang N, Ooi SE, Kok SY, Sarpan N, et al. (Září 2015). „Ztráta metylace transpozonů karmy je základem plášťové somaklonální varianty olejové palmy“. Příroda. 525 (7570): 533–7. Bibcode:2015 Natur.525..533O. doi:10.1038 / příroda15365. PMC 4857894. PMID 26352475.
- ^ Horn AV, Klawitter S, Held U, Berger A, Vasudevan AA, Bock A, et al. (Leden 2014). „Omezení lidské LINE-1 pomocí APOBEC3C je nezávislé na deamináze a je zprostředkováno interakcí ORF1p, která ovlivňuje aktivitu reverzní transkriptázy LINE.“. Výzkum nukleových kyselin. 42 (1): 396–416. doi:10.1093 / nar / gkt898. PMC 3874205. PMID 24101588.
- ^ Kazazian HH, Wong C, Youssoufian H, Scott AF, Phillips DG, Antonarakis SE (březen 1988). „Hemofilie A vyplývající z de novo inzerce sekvencí L1 představuje nový mechanismus mutace u člověka“. Příroda. 332 (6160): 164–6. Bibcode:1988Natur.332..164K. doi:10.1038 / 332164a0. PMID 2831458.
- ^ Solyom S, Kazazian HH (únor 2012). „Mobilní prvky v lidském genomu: důsledky pro nemoc“. Genomová medicína. 4 (2): 12. doi:10,1186 / gm311. PMC 3392758. PMID 22364178.
- ^ Carreira PE, Richardson SR, Faulkner GJ (leden 2014). „L1 retrotranspozony, rakovinové kmenové buňky a onkogeneze“. FEBS Journal. 281 (1): 63–73. doi:10.1111 / febs.12601. PMC 4160015. PMID 24286172.
- ^ A b Kitkumthorn N, Mutirangura A (srpen 2011). „Dlouho rozptýlená hypomethylace jaderného prvku-1 v rakovině: biologie a klinické aplikace“. Klinická epigenetika. 2 (2): 315–30. doi:10.1007 / s13148-011-0032-8. PMC 3365388. PMID 22704344.
- ^ Estécio MR, Gharibyan V, Shen L, Ibrahim AE, Doshi K, He R, Jelinek J, Yang AS, Yan PS, Huang TH, Tajara EH, Issa JP (květen 2007). „Hypomethylace LINE-1 u rakoviny je vysoce variabilní a nepřímo koreluje s nestabilitou mikrosatelitu“. PLOS ONE. 2 (5): e399. Bibcode:2007PLoSO ... 2..399E. doi:10,1371 / journal.pone.0000399. PMC 1851990. PMID 17476321.
- ^ Wolff EM, Byun HM, Han HF, Sharma S, Nichols PW, Siegmund KD a kol. (Duben 2010). „Hypomethylace promotoru LINE-1 aktivuje alternativní transkript onkogenu MET v močových měchýřích s rakovinou“. Genetika PLOS. 6 (4): e1000917. doi:10.1371 / journal.pgen.1000917. PMC 2858672. PMID 20421991.
- ^ Spadafora C (duben 2015). „Regulační mechanismus závislý na reverzní transkriptáze závislý na LINE-1 je aktivní v embryogenezi a tumorigenezi“. Annals of the New York Academy of Sciences. 1341 (1): 164–71. Bibcode:2015NYASA1341..164S. doi:10.1111 / nyas.12637. PMID 25586649.
- ^ deHaro D, Kines KJ, Sokolowski M, Dauchy RT, Streva VA, Hill SM a kol. (Červenec 2014). „Regulace exprese L1 a retrotranspozice melatoninem a jeho receptorem: důsledky pro riziko rakoviny spojené s expozicí světla v noci“. Výzkum nukleových kyselin. 42 (12): 7694–707. doi:10.1093 / nar / gku503. PMC 4081101. PMID 24914052.
