Hybridní genom eukaryotu - Eukaryote hybrid genome - Wikipedia
| Glosář | |
|---|---|
|
Hybridní genomy eukaryot výsledkem mezidruhová hybridizace, kde úzce souvisí druh pářit se a plodit potomky smíchané genomy. Ukázal to příchod rozsáhlého genomového sekvenování hybridizace je běžné a že může představovat důležitý zdroj románu variace. Ačkoli většina mezidruhových hybridů je sterilní nebo méně vhodné než jejich rodiče, někteří mohou přežít a rozmnožovat se, což umožňuje převod adaptivních variant přes hranice druhů a dokonce vést k tvorbě nových evoluční linie. Existují dvě hlavní varianty hybridních genomů druhů: alopolyploid, které mají jeden plný chromozomová sada - od každého rodičovského druhu a - homoploidní, která oblast mozaika genomů rodičovských druhů bez zvýšení počtu chromozomů. Vytvoření hybridních druhů vyžaduje vývoj reprodukční izolace proti rodičovským druhům. Allopolyploidní druhy mají často silné vnitřní reprodukční bariéry kvůli rozdílům v chromozóm a homoploidní hybridy se mohou reprodukčně izolovat od mateřského druhu prostřednictvím sortimentu genetických inkompatibilit. Oba typy hybridů se však mohou dále reproduktivně izolovat a získat vnější izolační bariéry využitím nových ekologické výklenky ve vztahu k jejich rodičům. Hybridy představují sloučení odlišných genomů a čelí tak problémům vyplývajícím z nekompatibilních kombinací genů. Hybridní genomy jsou tedy vysoce dynamické a mohou podstoupit rychlou evoluční změnu, včetně stabilizace genomu ve kterém selekce proti nekompatibilním kombinacím vede k fixace kompatibilní blok předků kombinace v rámci hybridních druhů. Potenciál rychlého přizpůsobování nebo speciace dělá z hybridních genomů obzvláště vzrušující téma evoluční biologie. Článek shrnuje jak introgresní alely nebo hybridní druhy mohou stanovit a jak se výsledné hybridní genomy vyvíjejí.
Pozadí
Genetická výměna mezi druhy může bránit vývoji biodiverzity, protože tok genů mezi odlišnými druhy působí proti jejich diferenciaci a hybridizace mezi nedávno odlišnými druhy může vést ke ztrátě genetiky úpravy nebo fúze druhů.[1] Tradičně, zoologové pohlíželi na mezidruhovou hybridizaci jako na maladaptivní chování[2] což může vést k rozpadu společně adaptovaného genové komplexy.[3] Naproti tomu rostlinní biologové brzy uznali, že hybridizace může být někdy důležitou evoluční silou, která přispívá ke zvyšování biologické rozmanitosti.[4][5] V poslední době se hromadí důkazy ukazující, že hybridizace je také důležitým evolučním procesem u zvířat.[1][6][7] Mezidruhová hybridizace může obohatit genetickou rozmanitost introgrovaného taxonu, vést k introgrese prospěšné genetické variace nebo dokonce generovat nové hybridní druhy.[1] Hybridizace je nyní také známa tím, že přispívá k evolučnímu potenciálu v několika učebnicových příkladech adaptivní záření, včetně Geospiza Galapágy pěnkavy,[8] Afričan cichlida Ryby,[9] Heliconius motýli[10][11][12] a havajské Madiinae tarweeds a stříbrná slova.[13] Tento článek shrnuje vývojové výsledky mezidruhové hybridizace a vlastnosti genomů hybridních genomů. Mnoho diskutovaných témat se vztahuje také na hybridizaci mezi různými poddruhy nebo populacemi stejného druhu, ale tento článek se zaměřuje na mezidruhovou hybridizaci (v tomto přehledu označovanou jako hybridizace).
Evoluční výsledky
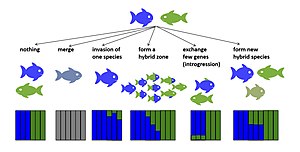
Existuje několik potenciálních evolučních výsledků hybridizace. Pokud hybridy rané generace nejsou životaschopné nebo sterilní, může hybridizace snížit reprodukční úspěch rodičovských druhů.[14][15] To by mohlo potenciálně vést k výztuž, výběr k posílení premating izolace[16] nebo pokud se u druhů nepodaří vyvinout premating izolaci, mohlo by to zvýšit jejich riziko vyhynutí v důsledku promarněného reprodukčního úsilí.[14] Pokud zdatnost hybridů rané generace je nenulová a u některých hybridů pozdější generace je stejně vysoká nebo dokonce vyšší než vhodnost jednoho nebo obou mateřských taxonů, hybridy mohou přemístit mateřské taxony a hybridizující taxony se mohou spojit (obrácení specie[17][18]). Pokud je vhodnost hybridů rané generace snížena, ale nenulová, mohou se v kontaktní zóně taxonů objevit hybridní zóny.[19] Pokud jsou hybridy plodné, může hybridizace přispět novými variacemi prostřednictvím vzácných hybridů zpětný kříž s rodičovskými druhy. Taková introgresivní hybridizace může umožnit neutrální nebo selektivně prospěšné alely přenášet přes hranice druhů, a to i v párech druhů, které zůstávají zřetelné i přes příležitost tok genů.[20][21] Hybridní zdatnost se může lišit s dobou odchylky mezi hybridizujícími taxony. Tento vzor byl prokázán pro různé taxony včetně Drosophila,[22] ptactvo[23] a ryby.[24] Hybridní fitness se může také lišit v příčném směru,[25] mezi hybridy první generace a pozdější generace,[26] a mezi jednotlivci v rámci generací stejného křížového typu.[27][28] V některých případech se hybridy mohou vyvinout do nových hybridních druhů s reprodukční izolace na oba mateřské taxony.[29][30] Níže jsou popsány evoluční výsledky hybridizace, které vedou k trvalým hybridním genomům.
Adaptivní introgrese
Když mohou být vzácné hybridy zpětně kříženy s rodičovskými alelami kódujícími vlastnosti, které jsou prospěšné pro oba rodičovské druhy, mohou být přeneseny přes hranice druhů, i když rodičovské druhy zůstávají odlišnými taxony. Tento proces se označuje jako adaptivní introgrese (poněkud zavádějící termín, protože zpětné křížení samo o sobě nemusí být adaptivní, ale některé introgresivní varianty mohou být prospěšné[1]). Simulace naznačují, že adaptivní introgrese je možná, pokud není podstatně snížena hybridní kondice,[31][32] nebo adaptivní loci jsou úzce spojeny se škodlivými.[33] Příklady adaptivních znaků, které byly přeneseny prostřednictvím introgrese, zahrnují gen rezistence vůči insekticidu, ze kterého byl přenesen Anopheles gambiae na A. coluzzii[21] a znak zabarvení červeného výstražného křídla Heliconius motýly, které jsou v přirozeném výběru od predátorů, které byly introgresovány např. H. melpomene na H. timareta[34] a další Heliconius druh.[20] V rostlině Arabidopsis arenosa některé alely způsobující adaptaci na sucho a fytotoxický úrovně kovu byly proniknuty dovnitř A. lyrata.[35] Dokonce i u lidí existují důkazy o adaptivní introgrese např. alely imunity, alely pigmentace kůže a alely způsobující adaptaci na prostředí ve vysokých nadmořských výškách od neandertálců a denisovanů.[36] Pokud se vlastnosti důležité pro rozpoznávání druhů nebo reprodukční izolaci dostanou do populace jiného druhu, může se introgresovaná populace stát reprodukčně izolovanou proti ostatním populacím stejného druhu. Mezi příklady patří Heliconius motýly, kde dochází k selektivní introgrese genů vzoru křídla mezi odlišnými liniemi,[37] a křídlové vzory přispívají k reprodukční izolaci u párů některých druhů s nízkou (např. mezi H. t. florencia a H. t. linaresi) a střední úrovně (např. H. c. Galantus/H. pachinus) divergence.[38]
Detekce a studium pomocí genomických nástrojů
Mnoho empirických případových studií začíná průzkumnou detekcí domnělých hybridních taxonů nebo jedinců s přístupy genomického shlukování, jako jsou ty, které se používají v softwarové STRUKTURĚ,[39] PŘÍSADA[40] nebo jemná KONSTRUKCE.[41] Tyto metody odvozují z dat uživatelem zadaný počet genetických skupin a přiřazují každého jednotlivce k jedné nebo ke směsi těchto skupin. Mohou být použity na blízce příbuzné taxony, aniž by bylo nutné předem určovat jednotlivce na taxony, a mohou tak být zvláště užitečné při studiu blízce příbuzných taxonů nebo komplexů druhů. Nerovnoměrný odběr vzorků rodičovských taxonů nebo rozdílné množství driftu v zahrnutých taxonech však může vést k mylným závěrům ohledně důkazů o hybridizaci.[42]
Pokud jsou k dispozici genomová data více druhů, mohou být pro identifikaci introgrese vhodnější fylogenetické metody. Introgresivní hybridizace vede ke genovým stromům, které jsou nesouhlasné s druhovým stromem, přičemž introgresivní jedinci jsou fylogeneticky blíže ke zdroji introgrese než ke svým neintrogrovaným stejným druhům. Takové nesouladné genové stromy mohou také vzniknout náhodou neúplným tříděním řádků, zvláště pokud jsou porovnávané druhy stále mladé. Diskordantní genové stromy jsou proto pouze důkazem introgrese, pokud je genový strom produkovaný nadměrným sdílením alel mezi hybridizujícími taxony silně nadměrně zastoupen ve srovnání s alternativními nesouhlasnými genovými stromy. K detekci takového nadměrného sdílení alel mezi hybridizujícími taxony byla vyvinuta celá sada metod, včetně Pattersonových statistik D nebo testů ABBA-BABA[43][44][45] nebo f-statistiky.[46][47] Upravené verze těchto testů lze použít k odvození introgresních genomových oblastí,[48] směr toku genů[49][50] nebo množství toku genů.[47]
U souborů dat s velkým počtem taxonů může být obtížné vypočítat všechny možné testy hybridizace. V takových případech mohou být vhodnější metody konstrukce grafů.[51][52][53] Tyto metody rekonstruují složité fylogenetické modely s hybridizací, které nejlépe odpovídají genetickým vztahům mezi odebranými taxony a poskytují odhady driftu a introgrese. Mohou také pomoci další metody fylogenetické sítě, které zohledňují neúplné třídění a hybridizaci linií.[54][55] Metody založené na rozkladu nerovnováhy vazeb nebo metody odvozující trakty předků lze použít k dnešnímu dni nedávných událostí příměsí nebo introgrese, protože trakty předků jsou průběžně rozděleny rekombinací.[52][56][57][58][59] Se zvyšující se stabilizací genomu by se jednotlivci měli u místních předků lišit méně. Úrovně stabilizace genomu lze tedy vyhodnotit výpočtem proporcí předků (např. Pomocí fd) v genomických oknech a testováním, zda tyto korelace napříč jednotlivci. Kromě toho, pokud hybridizace stále probíhá, proporce předků by se měly u jednotlivců a ve vesmíru lišit.
Jiným přístupem je použití demografického modelování k nalezení zjednodušení evoluční historie studovaných taxonů.[60] Demografické modelování by mělo být použito pouze na malé sady taxonů, protože s rostoucím počtem modelů se zvyšuje složitost modelu a počet parametrů modelu, jako je načasování, množství a směr toku genů, a velikost populace a mezičasy mohou být rychle příliš vysoké. Vhodnost demografických modelů na data lze posoudit pomocí frekvenčního spektra místa[61][62] nebo se souhrnnými statistikami v rámci přibližného bayesovského výpočtu.[63] Je také možné získat více energie kombinací informací z vazebných nerovnovážných rozpadových vzorců a frekvenčního spektra alely.[64]
Definice hybridních druhů
Jedním z potenciálních evolučních výsledků hybridizace je vytvoření nové, reprodukčně izolované linie, tj. Hybridní speciace.[1][29] Hybridní druh má přimíchaný genom a vytváří stabilní geneticky odlišné populace.[29] Někteří vědci tvrdí, že důkazy o základu reprodukční izolace odvozené z hybridizace by měly být dalším určujícím kritériem pro hybridní speciaci,[65] ale uvidíš.[66] Tato přísnější definice zahrnuje polyploidní hybridní taxony, ale zahrnuje jen několik dobře studovaných případů homoploidní hybridní specie, např. Heliconius heurippa,[10][11][12] Passer italiae,[28] a tři Helianthus druh slunečnice[67] protože u většiny doporučených příkladů homoploidní hybridní specie je genetický základ reprodukční izolace stále neznámý.[65]
Hybridní druhy mohou zabírat ekologickou niku odlišnou od rodičovských druhů a mohou být izolovány od rodičovských druhů primárně prostřednictvím pre-páření překážek (hybridní speciace s vnějšími překážkami)[68]). Hybridní druhy mohou být také reprodukčně izolovány z rodičovských druhů prostřednictvím třídění nekompatibilit vedoucích k novým kombinacím rodičovských alel, které jsou nekompatibilní s oběma rodičovskými druhy, ale kompatibilní s hybridním taxonem (rekombinační hybridní speciace).[29] Rekombinační hybridní taxon má typicky také podstatnou část genomu odvozeného od dárce introgresního materiálu, i když existují variace jak mezi taxony, tak v rámci linií hybridních taxonů.[69][70]
Homoploidní a polyploidní hybridní speciace
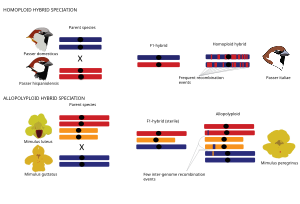
Obecně platí, že hybridní druhy mohou vzniknout ze dvou hlavních typů hybridní specie, definované tím, zda je speciační událost spojena s duplikací genomu (polyploidie ) nebo ne. Homoploidní hybridní speciace Homoploidní hybridní speciace je definována jako evoluce nového hybridního druhu s reprodukční izolací k oběma rodičovským taxonům beze změny ploidie, tj. počtu sad chromozomů.[1] Genomy homoploidních hybridních druhů jsou mozaiky rodičovských genomů, protože trakty předků z rodičovských druhů jsou rozděleny rekombinace.[66][67][71][72][73][74][75] V případě speciace polyploidního hybridu je hybridizace spojena s duplikací genomu, což vede k alopolyploid se zvýšenou ploidií ve srovnání s jejich rodičovskými taxony. Na rozdíl od alopolyploidů autopolyploidy jsou charakterizovány duplikací genomu u stejného druhu, a proto nejsou dále diskutovány v kontextu tohoto přehledu. Allopolyploidní speciace je častější u rostlin než u zvířat.[76] Polyploidní hybridy lze okamžitě izolovat z jejich rodičovských druhů chromozóm početní rozdíly.[76]
Reprodukční izolace proti původním druhům
Pro úspěšné založení hybridního druhu je nutná dostatečná reprodukční izolace od obou rodičovských druhů.[1][65][77] Reprodukční izolace proti původním druhům je obtížnější dosáhnout u homoploidních hybridů, kde karyotyp rozdíly nepřispívají k vnitřní izolaci. Reprodukční izolace mezi hybridním druhem a jeho rodičovskými druhy může vzniknout z různých reprodukčních bariér buď před nebo po oplodnění (prezygotických nebo postzygotických), které samy o sobě mohou být závislé nebo nezávislé na podmínkách prostředí (vnější nebo vnitřní bariéry).[78] Například vnitřní postzygotické bariéry způsobují hybridní neviditelnost nebo sterilitu bez ohledu na prostředí, ve kterém se vyskytují, zatímco vnější postzygotické bariéry mají za následek hybridy s nízkou kondicí v důsledku maladaptace na konkrétní prostředí.[30]
Ukázalo se také, že prezygotické vnitřní a vnější rozdíly jsou důležité při izolaci hybridů z jejich původních druhů. V rostlinách může být izolace zprostředkovaná opylovačem způsobená změnami květinových charakteristik důležitou vnější prezygotickou ekologickou bariérou.[79][80][81][82] Ukázalo se, že silné vnější preygotické izolují hybridní druhy Senecio eboracensis z jeho mateřských druhů, kde hybridy ve volné přírodě prakticky chybí, ačkoli zlomek hybridních potomků je při laboratorních experimentech plodný.[83] Lowe a Abbott k tomu dospěli selfing, načasování kvetení a postavy zapojené do přitažlivosti opylovače pravděpodobně přispívají k této vnější izolaci.[83] U několika taxonů byla také hlášena izolace řízená preferencemi prezygotických matek generovaná z vnitřního třídění páření mezi hybridy. U afrických cichlíd ryby vykazovaly experimentální hybridy kombinace rodičovských rysů a preferencí, které vedly k tomu, že hybridy se páří převážně s jinými hybridy.[84] Podobný vzor byl nalezen v Geospiza Galapágy pěnkavy, kde specifická hybridní píseň vyplynula z transgresivní morfologie zobáku,[8] a hybridní Heliconius motýli upřednostňovali hybridní vzorování křídel před vzorem obou rodičů.[12] Vnitřní rozdíly v používání stanovišť[85] nebo ve fenologii[86] může mít za následek určitý stupeň reprodukční izolace proti rodičovským druhům, pokud je páření specifické pro čas a stanoviště. Například závod Apple Host v Rhagoletis pomonella červy červů se vyvinuly po introgresi genů souvisejících s diapauzou z mexických mušek altiplano, které umožnily přechod z rodového hostitele hawthorne na později kvetoucí jablko [87][88] a izoloval dva hostitelské rasy pomocí allochronické vnitřní pre-zygotické izolace. v Xiphophorus mečoun ryby silné předky asortativní páření udržovaly hybridní genetický shluk odděleně po 25 generací, ale zmizely za manipulovaných podmínek.[89] Prezygotické reprodukční bariéry toku genů proto mohou záviset na prostředí.
Ukázalo se také, že postzygotické izolační bariéry jsou důležité v různých hybridních liniích. Pracovat na Helianthus slunečnice odhalily, že vnitřní postzygotické látky mohou způsobit reprodukční izolaci proti původním druhům. Postzygotické bariéry spočívají v již existujících strukturálních rozdílech,[73][90] v kombinaci s hybridizací vyvolanými strukturálními rozdíly.[73] Třídění nekompatibilit mezi mateřskými druhy, kde jedna podskupina těchto izoluje hybridní taxon proti jednomu z rodičů a jiná podskupina izoluje jej proti druhému rodiči, má za následek vnitřní postzygotickou izolaci mezi italským vrabcem Passer italiae a jeho rodičovské druhy.[28] Simulační studie ukazují, že pravděpodobnost hybridní speciace prostřednictvím tohoto mechanismu závisí na době divergence mezi mateřskými druhy,[91] velikost populace hybridního druhu,[92] - povaha výběru působícího na hybridy a - vazba mezi vzájemnou nekompatibilitou a adaptivními variantami.[93] Vnější ekologické bariéry proti rodičovským druhům mohou vznikat jako vedlejší produkty ekologické diferenciace, pokud je páření specifické pro čas a / nebo stanoviště. Bylo prokázáno, že hybridní druhy se přizpůsobují novým ekologickým výklenkům transgresivní fenotypy,[85] nebo prostřednictvím nových kombinací ekologických vlastností původních druhů,[94] a ekologická selekce proti kříženým fenotypům rodič-hybrid by vedla k vnější postzygotické izolaci.
Stabilizace
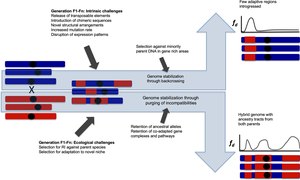
Hybridizace může mít mnoho různých výsledků. Hybridní speciace vede k reprodukční izolaci jak vůči původním druhům, tak proti genomům, které se vyvíjejí nezávisle na genomech původních druhů. Introgresivní hybridizace může přenést důležité nové varianty do genomů druhů, které i přes příležitostný tok genů zůstávají odlišné od ostatních taxonů. V tomto článku jsou oba typy hybridomů odvozených genomů označovány jako persistentní hybridní genomy. Po počáteční hybridizaci jsou introgresní trakty, genetické bloky zděděné od každého mateřského druhu, rozděleny s postupnými generacemi a rekombinačními událostmi. Rekombinace je častější v homoploidních hybridních genomech než v alopolyploidních hybridních genomech. V alopolyploidech rekombinace může destabilizovat karyotyp a vést k aberantu meiotický chování a snížená plodnost, ale může také generovat nové kombinace genů a výhodné fenotypové vlastnosti [95] jako u homoploidních hybridů. Jakmile hybridizace mezi hybridním taxonem a jeho mateřskými taxony skončí, mohou se zafixovat různé bloky předků nebo introgresní trakty, což je proces označovaný jako „stabilizace genomu“.[71] Některé oblasti introgrese jsou odstraněny výběrem proti nekompatibilitě a jiné jsou opraveny. Teoretické modely na hybridních zónách naznačují, že rozpad bloků předků prostřednictvím rekombinace je potlačen poblíž genů poskytujících reprodukční izolaci kvůli nižší kondici rekombinantních hybridů.[96] Síla potlačení je ovlivněna formou výběru, dominance, a zda se lokus nacházel na autosome nebo pohlavní chromozom.[96] Doba do stabilizace genomu je variabilní. Bylo zjištěno, že fixace bloků předků byla u experimentálního hybridu rychlá Helianthus genomy slunečnicových druhů,[97] a stabilizace genomu hybridních druhů slunečnice se odhaduje na stovky generací.[71] v Zymoseptoria genomy hub byly stabilizovány během ca. 400 generací,[98] zatímco v hybridním Xiphophorus genomy mečouna[99] stabilizace genomu byla dosažena až po ca. 2000 a 2500 generací. Několik neandertálských oblastí se zafixovalo v lidských genomech během ca. 2000 generací po hybridizaci,[100] a segregační inkompatibility jsou přítomny v hybridním italském vrabci přibližně 5000 generací po počáteční hybridizační události.[101]
Daný čas, genetický drift nakonec stochasticky zafixuje bloky odvozené od dvou původních druhů v konečných izolovaných hybridních populacích.[71] Výběr proti lokusům nekompatibility může urychlit proces fixace rodičovských alel, protože hybridy, které obsahují alely, u nichž je méně pravděpodobné, že způsobí nekompatibilitu, budou mít vyšší kondici a příznivé alely se rozšíří v populaci. Fixace recesivních slabě škodlivých alel v rodičovských taxonech však může také vést k tomu, že hybridy udrží obě rodičovské alely: protože hybridy s haplotypy od obou rodičů nejsou homozygotní u všech slabě škodlivých alel mají vyšší kondici než hybridy pouze s jedním rodičovským haplotypem. Toto asociativní naddominance,[102][103] může zpomalit proces fixace rodičovských alel upřednostňováním retence obou rodičovských haplotypů. Účinek asociativní naddominance je nejsilnější v oblastech s nízkou rekombinací, včetně inverzí.[104] Rovnováha mezi alelami a alelickými kombinacemi poskytujícími příznivé fenotypové znaky a síla selekce proti nekompatibilitám určují, jaké introgresní trakty budou po hybridizaci zděděny od kterých rodičů.[21][105][106] Oblast odolnosti vůči insekticidu byla zachována po hybridizační události v Anopheles coluzzi,[21] navrhování role výběru při udržování příznivých introgresních regionů. Místní míra rekombinace je důležitý pro pravděpodobnost introgrese, protože v případě rozsáhlých inkompatibilit se introgrované alely pravděpodobně rekombinují mimo inkompatibility v oblastech s vysokou rekombinací. Tento vzor byl zjištěn u opičích květů Mimulus,[107] v Mus domesticus domácí myši,[108] v Heliconius motýli[106] a v Xiphophorus mečoun ryby.[69]
Byly identifikovány nekompatibility celého genomu Xipoforní Ryba,[109] chimérické geny a mutace ortologních genů způsobují inkompatibility v experimentech rané generace Cyprinidae zlaté rybky - hybridy kaprů[110] a shledává se, že klíčovou roli mají mimomukleární nekompatibility, např. v italských vrabcích,[75][111] houba[112] a cyto-nukleární nekompatibility v Mimulus rostliny.[113] Důkazy ze změněných vzorců exprese u syntetických hybridů a chybějící kombinace genů u hybridních druhů také naznačují, že oprava DNA[75][110][114] a geny zapojené do mutageneze a cest souvisejících s rakovinou[110] může způsobit nekompatibilitu hybridů. Tvorba genomu u hybridních druhů je formována selekcí proti nekompatibilním kombinacím.[69][99][105]
Změněné vlastnosti genomu
Hybridní původ může ovlivnit strukturu a vlastnosti genomu. Ukázalo se, že se zvyšuje rychlosti mutací,[78][115][116] aktivovat transponovatelné prvky,[117][118][119] a přimět chromozomální přesmyky.[120][121] Zvýšené transposon aktivace, jak je navrženo v McClintockově teorii „genomového šoku“, může vést ke změnám genové exprese. Transponovatelné prvky mohou, kromě změny genových produktů, pokud jsou vloženy do genu, také změnit aktivitu promotoru pro geny, pokud jsou vloženy před kódující oblasti, nebo mohou vyvolat umlčení genu v důsledku narušení genu.[122][123] U alopolyploidních genomů mohou chromozomální přesmyky vyplynout z „genomového šoku“ vyvolaného hybridizací, přičemž vzdáleněji příbuzné druhy jsou náchylnější k reorganizaci genomu, např. v Nicotiana.[124] Chromozomální přesmyky, které jsou výsledkem buď genomového šoku, nebo rekombinačních událostí mezi nehomologními subgenomy, mohou způsobit zvětšení nebo zmenšení velikosti genomu.[125] Nárůsty i poklesy byly zjištěny v EU Nicotiana rodu a nesouvisely s věkem od hybridizace.[126]
Po duplikaci genomu v alopolyploidech genom prochází diploidizace, což je proces, při kterém je genom přeuspořádán tak, aby působil jako meiotický diploid. [127][128] Po takové diploidizaci je velká část genomu ztracena kvůli frakcionaci genomu, ztrátě funkce jednoho nebo druhého z nově duplikovaných genů.[128][129] V metaanalýze Sankoff a spolupracovníci našli důkazy v souladu s páry rezistentními na redukci a koncentrací funkčních genů na jednom chromozomu a naznačují, že proces redukce je částečně omezen.[129]
Souvisejícím fenoménem specifickým pro alopolyploidy je dominance subgenomu. Například v octoploidní jahodě Fragaria je jeden ze čtyř subgenomů dominantní a má výrazně vyšší obsah genů, častěji má své geny exprimované a výměny mezi homologní chromozomy jsou předpojaté ve prospěch tohoto subgenomu ve srovnání s ostatními subgenomy.[130] Tato studie také ukázala, že určité rysy, např. odolnost vůči chorobám, jsou do značné míry kontrolovány dominantním subgenomem.[130] Navrhovaný mechanismus, jak dominance subgenomu vzniká, naznačuje, že relativní dominance souvisí s hustotou transponovatelných prvků v každém subgenomu. Subgenomy s vyšší hustotou transponovatelných prvků mají tendenci se chovat submisivně ve srovnání s ostatními subgenomy, když jsou spojeny v alopolyploidním genomu.[128][131] Interestingly, subgenome dominance can arise immediately in allopolyploids, as shown in synthetic and recently evolved monkeyflowers.[131]
In addition to these changes to genome structure and properties, studies of allopolyploid rice and whitefish suggest that patterns of gene expression may be disrupted in hybrid species.[132][133] Studies of synthetic and natural allopolyploids of Tragopogon miscellus show that gene expression is less strictly regulated directly after hybridization, and that novel patterns of expression emerge and are stabilized during 40 generations.[134] While expression variation in miRNA alters gene expression and affects growth in the natural allopolyploid Arabidopsis suecica and experimental lineages, inheritance of siRNAs is stable and maintains chromatin and genome stability,[135] potentially buffering against a transcriptomic shock.
Factors influencing formation and persistence
Whereas hybridization is required for the generation of persistent hybrid genomes, it is not sufficient. For the persistence of hybrid genomes in hybrid species they need to be sufficiently reproductively isolated from their parent species to avoid species fusion. Selection on introgressed variants allows the persistence of hybrid genomes in introgressed lineages. Frequency of hybridization, viability of hybrids, and the ease at which reproductive isolation against the parent species arises or strength of selection to maintain introgressed regions are hence factors influencing the rate of formation of stable hybrid lineages.
Few general conclusions about the relative prevalence of hybridization can be drawn, as sampling is not evenly distributed, even if there is evidence for hybridization in an increasing number of taxa. One pattern that emerges is that hybridization is more frequent in plants where it occurs in 25% of the species, whereas it only occurs in 10% of animal species.[136] Most plants, as well as many groups of animals, lack heteromorphic sex chromosomes.[137] The absence of heteromorphic sex chromosomes results in slower accumulation of reproductive isolation,[138][139] and may hence enable hybridization between phylogenetically more distant taxa. Haldanovo pravidlo[140] states that ”when F1 offspring of two different animal races one sex is absent, rare, or sterile, that sex is the heterozygous sex”. Empirical evidence supports a role for heteromorphic sex chromosomes in hybrid sterility and inviability. A closely related observation is the large X effect stating that there is a disproportionate contribution of the X/Z-chromosome in fitness reduction of heterogametic hybridy.[22] These patterns likely arise as recessive alleles with deleterious effects in hybrids have a stronger impacts on the heterogametic than the homogametic sex, due to hemizygous výraz.[141] In taxa with well-differentiated sex chromosomes, Haldane’s rule has shown to be close to universal, and heteromorphic sex chromosomes show reduced introgression on the X in XY.[142] In line with a role for heteromorphic sex chromosomes in constraining hybrid genome formation, elevated differentiation on sex chromosomes has been observed in both ZW and XY systems.[143] This pattern may reflect the lower effective population sizes and higher susceptibility to drift on the sex chromosomes,[144] the elevated frequency of loci involved in reproductive isolation[145] and/or the heightened conflict on sex chromosomes.[146] Findings of selection for uniparental inheritance of e.g. mitonuclear loci residing on the Z chromosome in hybrid Italian sparrows[75] is consistent with compatible sex chromosomes being important for the formation of a viable hybrid genomes.
There are also several ecological factors that affect the probability of hybridization. Generally, hybridization is more frequently observed in species with external fertilization including plants but also fishes, than in internally fertilized clades.[4] In plants, high rates of selfing in some species may prevent hybridization, and breeding system may also affect the frequency of heterospecific pollen transfer.[147][148] In fungi, hybrids can be generated by ameiotic fusion of cells or hyphae[149] in addition to mechanisms available to plants and animals. Such fusion of vegetative cells and subsequent parasexual mating with mitotic crossover may generate recombined hybrid cells.[149]
For hybrid species to evolve, reproductive isolation against the parent species is required. The ease by which such reproductive isolation arises is thus also important for the rate at which stable hybrid species arise. Polyploidisation and asexuality are both mechanisms that result in instantaneous isolation and may increase the rate of hybrid lineage formation. The ability to self-pollinate may also act in favour of stabilising allopolyploid taxa by providing a compatible mate (itself) in the early stages of allopolyploid speciation when rare cytotypes are at a reproductive disadvantage due to inter-cytotype mating.[150] Selfing is also expected to increase the likelihood of establishment for homoploid hybrids according to a modelling study,[151] and the higher probability of selfing may contribute to the higher frequency of hybrid species in plants. Fungal hybridization may result in asexual hybrid species, as Epichloe fungi where hybrids species are asexual while nonhybrids include both asexual and sexual species.[152] Hybridization between strongly divergent animal taxa may also generate asexual hybrid species, as shown e.g. in the European spined loaches, Cobitis,[153] and most if not all asexual vertebrate species are of hybrid origin.[154] Interestingly, Arctic floras harbour an unusually high proportion of allopolyploid plants,[155] suggesting that these hybrid taxa could have an advantage in extreme environments, potentially through reducing the negative effects of inbreeding. Hence both genomic architecture and ecological properties may affect the probability of hybrid species formation.
For introgressed taxa, the strength of selection on introgressed variants decides whether introgressed sections will spread in the population and stable introgressed genomes will be formed. Strong selection for insecticide resistance has been shown to increase introgression of an Anopheles gambiae resistance allele into A. coluzzi malaria mosquitoes.[156] v Heliconius butterflies, strong selection on having the locally abundant wing colour patterns repeatedly led to fixation of alleles that introgressed from locally adapted butterflies into newly colonizing species or subspecies.[34] Chances of fixation of beneficial introgressed variants depend on the type and strength of selection on the introgressed variant and linkage with other introgressed variants that are selected against.
Factors influencing affected genes and genomic regions
Genetic exchange can occur between populations or incipient species diverging in geographical proximity or between divergent taxa that come into secondary contact. Hybridization between more diverged lineages is expected to have a greater potential to contribute beneficial alleles or generate novelty than hybridization between less diverged populations because more divergent alleles are combined, and are thus more likely to have a large fitness effect, to generate transgressive phenotypes.[157] Hybridization between more diverged lineages is also more likely to generate incompatible allele combinations, reducing initial hybrid fitness[158] but potentially also contributing to hybrid speciation if they are sorted reciprocally as described above.[157] An intermediate genetic distance may thus be most conducive to hybrid speciation.[157] Experimental lab crosses support this hypothesis.[91]
The proportion of the genome that is inherited from the recipient of introgressed material varies strongly among and within species. After the initial hybridization event the representation is 50% in many polyploid taxa, although parental gene copies are successively lost and might bias the contribution to one majority parent genome.[159] Relatively equal parental contributions are also found in some homoploid hybrid species[74] but in other cases they are highly unequal such as in some Heliconius druh.[160] The majority ancestry may even be that from the donor of introgressed material, as was shown for Anopheles gambiae komáři.[161] Interestingly there may also be variation in parental contribution within a hybrid species. In both swordtail fish and Italian sparrows there are populations which differ strongly in what proportions of the parent genomes they have inherited.[69][70]
Patterns of introgression can vary strongly across the genome, even over short chromosomal distances. Examples of adaptive introgression of well defined regions, include an inversed region containing genes involved in insecticide resistance[21] and introgression of a divergent, inverted chromosomal segment has resulted in a ”super gene ” that encodes mimicry polymorphism in the butterfly Heliconius numata.[162] These findings are consistent with models suggesting that genomic rearrangements are important for the coupling of locally adaptive loci.[163] Genes and genomic regions that are adaptive may be readily introgressed between species e.g. in hybrid zones if they are not linked to incompatibility loci. This often referred to semi-permeable species boundaries,[19][164][165] and examples include e.g. genes involved in olfaction that are introgressed across a Mus musculus a M. domesticus hybrid zone.[166] In hybrid zones with mainly permeable species boundaries, patterns of introgressed regions enable deducing what genomic regions involved in incompatibilities and reproductive isolation.[167]
Reference
![]() This article was adapted from the following source under a CC BY 4.0 license (2019 ) (reviewer reports ): "Eukaryote hybrid genomes", Genetika PLOS, 15 (11): e1008404, 27 November 2019, doi:10.1371/JOURNAL.PGEN.1008404, ISSN 1553-7390, PMC 6880984, PMID 31774811, Wikidata Q86320147
This article was adapted from the following source under a CC BY 4.0 license (2019 ) (reviewer reports ): "Eukaryote hybrid genomes", Genetika PLOS, 15 (11): e1008404, 27 November 2019, doi:10.1371/JOURNAL.PGEN.1008404, ISSN 1553-7390, PMC 6880984, PMID 31774811, Wikidata Q86320147
- ^ A b C d E F G Abbott R, Albach D, Ansell S, Arntzen JW, Baird SJ, Bierne N, et al. (Únor 2013). "Hybridization and speciation". Journal of Evolutionary Biology. 26 (2): 229–46. doi:10.1111/j.1420-9101.2012.02599.x. PMID 23323997. S2CID 830823.
- ^ Fisher RA (1930). The genetical theory of natural selection. Oxford: Clarendon Press. doi:10.5962/bhl.title.27468.
- ^ Mayr E (1963). Animal Species and Evolution. Cambridge, MA and London, England: Harvard University Press. doi:10.4159/harvard.9780674865327. ISBN 9780674865327.
- ^ A b Stebbins GL (1959). "The Role of Hybridization in Evolution". Sborník americké filozofické společnosti. 103 (2): 231–251. ISSN 0003-049X. JSTOR 985151.
- ^ Anderson E, Stebbins GL (1954). "Hybridization as an evolutionary stimulus". Vývoj. 8 (4): 378–388. doi:10.1111/j.1558-5646.1954.tb01504.x.
- ^ Arnold ML (1997). Přirozená hybridizace a evoluce. Cary: Oxford University Press. ISBN 9780195356687. OCLC 960164734.
- ^ Mallet J, Besansky N, Hahn MW (February 2016). "How reticulated are species?". BioEssays. 38 (2): 140–9. doi:10.1002/bies.201500149. PMC 4813508. PMID 26709836.
- ^ A b Lamichhaney S, Han F, Webster MT, Andersson L, Grant BR, Grant PR (January 2018). „Rychlá hybridní speciace v Darwinových finkách“. Věda. 359 (6372): 224–228. Bibcode:2018Sci...359..224L. doi:10.1126 / science.aao4593. PMID 29170277.
- ^ Meier JI, Marques DA, Mwaiko S, Wagner CE, Excoffier L, Seehausen O (February 2017). "Ancient hybridization fuels rapid cichlid fish adaptive radiations". Příroda komunikace. 8 (1): 14363. Bibcode:2017NatCo...814363M. doi:10.1038/ncomms14363. PMC 5309898. PMID 28186104.
- ^ A b Mavárez J, Salazar CA, Bermingham E, Salcedo C, Jiggins CD, Linares M (June 2006). "Speciace hybridizací u motýlů Heliconius". Příroda. 441 (7095): 868–71. Bibcode:2006Natur.441..868M. doi:10.1038 / nature04738. PMID 16778888.
- ^ A b Salazar C, Baxter SW, Pardo-Diaz C, Wu G, Surridge A, Linares M, et al. (Duben 2010). Walsh B (ed.). "Genetic evidence for hybrid trait speciation in heliconius butterflies". Genetika PLOS. 6 (4): e1000930. doi:10.1371/journal.pgen.1000930. PMC 2861694. PMID 20442862.
- ^ A b C Melo MC, Salazar C, Jiggins CD, Linares M (June 2009). "Assortative mating preferences among hybrids offers a route to hybrid speciation". Vývoj; International Journal of Organic Evolution. 63 (6): 1660–5. doi:10.1111/j.1558-5646.2009.00633.x. PMID 19492995.
- ^ Carlquist SJ, Baldwin BG, Carr GD (2003). Tarweeds & silverswords : evolution of the Madiinae (Asteraceae). St. Louis: Missouri Botanical Garden Press. ISBN 1930723202. OCLC 52892451.
- ^ A b Wolf DE, Takebayashi N, Rieseberg LH (2001). "Predicting the Risk of Extinction through Hybridization". Biologie ochrany. 15 (4): 1039–1053. doi:10.1046/j.1523-1739.2001.0150041039.x. ISSN 0888-8892.
- ^ Prentis PJ, White EM, Radford IJ, Lowe AJ, Clarke AR (2007). "Can hybridization cause local extinction: a case for demographic swamping of the Australian native Senecio pinnatifolius by the invasive Senecio madagascariensis?" (PDF). Nový fytolog. 176 (4): 902–12. doi:10.1111/j.1469-8137.2007.02217.x. PMID 17850249.
- ^ Servedio MR, Noor MA (2003). "The Role of Reinforcement in Speciation: Theory and Data". Výroční přehled ekologie, evoluce a systematiky. 34 (1): 339–364. doi:10.1146/annurev.ecolsys.34.011802.132412. ISSN 1543-592X.
- ^ Rhymer JM, Simberloff D (1996). "Vyhynutí hybridizací a introgrese". Výroční přehled ekologie a systematiky. 27 (1): 83–109. doi:10.1146 / annurev.ecolsys.27.1.83. ISSN 0066-4162.
- ^ Seehausen O (May 2006). "Conservation: losing biodiversity by reverse speciation". Aktuální biologie. 16 (9): R334-7. doi:10.1016/j.cub.2006.03.080. PMID 16682344.
- ^ A b Thompson JD (1994). "Harrison, R. G. (ed.). Hybrid Zones and the Evolutionary Process. Oxford University Press, Oxford. 364 pp. Price f45.00". Journal of Evolutionary Biology. 7 (5): 631–634. doi:10.1046/j.1420-9101.1994.7050631.x. ISBN 0-19-506917-X. ISSN 1010-061X.
- ^ A b Dasmahapatra KK, Walters JR, Briscoe AD, Davey JW, Whibley A, Nadeau NJ, et al. (Heliconius Genome Consortium) (July 2012). "Butterfly genome reveals promiscuous exchange of mimicry adaptations among species". Příroda. 487 (7405): 94–8. Bibcode:2012Natur.487...94T. doi:10.1038/nature11041. PMC 3398145. PMID 22722851.
- ^ A b C d E Hanemaaijer MJ, Collier TC, Chang A, Shott CC, Houston PD, Schmidt H, et al. (Prosinec 2018). "The fate of genes that cross species boundaries after a major hybridization event in a natural mosquito population". Molekulární ekologie. 27 (24): 4978–4990. doi:10.1111/mec.14947. PMID 30447117.
- ^ A b Coyne JA, Orr HA (2004). Speciace. Sunderland: Sinauer Associates. ISBN 0878930914. OCLC 55078441.
- ^ Price TD, Bouvier MM (2002). "The evolution of F1 postzygotic incompatibilities in birds". Vývoj. 56 (10): 2083–9. doi:10.1554/0014-3820(2002)056[2083:teofpi]2.0.co;2. ISSN 0014-3820. PMID 12449494.
- ^ Stelkens RB, Young KA, Seehausen O (March 2010). "The accumulation of reproductive incompatibilities in African cichlid fish". Vývoj; International Journal of Organic Evolution. 64 (3): 617–33. doi:10.1111/j.1558-5646.2009.00849.x. PMID 19796149. S2CID 10319450.
- ^ Rebernig CA, Lafon-Placette C, Hatorangan MR, Slotte T, Köhler C (June 2015). Bomblies K (ed.). "Non-reciprocal Interspecies Hybridization Barriers in the Capsella Genus Are Established in the Endosperm". Genetika PLOS. 11 (6): e1005295. doi:10.1371/journal.pgen.1005295. PMC 4472357. PMID 26086217.
- ^ Pritchard VL, Knutson VL, Lee M, Zieba J, Edmands S (February 2013). "Fitness and morphological outcomes of many generations of hybridization in the copepod Tigriopus californicus". Journal of Evolutionary Biology. 26 (2): 416–33. doi:10.1111/jeb.12060. PMID 23278939. S2CID 10092426.
- ^ Rieseberg LH, Archer MA, Wayne RK (October 1999). "Transgressive segregation, adaptation and speciation". Dědičnost. 83 ( Pt 4) (4): 363–72. doi:10.1038/sj.hdy.6886170. PMID 10583537.
- ^ A b C Burke JM, Arnold ML (2001). "Genetics and the fitness of hybrids". Výroční přehled genetiky. 35 (1): 31–52. doi:10.1146/annurev.genet.35.102401.085719. PMID 11700276. S2CID 26683922.
- ^ A b C d Mallet J (March 2007). "Hybrid speciation". Příroda. 446 (7133): 279–83. Bibcode:2007Natur.446..279M. doi:10.1038/nature05706. PMID 17361174.
- ^ A b Vallejo-Marín M, Hiscock SJ (September 2016). "Hybridization and hybrid speciation under global change". Nový fytolog. 211 (4): 1170–87. doi:10.1111/nph.14004. hdl:1893/23581. PMID 27214560.
- ^ Barton N, Bengtsson BO (December 1986). "The barrier to genetic exchange between hybridising populations". Dědičnost. 57 ( Pt 3) (3): 357–76. doi:10.1038/hdy.1986.135. PMID 3804765.
- ^ Demon I, Haccou P, van den Bosch F (September 2007). "Introgression of resistance genes between populations: a model study of insecticide resistance in Bemisia tabaci". Teoretická populační biologie. 72 (2): 292–304. doi:10.1016/j.tpb.2007.06.005. PMID 17658572.
- ^ Uecker H, Setter D, Hermisson J (June 2015). "Adaptive gene introgression after secondary contact". Journal of Mathematical Biology. 70 (7): 1523–80. doi:10.1007/s00285-014-0802-y. PMC 4426140. PMID 24992884.
- ^ A b Pardo-Diaz C, Salazar C, Baxter SW, Merot C, Figueiredo-Ready W, Joron M, et al. (2012). R Kronforst M (ed.). "Adaptive introgression across species boundaries in Heliconius butterflies". Genetika PLOS. 8 (6): e1002752. doi:10.1371/journal.pgen.1002752. PMC 3380824. PMID 22737081.
- ^ Arnold BJ, Lahner B, DaCosta JM, Weisman CM, Hollister JD, Salt DE, et al. (Červenec 2016). "Borrowed alleles and convergence in serpentine adaptation". Sborník Národní akademie věd Spojených států amerických. 113 (29): 8320–5. doi:10.1073/pnas.1600405113. PMC 4961121. PMID 27357660.
- ^ Racimo F, Sankararaman S, Nielsen R, Huerta-Sánchez E (June 2015). "Evidence for archaic adaptive introgression in humans". Recenze přírody. Genetika. 16 (6): 359–71. doi:10.1038/nrg3936. PMC 4478293. PMID 25963373.
- ^ Kronforst MR, Papa R (May 2015). "The functional basis of wing patterning in Heliconius butterflies: the molecules behind mimicry". Genetika. 200 (1): 1–19. doi:10.1534/genetics.114.172387. PMC 4423356. PMID 25953905.
- ^ Mérot C, Salazar C, Merrill RM, Jiggins CD, Joron M (June 2017). "Heliconius butterflies". Řízení. Biologické vědy. 284 (1856): 20170335. doi:10.1098/rspb.2017.0335. PMC 5474069. PMID 28592669.
- ^ Pritchard JK, Stephens M, Donnelly P (June 2000). "Inference of population structure using multilocus genotype data". Genetika. 155 (2): 945–59. PMC 1461096. PMID 10835412.
- ^ Alexander DH, Novembre J, Lange K (September 2009). "Fast model-based estimation of ancestry in unrelated individuals". Výzkum genomu. 19 (9): 1655–64. doi:10.1101/gr.094052.109. PMC 2752134. PMID 19648217.
- ^ Lawson DJ, Hellenthal G, Myers S, Falush D (January 2012). Copenhaver GP (ed.). "Inference of population structure using dense haplotype data". Genetika PLOS. 8 (1): e1002453. doi:10.1371/journal.pgen.1002453. PMC 3266881. PMID 22291602.
- ^ Lawson DJ, van Dorp L, Falush D (August 2018). "A tutorial on how not to over-interpret STRUCTURE and ADMIXTURE bar plots". Příroda komunikace. 9 (1): 3258. Bibcode:2018NatCo...9.3258L. doi:10.1038/s41467-018-05257-7. PMC 6092366. PMID 30108219.
- ^ Kulathinal RJ, Stevison LS, Noor MA (July 2009). Nachman MW (ed.). "The genomics of speciation in Drosophila: diversity, divergence, and introgression estimated using low-coverage genome sequencing". Genetika PLOS. 5 (7): e1000550. doi:10.1371/journal.pgen.1000550. PMC 2696600. PMID 19578407.
- ^ Green RE, Krause J, Briggs AW, Maricic T, Stenzel U, Kircher M, et al. (Květen 2010). "A draft sequence of the Neandertal genome". Věda. 328 (5979): 710–722. Bibcode:2010Sci...328..710G. doi:10.1126/science.1188021. PMC 5100745. PMID 20448178.
- ^ Durand EY, Patterson N, Reich D, Slatkin M (August 2011). "Testing for ancient admixture between closely related populations". Molekulární biologie a evoluce. 28 (8): 2239–52. doi:10.1093/molbev/msr048. PMC 3144383. PMID 21325092.
- ^ Peter BM (April 2016). "Admixture, Population Structure, and F-Statistics". Genetika. 202 (4): 1485–501. doi:10.1534/genetics.115.183913. PMC 4905545. PMID 26857625.
- ^ A b Reich D, Thangaraj K, Patterson N, Price AL, Singh L (September 2009). "Reconstructing Indian population history". Příroda. 461 (7263): 489–94. Bibcode:2009Natur.461..489R. doi:10.1038/nature08365. PMC 2842210. PMID 19779445.
- ^ Martin SH, Davey JW, Jiggins CD (January 2015). "Evaluating the use of ABBA-BABA statistics to locate introgressed loci". Molekulární biologie a evoluce. 32 (1): 244–57. doi:10.1093/molbev/msu269. PMC 4271521. PMID 25246699.
- ^ Pease JB, Hahn MW (July 2015). "Detection and Polarization of Introgression in a Five-Taxon Phylogeny". Systematická biologie. 64 (4): 651–62. doi:10.1093/sysbio/syv023. PMID 25888025.
- ^ Eaton DA, Ree RH (September 2013). "Inferring phylogeny and introgression using RADseq data: an example from flowering plants (Pedicularis: Orobanchaceae)". Systematická biologie. 62 (5): 689–706. doi:10.1093/sysbio/syt032. PMC 3739883. PMID 23652346.
- ^ Pickrell JK, Pritchard JK (2012). Tang H (ed.). "Inference of population splits and mixtures from genome-wide allele frequency data". Genetika PLOS. 8 (11): e1002967. arXiv:1206.2332. Bibcode:2012arXiv1206.2332P. doi:10.1371/journal.pgen.1002967. PMC 3499260. PMID 23166502.
- ^ A b Patterson N, Moorjani P, Luo Y, Mallick S, Rohland N, Zhan Y, et al. (Listopad 2012). "Ancient admixture in human history". Genetika. 192 (3): 1065–93. doi:10.1534/genetics.112.145037. PMC 3522152. PMID 22960212.
- ^ Lipson M, Loh PR, Levin A, Reich D, Patterson N, Berger B (August 2013). "Efficient moment-based inference of admixture parameters and sources of gene flow". Molekulární biologie a evoluce. 30 (8): 1788–802. doi:10.1093/molbev/mst099. PMC 3708505. PMID 23709261.
- ^ Yu Y, Barnett RM, Nakhleh L (September 2013). "Parsimonious inference of hybridization in the presence of incomplete lineage sorting". Systematická biologie. 62 (5): 738–51. doi:10.1093/sysbio/syt037. PMC 3739885. PMID 23736104.
- ^ Wen D, Yu Y, Nakhleh L (May 2016). Edwards S (ed.). "Bayesian Inference of Reticulate Phylogenies under the Multispecies Network Coalescent". Genetika PLOS. 12 (5): e1006006. doi:10.1371/journal.pgen.1006006. PMC 4856265. PMID 27144273.
- ^ Moorjani P, Patterson N, Hirschhorn JN, Keinan A, Hao L, Atzmon G, et al. (Duben 2011). McVean G (ed.). "The history of African gene flow into Southern Europeans, Levantines, and Jews". Genetika PLOS. 7 (4): e1001373. doi:10.1371/journal.pgen.1001373. PMC 3080861. PMID 21533020.
- ^ Moorjani P, Sankararaman S, Fu Q, Przeworski M, Patterson N, Reich D (May 2016). "A genetic method for dating ancient genomes provides a direct estimate of human generation interval in the last 45,000 years". Sborník Národní akademie věd Spojených států amerických. 113 (20): 5652–7. Bibcode:2016PNAS..113.5652M. doi:10.1073/pnas.1514696113. PMC 4878468. PMID 27140627.
- ^ Loh PR, Lipson M, Patterson N, Moorjani P, Pickrell JK, Reich D, Berger B (April 2013). "Inferring admixture histories of human populations using linkage disequilibrium". Genetika. 193 (4): 1233–54. doi:10.1534/genetics.112.147330. PMC 3606100. PMID 23410830.
- ^ Sankararaman S, Patterson N, Li H, Pääbo S, Reich D (2012). Akey JM (ed.). "The date of interbreeding between Neandertals and modern humans". Genetika PLOS. 8 (10): e1002947. arXiv:1208.2238. Bibcode:2012arXiv1208.2238S. doi:10.1371/journal.pgen.1002947. PMC 3464203. PMID 23055938.
- ^ Pinho C, Hey J (2010). "Divergence with Gene Flow: Models and Data". Výroční přehled ekologie, evoluce a systematiky. 41 (1): 215–230. doi:10.1146/annurev-ecolsys-102209-144644. ISSN 1543-592X. S2CID 45813707.
- ^ Excoffier L, Dupanloup I, Huerta-Sánchez E, Sousa VC, Foll M (October 2013). Akey JM (ed.). "Robust demographic inference from genomic and SNP data". Genetika PLOS. 9 (10): e1003905. doi:10.1371/journal.pgen.1003905. PMC 3812088. PMID 24204310.
- ^ Gutenkunst RN, Hernandez RD, Williamson SH, Bustamante CD (October 2009). McVean G (ed.). "Inferring the joint demographic history of multiple populations from multidimensional SNP frequency data". Genetika PLOS. 5 (10): e1000695. arXiv:0909.0925. Bibcode:2009arXiv0909.0925G. doi:10.1371/journal.pgen.1000695. PMC 2760211. PMID 19851460.
- ^ Beaumont MA (2010). "Approximate Bayesian Computation in Evolution and Ecology". Výroční přehled ekologie, evoluce a systematiky. 41 (1): 379–406. doi:10.1146/annurev-ecolsys-102209-144621.
- ^ Theunert C, Slatkin M (February 2017). "Distinguishing recent admixture from ancestral population structure". Biologie genomu a evoluce. 9 (3): 427–437. doi:10.1093/gbe/evx018. PMC 5381645. PMID 28186554.
- ^ A b C Schumer M, Rosenthal GG, Andolfatto P (June 2014). "How common is homoploid hybrid speciation?". Vývoj; International Journal of Organic Evolution. 68 (6): 1553–60. doi:10.1111/evo.12399. PMID 24620775. S2CID 22702297.
- ^ A b Nieto Feliner G, Álvarez I, Fuertes-Aguilar J, Heuertz M, Marques I, Moharrek F, et al. (Červen 2017). "Is homoploid hybrid speciation that rare? An empiricist's view". Dědičnost. 118 (6): 513–516. doi:10.1038/hdy.2017.7. PMC 5436029. PMID 28295029.
- ^ A b Rieseberg LH, Raymond O, Rosenthal DM, Lai Z, Livingstone K, Nakazato T, et al. (Srpen 2003). "Major ecological transitions in wild sunflowers facilitated by hybridization". Věda. 301 (5637): 1211–6. Bibcode:2003Sci...301.1211R. doi:10.1126/science.1086949. PMID 12907807. S2CID 9232157.
- ^ Grant V (1981). Plant speciation (2. vyd.). New York: Columbia University Press. ISBN 0231051123. OCLC 7552165.
- ^ A b C d Schumer M, Xu C, Powell DL, Durvasula A, Skov L, Holland C, et al. (Květen 2018). "Natural selection interacts with recombination to shape the evolution of hybrid genomes". Věda. 360 (6389): 656–660. Bibcode:2018Sci...360..656S. doi:10.1126/science.aar3684. PMC 6069607. PMID 29674434.
- ^ A b Runemark A, Trier CN, Eroukhmanoff F, Hermansen JS, Matschiner M, Ravinet M, et al. (Březen 2018). "Variation and constraints in hybrid genome formation". Ekologie a evoluce přírody. 2 (3): 549–556. doi:10.1038/s41559-017-0437-7. PMID 29335572.
- ^ A b C d Buerkle CA, Rieseberg LH (February 2008). "The rate of genome stabilization in homoploid hybrid species". Vývoj; International Journal of Organic Evolution. 62 (2): 266–75. doi:10.1111/j.1558-5646.2007.00267.x. PMC 2442919. PMID 18039323.
- ^ Ungerer MC, Baird SJ, Pan J, Rieseberg LH (September 1998). "Rapid hybrid speciation in wild sunflowers". Sborník Národní akademie věd Spojených států amerických. 95 (20): 11757–62. Bibcode:1998PNAS...9511757U. doi:10.1073/pnas.95.20.11757. PMC 21713. PMID 9751738.
- ^ A b C Lai Z, Nakazato T, Salmaso M, Burke JM, Tang S, Knapp SJ, Rieseberg LH (September 2005). "Extensive chromosomal repatterning and the evolution of sterility barriers in hybrid sunflower species". Genetika. 171 (1): 291–303. doi:10.1534/genetics.105.042242. PMC 1456521. PMID 16183908.
- ^ A b Elgvin TO, Trier CN, Tørresen OK, Hagen IJ, Lien S, Nederbragt AJ, et al. (Červen 2017). "The genomic mosaicism of hybrid speciation". Vědecké zálohy. 3 (6): e1602996. Bibcode:2017SciA....3E2996E. doi:10.1126/sciadv.1602996. PMC 5470830. PMID 28630911.
- ^ A b C d Runemark A, Trier CN, Eroukhmanoff F, Hermansen JS, Matschiner M, Ravinet M, et al. (Březen 2018). "Variation and constraints in hybrid genome formation". Ekologie a evoluce přírody. 2 (3): 549–556. doi:10.1038/s41559-017-0437-7. PMID 29335572.
- ^ A b Otto SP, Whitton J (2000). "Polyploid incidence and evolution". Výroční přehled genetiky. 34 (1): 401–437. doi:10.1146/annurev.genet.34.1.401. PMID 11092833.
- ^ Abbott RJ, Rieseberg LH (2012). Hybrid Speciation. eLS. John Wiley & Sons, Ltd. doi:10.1002/9780470015902.a0001753.pub2. ISBN 9780470016176.
- ^ A b Coyne JA (October 1989). "Mutation rates in hybrids between sibling species of Drosophila". Dědičnost. 63 ( Pt 2) (2): 155–62. doi:10.1038/hdy.1989.87. PMID 2553645.
- ^ Chase MW, Paun O, Fay MF (2010). "Hybridization and speciation in angiosperms: arole for pollinator shifts?". Journal of Biology. 9 (3): 21. doi:10.1186/jbiol231. ISSN 1475-4924.
- ^ Grant V (March 1949). "Pollination systems as isolating mechanisms in angiosperms". Vývoj; International Journal of Organic Evolution. 3 (1): 82–97. doi:10.1111/j.1558-5646.1949.tb00007.x. PMID 18115119.
- ^ Segraves KA, Thompson JN (August 1999). "Plant Polyploidy and Pollination: Floral Traits and Insect Visits to Diploid and Tetraploidheuchera Grossulariifolia". Vývoj; International Journal of Organic Evolution. 53 (4): 1114–1127. doi:10.1111/j.1558-5646.1999.tb04526.x. PMID 28565509.
- ^ Moe AM, Weiblen GD (December 2012). "Pollinator-mediated reproductive isolation among dioecious fig species (Ficus, Moraceae)". Vývoj; International Journal of Organic Evolution. 66 (12): 3710–21. doi:10.1111/j.1558-5646.2012.01727.x. PMID 23206130.
- ^ A b Lowe AJ, Abbott RJ (May 2004). "Reproductive isolation of a new hybrid species, Senecio eboracensis Abbott & Lowe (Asteraceae)". Dědičnost. 92 (5): 386–95. doi:10.1038/sj.hdy.6800432. PMID 15014422.
- ^ Selz OM, Thommen R, Maan ME, Seehausen O (February 2014). "Behavioural isolation may facilitate homoploid hybrid speciation in cichlid fish". Journal of Evolutionary Biology. 27 (2): 275–89. doi:10.1111/jeb.12287. PMID 24372872.
- ^ A b Schwarzbach AE, Donovan LA, Rieseberg LH (2001). "Transgressive character expression in a hybrid sunflower species". American Journal of Botany. 88 (2): 270–277. doi:10.2307/2657018. ISSN 0002-9122. JSTOR 2657018.
- ^ Mameli G, López-Alvarado J, Farris E, Susanna A, Filigheddu R, Garcia-Jacas N (2014). "The role of parental and hybrid species in multiple introgression events: evidence of homoploid hybrid speciation in Centaurea (Cardueae, Asteraceae): Introgression in Centaurea". Botanical Journal of the Linnean Society. 175 (3): 453–467. doi:10.1111/boj.12177.
- ^ Xie X, Michel AP, Schwarz D, Rull J, Velez S, Forbes AA, et al. (Květen 2008). "Radiation and divergence in the Rhagoletis pomonella species complex: inferences from DNA sequence data". Journal of Evolutionary Biology. 21 (3): 900–13. doi:10.1111/j.1420-9101.2008.01507.x. PMID 18312319.
- ^ Feder JL, Xie X, Rull J, Velez S, Forbes A, Leung B, et al. (Květen 2005). "Mayr, Dobzhansky, and Bush and the complexities of sympatric speciation in Rhagoletis". Sborník Národní akademie věd Spojených států amerických. 102 Suppl 1 (Supplement 1): 6573–80. Bibcode:2005PNAS..102.6573F. doi:10.1073/pnas.0502099102. PMC 1131876. PMID 15851672.
- ^ Schumer M, Powell DL, Delclós PJ, Squire M, Cui R, Andolfatto P, Rosenthal GG (October 2017). "Assortative mating and persistent reproductive isolation in hybrids". Sborník Národní akademie věd Spojených států amerických. 114 (41): 10936–10941. doi:10.1073/pnas.1711238114. PMC 5642718. PMID 28973863.
- ^ Rieseberg LH, Linder CR, Seiler GJ (November 1995). "Chromosomal and genic barriers to introgression in Helianthus". Genetika. 141 (3): 1163–71. PMC 1206838. PMID 8582621.
- ^ A b Comeault AA, Matute DR (September 2018). "Genetic divergence and the number of hybridizing species affect the path to homoploid hybrid speciation". Sborník Národní akademie věd Spojených států amerických. 115 (39): 9761–9766. doi:10.1073/pnas.1809685115. PMC 6166845. PMID 30209213.
- ^ Blanckaert A, Bank C (September 2018). Zhang J (ed.). "In search of the Goldilocks zone for hybrid speciation". Genetika PLOS. 14 (9): e1007613. doi:10.1371/journal.pgen.1007613. PMC 6145587. PMID 30192761.
- ^ Schumer M, Cui R, Rosenthal GG, Andolfatto P (March 2015). Payseur BA (ed.). "Reproductive isolation of hybrid populations driven by genetic incompatibilities". Genetika PLOS. 11 (3): e1005041. doi:10.1371/journal.pgen.1005041. PMC 4359097. PMID 25768654.
- ^ Vereecken NJ, Cozzolino S, Schiestl FP (April 2010). "Hybrid floral scent novelty drives pollinator shift in sexually deceptive orchids". BMC Evoluční biologie. 10 (1): 103. doi:10.1186/1471-2148-10-103. PMC 2875231. PMID 20409296.
- ^ Gaeta RT, Chris Pires J (April 2010). "Homoeologous recombination in allopolyploids: the polyploid ratchet". Nový fytolog. 186 (1): 18–28. doi:10.1111/j.1469-8137.2009.03089.x. PMID 20002315.
- ^ A b Hvala JA, Frayer ME, Payseur BA (May 2018). "Signatures of hybridization and speciation in genomic patterns of ancestry". Vývoj; International Journal of Organic Evolution. 72 (8): 1540–1552. doi:10.1111/evo.13509. PMC 6261709. PMID 29806154.
- ^ Rieseberg LH, Sinervo B, Linder CR, Ungerer MC, Arias DM (May 1996). "Role of Gene Interactions in Hybrid Speciation: Evidence from Ancient and Experimental Hybrids". Věda. 272 (5262): 741–5. Bibcode:1996Sci...272..741R. doi:10.1126/science.272.5262.741. PMID 8662570. S2CID 39005242.
- ^ Stukenbrock EH, Christiansen FB, Hansen TT, Dutheil JY, Schierup MH (July 2012). "Fusion of two divergent fungal individuals led to the recent emergence of a unique widespread pathogen species". Sborník Národní akademie věd Spojených států amerických. 109 (27): 10954–9. Bibcode:2012PNAS..10910954S. doi:10.1073/pnas.1201403109. PMC 3390827. PMID 22711811.
- ^ A b Schumer M, Brandvain Y (June 2016). "Determining epistatic selection in admixed populations". Molekulární ekologie. 25 (11): 2577–91. doi:10.1111/mec.13641. PMID 27061282.
- ^ Sankararaman S, Mallick S, Dannemann M, Prüfer K, Kelso J, Pääbo S, et al. (Březen 2014). "The genomic landscape of Neanderthal ancestry in present-day humans". Příroda. 507 (7492): 354–7. Bibcode:2014Natur.507..354S. doi:10.1038/nature12961. PMC 4072735. PMID 24476815.
- ^ Eroukhmanoff F, Bailey RI, Elgvin TO, Hermansen JS, Runemark AR, Trier CN, Sætre G (2017). "Resolution of conflict between parental genomes in a hybrid species". bioRxiv. doi:10.1101/102970.
- ^ Ohta T (1971). "Associative overdominance caused by linked detrimental mutations". Genetical Research. 18 (3): 277–286. doi:10.1017/s0016672300012684. ISSN 0016-6723.
- ^ Zhao L, Charlesworth B (July 2016). "Resolving the Conflict Between Associative Overdominance and Background Selection". Genetika. 203 (3): 1315–34. doi:10.1534/genetics.116.188912. PMC 4937488. PMID 27182952.
- ^ Faria R, Johannesson K, Butlin RK, Westram AM (March 2019). "Evolving Inversions". Trendy v ekologii a evoluci. 34 (3): 239–248. doi:10.1016/j.tree.2018.12.005. PMID 30691998.
- ^ A b Barton NH (December 2018). "The consequences of an introgression event". Molekulární ekologie. 27 (24): 4973–4975. doi:10.1111/mec.14950. PMID 30599087.
- ^ A b Martin SH, Davey JW, Salazar C, Jiggins CD (February 2019). Moyle L (ed.). "Recombination rate variation shapes barriers to introgression across butterfly genomes". PLOS Biology. 17 (2): e2006288. doi:10.1371/journal.pbio.2006288. PMC 6366726. PMID 30730876.
- ^ Brandvain Y, Kenney AM, Flagel L, Coop G, Sweigart AL (June 2014). Jiggins CD (ed.). "Speciation and introgression between Mimulus nasutus and Mimulus guttatus". Genetika PLOS. 10 (6): e1004410. doi:10.1371/journal.pgen.1004410. PMC 4072524. PMID 24967630.
- ^ Janoušek V, Munclinger P, Wang L, Teeter KC, Tucker PK (May 2015). "Functional organization of the genome may shape the species boundary in the house mouse". Molekulární biologie a evoluce. 32 (5): 1208–20. doi:10.1093/molbev/msv011. PMC 4408407. PMID 25631927.
- ^ Schumer M, Cui R, Powell DL, Dresner R, Rosenthal GG, Andolfatto P (June 2014). "High-resolution mapping reveals hundreds of genetic incompatibilities in hybridizing fish species". eLife. 3. doi:10.7554/eLife.02535. PMC 4080447. PMID 24898754.
- ^ A b C Liu S, Luo J, Chai J, Ren L, Zhou Y, Huang F, et al. (Únor 2016). "Genomic incompatibilities in the diploid and tetraploid offspring of the goldfish × common carp cross". Sborník Národní akademie věd Spojených států amerických. 113 (5): 1327–32. Bibcode:2016PNAS..113.1327L. doi:10.1073/pnas.1512955113. PMC 4747765. PMID 26768847.
- ^ Trier CN, Hermansen JS, Sætre GP, Bailey RI (January 2014). Jiggins CD (ed.). "Evidence for mito-nuclear and sex-linked reproductive barriers between the hybrid Italian sparrow and its parent species". Genetika PLOS. 10 (1): e1004075. doi:10.1371/journal.pgen.1004075. PMC 3886922. PMID 24415954.
- ^ Giordano L, Sillo F, Garbelotto M, Gonthier P (January 2018). "Mitonuclear interactions may contribute to fitness of fungal hybrids". Vědecké zprávy. 8 (1): 1706. Bibcode:2018NatSR...8.1706G. doi:10.1038/s41598-018-19922-w. PMC 5786003. PMID 29374209.
- ^ Case AL, Finseth FR, Barr CM, Fishman L (September 2016). "Selfish evolution of cytonuclear hybrid incompatibility in Mimulus". Řízení. Biologické vědy. 283 (1838): 20161493. doi:10.1098/rspb.2016.1493. PMC 5031664. PMID 27629037.
- ^ David WM, Mitchell DL, Walter RB (July 2004). "DNA repair in hybrid fish of the genus Xiphophorus". Srovnávací biochemie a fyziologie. Toxikologie a farmakologie. 138 (3): 301–9. doi:10.1016/j.cca.2004.07.006. PMID 15533788.
- ^ Avila V, Chavarrías D, Sánchez E, Manrique A, López-Fanjul C, García-Dorado A (May 2006). "Increase of the spontaneous mutation rate in a long-term experiment with Drosophila melanogaster". Genetika. 173 (1): 267–77. doi:10.1534/genetics.106.056200. PMC 1461422. PMID 16547099.
- ^ Bashir T, Sailer C, Gerber F, Loganathan N, Bhoopalan H, Eichenberger C, et al. (Květen 2014). "Hybridization alters spontaneous mutation rates in a parent-of-origin-dependent fashion in Arabidopsis". Fyziologie rostlin. 165 (1): 424–37. doi:10.1104/pp.114.238451. PMC 4012600. PMID 24664208.
- ^ Dennenmoser S, Sedlazeck FJ, Iwaszkiewicz E, Li XY, Altmüller J, Nolte AW (September 2017). "Copy number increases of transposable elements and protein-coding genes in an invasive fish of hybrid origin". Molekulární ekologie. 26 (18): 4712–4724. doi:10.1111/mec.14134. PMC 5638112. PMID 28390096.
- ^ Dion-Côté AM, Renaut S, Normandeau E, Bernatchez L (May 2014). "RNA-seq reveals transcriptomic shock involving transposable elements reactivation in hybrids of young lake whitefish species". Molekulární biologie a evoluce. 31 (5): 1188–99. doi:10.1093/molbev/msu069. PMID 24505119.
- ^ Senerchia N, Felber F, Parisod C (April 2015). "Genome reorganization in F1 hybrids uncovers the role of retrotransposons in reproductive isolation". Řízení. Biologické vědy. 282 (1804): 20142874. doi:10.1098/rspb.2014.2874. PMC 4375867. PMID 25716787.
- ^ Ostberg CO, Hauser L, Pritchard VL, Garza JC, Naish KA (August 2013). "Chromosome rearrangements, recombination suppression, and limited segregation distortion in hybrids between Yellowstone cutthroat trout (Oncorhynchus clarkii bouvieri) and rainbow trout (O. mykiss)". BMC Genomics. 14 (1): 570. doi:10.1186/1471-2164-14-570. PMC 3765842. PMID 23968234.
- ^ Hirai H, Hirai Y, Morimoto M, Kaneko A, Kamanaka Y, Koga A (April 2017). "Night Monkey Hybrids Exhibit De Novo Genomic and Karyotypic Alterations: The First Such Case in Primates". Biologie genomu a evoluce. 9 (4): 945–955. doi:10.1093/gbe/evx058. PMC 5388293. PMID 28369492.
- ^ Barkan A, Martienssen RA (April 1991). "Inactivation of maize transposon Mu suppresses a mutant phenotype by activating an outward-reading promoter near the end of Mu1". Sborník Národní akademie věd Spojených států amerických. 88 (8): 3502–6. Bibcode:1991PNAS...88.3502B. doi:10.1073/pnas.88.8.3502. PMC 51476. PMID 1849660.
- ^ Raizada MN, Benito M, Walbot V (2008). "The MuDR transposon terminal inverted repeat contains a complex plant promoter directing distinct somatic and germinal programs: Transposon promoter expression pattern". The Plant Journal. 25 (1): 79–91. doi:10.1111/j.1365-313X.2001.00939.x. S2CID 26084899.
- ^ Lim KY, Matyasek R, Kovarik A, Leitch AR (2004). "Genome evolution in allotetraploid Nicotiana". Biologický žurnál společnosti Linnean. 82 (4): 599–606. doi:10.1111/j.1095-8312.2004.00344.x.
- ^ Baack EJ, Whitney KD, Rieseberg LH (August 2005). "Hybridization and genome size evolution: timing and magnitude of nuclear DNA content increases in Helianthus homoploid hybrid species". Nový fytolog. 167 (2): 623–30. doi:10.1111/j.1469-8137.2005.01433.x. PMC 2442926. PMID 15998412.
- ^ Leitch IJ, Hanson L, Lim KY, Kovarik A, Chase MW, Clarkson JJ, Leitch AR (April 2008). "The ups and downs of genome size evolution in polyploid species of Nicotiana (Solanaceae)". Annals of Botany. 101 (6): 805–14. doi:10.1093/aob/mcm326. PMC 2710205. PMID 18222910.
- ^ Wolfe KH (May 2001). "Yesterday's polyploids and the mystery of diploidization". Recenze přírody. Genetika. 2 (5): 333–41. doi:10.1038/35072009. PMID 11331899.
- ^ A b C Freeling M, Scanlon MJ, Fowler JE (December 2015). "Fractionation and subfunctionalization following genome duplications: mechanisms that drive gene content and their consequences". Aktuální názor na genetiku a vývoj. 35: 110–8. doi:10.1016/j.gde.2015.11.002. PMID 26657818.
- ^ A b Sankoff D, Zheng C, Zhu Q (May 2010). "The collapse of gene complement following whole genome duplication". BMC Genomics. 11 (1): 313. doi:10.1186/1471-2164-11-313. PMC 2896955. PMID 20482863.
- ^ A b Edger PP, Poorten TJ, VanBuren R, Hardigan MA, Colle M, McKain MR, et al. (Březen 2019). "Origin and evolution of the octoploid strawberry genome". Genetika přírody. 51 (3): 541–547. doi:10.1038/s41588-019-0356-4. PMC 6882729. PMID 30804557.
- ^ A b Edger PP, Smith R, McKain MR, Cooley AM, Vallejo-Marin M, Yuan Y, et al. (Září 2017). "Subgenome Dominance in an Interspecific Hybrid, Synthetic Allopolyploid, and a 140-Year-Old Naturally Established Neo-Allopolyploid Monkeyflower". Rostlinná buňka. 29 (9): 2150–2167. doi:10.1105/tpc.17.00010. PMC 5635986. PMID 28814644.
- ^ Xu C, Bai Y, Lin X, Zhao N, Hu L, Gong Z, et al. (Květen 2014). "Genome-wide disruption of gene expression in allopolyploids but not hybrids of rice subspecies". Molekulární biologie a evoluce. 31 (5): 1066–76. doi:10.1093/molbev/msu085. PMC 3995341. PMID 24577842.
- ^ Renaut S, Nolte AW, Bernatchez L (April 2009). "Gene expression divergence and hybrid misexpression between lake whitefish species pairs (Coregonus spp. Salmonidae)". Molekulární biologie a evoluce. 26 (4): 925–36. doi:10.1093/molbev/msp017. PMID 19174479.
- ^ Buggs RJ, Zhang L, Miles N, Tate JA, Gao L, Wei W, et al. (Duben 2011). "Transcriptomic shock generates evolutionary novelty in a newly formed, natural allopolyploid plant". Aktuální biologie. 21 (7): 551–6. doi:10.1016/j.cub.2011.02.016. PMID 21419627.
- ^ Ha M, Lu J, Tian L, Ramachandran V, Kasschau KD, Chapman EJ, et al. (Říjen 2009). "Small RNAs serve as a genetic buffer against genomic shock in Arabidopsis interspecific hybrids and allopolyploids". Sborník Národní akademie věd Spojených států amerických. 106 (42): 17835–40. Bibcode:2009PNAS..10617835H. doi:10.1073/pnas.0907003106. PMC 2757398. PMID 19805056.
- ^ Mallet J (May 2005). "Hybridization as an invasion of the genome". Trendy v ekologii a evoluci. 20 (5): 229–37. doi:10.1016/j.tree.2005.02.010. PMID 16701374.
- ^ Charlesworth D (April 2016). "Plant Sex Chromosomes". Roční přehled biologie rostlin. 67 (1): 397–420. doi:10.1146/annurev-arplant-043015-111911. PMID 26653795.
- ^ Rieseberg LH (2001). "Chromozomální přesmyky a speciace". Trendy v ekologii a evoluci. 16 (7): 351–358. doi:10.1016 / s0169-5347 (01) 02187-5. ISSN 0169-5347. PMID 11403867.
- ^ Levin DA (listopad 2012). "Dlouhé čekání na hybridní sterilitu v kvetoucích rostlinách". Nový fytolog. 196 (3): 666–70. doi:10.1111 / j.1469-8137.2012.04309.x. PMID 22966819.
- ^ Haldane JB (1922). „Poměr pohlaví a unisexuální sterilita u hybridních zvířat“. Journal of Genetics. 12 (2): 101–109. doi:10.1007 / BF02983075. ISSN 0022-1333.
- ^ Turelli M, Orr HA (květen 1995). „Teorie dominance Haldanovy vlády“. Genetika. 140 (1): 389–402. PMC 1206564. PMID 7635302.
- ^ Runemark A, Eroukhmanoff F, Nava-Bolaños A, Hermansen JS, Meier JI (říjen 2018). „Hybridizace, genomová architektura specifická pro pohlaví a místní adaptace“. Filozofické transakce Královské společnosti v Londýně. Série B, Biologické vědy. 373 (1757): 20170419. doi:10.1098 / rstb.2017.0419. PMC 6125728. PMID 30150218.
- ^ Payseur BA, Rieseberg LH (červen 2016). „Genomická perspektiva hybridizace a speciace“. Molekulární ekologie. 25 (11): 2337–60. doi:10.1111 / mec.13557. PMC 4915564. PMID 26836441.
- ^ Lynch M (1998). Genetika a analýza kvantitativních znaků. Walsh, Bruce, 1957-. Sunderland, Massachusetts: Sinauer. ISBN 0878934812. OCLC 37030646.
- ^ Masly JP, Presgraves DC (září 2007). Barton NH (ed.). „Pitva dvou genů speciace v Drosophile v celém genomu. PLOS Biology. 5 (9): e243. doi:10.1371 / journal.pbio.0050243. PMC 1971125. PMID 17850182.
- ^ Mank JE, Hosken DJ, Wedell N (říjen 2014). „Konflikt na pohlavní chromozomy: příčina, účinek a složitost“. Perspektivy Cold Spring Harbor v biologii. 6 (12): a017715. doi:10.1101 / cshperspect.a017715. PMC 4292157. PMID 25280765.
- ^ Brys R, Vanden Broeck A, Mergeay J, Jacquemyn H (květen 2014). „Příspěvek variace systému páření k reprodukční izolaci u dvou blízce příbuzných druhů Centaurium (Gentianaceae) se zevšeobecněnou morfologií květů“. Vývoj; International Journal of Organic Evolution. 68 (5): 1281–93. doi:10.1111 / evo.12345. PMID 24372301.
- ^ Widmer A, Lexer C, Cozzolino S (leden 2009). "Vývoj reprodukční izolace v rostlinách". Dědičnost. 102 (1): 31–8. doi:10.1038 / hdy.2008.69. PMID 18648386.
- ^ A b Schardl CL, Craven KD (listopad 2003). „Mezidruhová hybridizace u hub a oomycet spojených s rostlinami: přehled“. Molekulární ekologie. 12 (11): 2861–73. doi:10.1046 / j.1365-294x.2003.01965.x. PMID 14629368.
- ^ Levin DA (1975). "Vyloučení menšinových cytotypů u místních populací rostlin". Taxon. 24 (1): 35–43. doi:10.2307/1218997. JSTOR 1218997.
- ^ McCarthy EM, Asmussen MA, Anderson WW (1995). „Teoretické zhodnocení rekombinační speciace“. Dědičnost. 74 (5): 502–509. doi:10.1038 / hdy.1995,71. ISSN 0018-067X.
- ^ Charlton ND, Craven KD, Afkhami ME, Hall BA, Ghimire SR, Young CA (říjen 2014). „Mezidruhová hybridizace a variace bioaktivních alkaloidů zvyšují rozmanitost endofytických druhů Epichloë druhu Bromus laevipes“. Ekologie mikrobiologie FEMS. 90 (1): 276–89. doi:10.1111/1574-6941.12393. PMID 25065688.
- ^ Janko K, Pačes J, Wilkinson-Herbots H, Costa RJ, Roslein J, Drozd P a kol. (Leden 2018). „Hybridní asexualita jako primární postzygotická bariéra mezi rodícími se druhy: O propojení mezi asexualitou, hybridizací a speciací“. Molekulární ekologie. 27 (1): 248–263. doi:10.1111 / mec.14377. PMC 6849617. PMID 28987005.
- ^ Neaves WB, Baumann P (březen 2011). "Unisexuální reprodukce mezi obratlovci". Trendy v genetice. 27 (3): 81–8. doi:10.1016 / j.tig.2010.12.002. PMID 21334090.
- ^ Brochmann C, Brysting AK, Alsos IG, Borgen L, Grundt HH, Scheen AC, Elven R (2004). „Polyploidie v arktických rostlinách“. Biologický žurnál společnosti Linnean. 82 (4): 521–536. doi:10.1111 / j.1095-8312.2004.00337.x.
- ^ Norris LC, Main BJ, Lee Y, Collier TC, Fofana A, Cornel AJ, Lanzaro GC (leden 2015). „Adaptivní introgrese v komáři africké malárie se shoduje se zvýšeným používáním lůžkových sítí ošetřených insekticidy“. Sborník Národní akademie věd Spojených států amerických. 112 (3): 815–20. Bibcode:2015PNAS..112..815N. doi:10.1073 / pnas.1418892112. PMC 4311837. PMID 25561525.
- ^ A b C Marques DA, Meier JI, Seehausen O (červen 2019). „Kombinatorický pohled na speciaci a adaptivní záření“. Trendy v ekologii a evoluci. 34 (6): 531–544. doi:10.1016 / j.strom.2019.02.008. PMID 30885412.
- ^ Maheshwari S, Barbash DA (2011). "Genetika hybridních nekompatibilit". Výroční přehled genetiky. 45 (1): 331–55. doi:10.1146 / annurev-genet-110410-132514. PMID 21910629.
- ^ Buggs RJ, Doust AN, Tate JA, Koh J, Soltis K, Feltus FA a kol. (Červenec 2009). „Ztráta a umlčování genů u Tragopogon miscellus (Asteraceae): srovnání přírodních a syntetických alotetraploidů“. Dědičnost. 103 (1): 73–81. doi:10.1038 / hdy.2009.24. PMID 19277058.
- ^ Jiggins CD, Salazar C, Linares M, Mavarez J (září 2008). "Recenze. Hybridní zvláštnost a Heliconius motýli". Filozofické transakce Královské společnosti v Londýně. Série B, Biologické vědy. 363 (1506): 3047–54. doi:10.1098 / rstb.2008.0065. PMC 2607310. PMID 18579480.
- ^ Fontaine MC, Pease JB, Steele A, Waterhouse RM, Neafsey DE, Sharakhov IV, et al. (Leden 2015). "Genomika komárů. Rozsáhlá introgrese v komplexu vektorových druhů malárie odhalená fylogenomikou". Věda. 347 (6217): 1258524. doi:10.1126 / science.1258524. PMC 4380269. PMID 25431491.
- ^ Jay P, Whibley A, Frézal L, Rodríguez de Cara MÁ, Nowell RW, Mallet J a kol. (Červen 2018). „Evoluce supergenu vyvolaná introgresí chromozomální inverze“. Aktuální biologie. 28 (11): 1839–1845.e3. doi:10.1016 / j.cub.2018.04.072. PMID 29804810.
- ^ Yeaman S (květen 2013). „Genomické přesmyky a vývoj klastrů lokálně adaptivních lokusů“. Sborník Národní akademie věd Spojených států amerických. 110 (19): E1743-51. Bibcode:2013PNAS..110E1743Y. doi:10.1073 / pnas.1219381110. PMC 3651494. PMID 23610436.
- ^ Wu C (2001). "Genický pohled na proces speciace: Genický pohled na proces speciace". Journal of Evolutionary Biology. 14 (6): 851–865. doi:10.1046 / j.1420-9101.2001.00335.x. S2CID 54863357.
- ^ Harrison RG, Larson EL (2014). „Hybridizace, introgrese a povaha hranic druhů“. The Journal of Heredity. 105 Suppl 1 (S1): 795–809. doi:10.1093 / jhered / esu033. PMID 25149255.
- ^ Teeter KC, Payseur BA, Harris LW, Bakewell MA, Thibodeau LM, O'Brien JE a kol. (Leden 2008). „Genomové vzorce toku genů hybridní zónou myší domu“. Výzkum genomu. 18 (1): 67–76. doi:10,1101 / gr. 6757907. PMC 2134771. PMID 18025268.
- ^ Hooper DM, Griffith SC, Price TD (březen 2019). „Inverze pohlavních chromozomů vynucují reprodukční izolaci v ptačí hybridní zóně“. Molekulární ekologie. 28 (6): 1246–1262. doi:10,1111 / mec.14874. PMID 30230092.
