Chemická knihovna kódovaná DNA - DNA-encoded chemical library
Chemické knihovny kódované DNA (DEL) je technologie pro syntéza a promítání v nebývalém rozsahu sbírek malá molekula sloučeniny. DEL se používá v léčivá chemie přemostit pole kombinatorická chemie a molekulární biologie. Cílem technologie DEL je urychlit objev drog proces a zejména činnosti zjišťování v počáteční fázi, jako je validace cíle a identifikace zásahu.
Technologie DEL zahrnuje zkratování chemických sloučenin nebo stavebních bloků DNA fragmenty, které slouží jako identifikační čárové kódy a v některých případech také řídí a řídí chemickou syntézu. Tato technika umožňuje hromadné vytváření a dotazování knihoven pomocí afinitní selekce, typicky na imobilizovaný proteinový cíl. Nedávno byla vyvinuta homogenní metoda screeningu knihoven kódovaných DNA, která využívá technologii emulze voda v oleji k izolaci, počítání a identifikaci jednotlivých komplexů ligand-cíl v přístupu jedné zkumavky. Na rozdíl od konvenčních screeningových postupů, jako jsou vysokovýkonný screening, pro identifikaci pojiva nejsou vyžadovány biochemické testy, což v zásadě umožňuje izolaci pojiv na širokou škálu proteinů, které je historicky obtížné zvládnout konvenčními screeningovými technologiemi. Kromě obecného objevu cílově specifických molekulárních sloučenin tedy dostupnost pojiv na farmakologicky důležité, ale dosud „nezničitelné“ cílové proteiny otevírá nové možnosti vývoje nových léků na nemoci, které dosud nebylo možné léčit. Při eliminaci požadavku na počáteční posouzení aktivity zásahů se doufá a očekává, že mnoho identifikovaných pojiv s vysokou afinitou bude aktivní v nezávislé analýze vybraných zásahů, a proto nabízí efektivní metodu pro identifikaci vysoce kvalitních zásahů a farmaceutických přívodů .
Chemické knihovny kódované DNA a zobrazovací technologie
Až donedávna byla aplikace molekulární evoluce v laboratoři omezena na zobrazovací technologie zahrnující biologické molekuly, kde se nad tímto biologickým přístupem uvažovalo o objevu malých molekul. Společnost DEL otevřela pole zobrazovací technologie, aby zahrnovala nepřirozené sloučeniny, jako jsou malé molekuly, a rozšířila tak použití molekulární evoluce a přirozeného výběru na identifikaci sloučenin s malými molekulami požadované aktivity a funkce. Chemické knihovny kódované DNA mají podobnost s biologickým zobrazením technologie jako technologie fágového displeje protilátky, kvasnicový displej, Zobrazení mRNA a aptamer SELEX. V zobrazení protilátkového fága jsou protilátky fyzicky spojeny s fágovými částicemi, které nesou gen kódující připojenou protilátku, což je ekvivalentní fyzické vazbě „fenotyp „(Protein) a„genotyp “(Gen kódující protein).[1] Fágově zobrazené protilátky lze izolovat z velkých knihoven protilátek napodobováním molekulární evoluce: cykly selekce (na cíli imobilizovaného proteinu), amplifikace a translace.[2]V DEL umožňuje spojení malé molekuly s identifikátorem DNA kód snadnou identifikaci vazebných molekul. Knihovny DEL jsou podrobeny afinitním selekčním postupům na imobilizovaném cílovém proteinu podle výběru, po kterém jsou nevázací látky odstraněny promývacími kroky a vazebné látky mohou být následně amplifikovány polymerázovou řetězovou reakcí (PCR) a identifikovány na základě jejich DNA kódu (např sekvenováním DNA). V technologiích DEL založených na evoluci (viz níže) lze zásahy dále obohatit provedením kol selekce, amplifikace PCR a translace analogicky k biologickým zobrazovacím systémům, jako je displej protilátkového fága. To umožňuje pracovat s mnohem většími knihovnami.
Dějiny
"Syntetizujte vícesložkovou směs sloučenin v jediném procesu a prověřte jej také v jednom procesu". Toto je princip kombinatorické chemie, který vynalezl profesor Furka Á. (Eötvös Loránd University Budapest Hungary) v roce 1982 a popsal to včetně metody syntézy kombinatorických knihoven a metody dekonvoluční strategie v dokumentu notářsky ověřeném ve stejném roce.[3] Motivace, které vedly k vynálezu, byly zveřejněny v roce 2002.[4] DEL jsou kombinované knihovny kódované DNA (DECL) a v jejich aplikaci jednoznačně převažuje kombinatorický princip.

Koncept kódování DNA byl poprvé popsán v teoretické práci Brennera a Lernera v roce 1992, ve které bylo navrženo spojení každé molekuly chemicky syntetizované entity s konkrétní oligonukleotid sekvence konstruovaná paralelně a k použití této kódující genetické značky k identifikaci a obohacení aktivních sloučenin.[5] V roce 1993 představili první praktickou implementaci tohoto přístupu S. Brenner a K. Janda a podobně skupina M.A. Gallop.[6][7] Brenner a Janda navrhli generovat jednotlivé zakódované členy knihovny střídavou paralelou kombinatorická syntéza heteropolymerní chemické sloučeniny a příslušné oligonukleotidové sekvence na stejné kuličce způsobem „split - & - pool“ (viz níže).[6]
Protože nechráněná DNA je omezena na úzké okno konvenčních reakčních podmínek, do konce 90. let se počítalo s řadou alternativních strategií kódování (tj. Založené na MS složené značkování, peptid kódování, haloaromatický tagování, kódování sekundárním aminy, polovodič devices.), hlavně aby se zabránilo nepohodlné syntéze DNA na pevné fázi a aby se vytvořily snadno prověřitelné kombinatorické knihovny s vysokou propustností.[8] Selektivní amplifikovatelnost DNA však velmi usnadňuje screening knihoven a stává se nepostradatelnou pro kódování knihoven organických sloučenin této nebývalé velikosti. V důsledku toho na začátku 2000. let došlo k oživení kombinatorické chemie DNA.
Na začátku tisíciletí bylo zavedeno několik nezávislých vývojových technologií DEL. Tyto technologie lze rozdělit do dvou obecných kategorií: neevoluční a evoluční technologie DEL schopné molekulární evoluce. První kategorie těží ze schopnosti používat běžná činidla, a proto umožňuje poměrně jednoduché generování knihovny. Hity lze identifikovat sekvenováním DNA, avšak pomocí těchto metod není proveditelná translace DNA, a proto molekulární evoluce. Do této kategorie spadají přístupy split a pool vyvinuté výzkumníky ve společnostech Praecis Pharmaceuticals (nyní ve vlastnictví GlaxoSmithKline), Nuevolution (Kodaň, Dánsko) a technologie ESAC vyvinuté v laboratoři prof. Neri (Institute of Pharmaceutical Science, Curych, Švýcarsko) . Technologie ESAC se odlišuje tím, že je kombinačním samo-sestavovacím přístupem, který se podobá objevování fragmentů na základě fragmentů (obr. 1b). Zde DNA hybridizace umožňuje vzorkování diskrétních kombinací stavebních bloků, ale mezi nimi neprobíhá žádná chemická reakce. Příkladem technologií DEL založených na evoluci je směrování DNA vyvinuté Prof.Dr.R. Halpin a Prof. P.B. Harbury (Stanford University, Stanford, CA), DNA-templated syntéza vyvinutá Prof. D. Liu (Harvard University, Cambridge, MA) a komercializován technologií Ensemble Therapeutics (Cambridge, MA) a technologií YoctoReactor.[9] vyvinut a komercializován společností Vipergen (Kodaň, Dánsko). Tyto technologie jsou podrobněji popsány níže. DNA-templovaná syntéza a technologie YoctoReactor vyžadují předchozí konjugaci chemických stavebních bloků (BB) na DNA oligonukleotidovou značku před sestavením knihovny, proto je před sestavením knihovny zapotřebí více práce předem. Navíc DNA značené BB umožňují generování genetického kódu pro syntetizované sloučeniny a je možný umělý překlad genetického kódu: To znamená, že BB lze vyvolat genetickým kódem amplifikovaným pomocí PCR a lze regenerovat sloučeniny knihovny. To zase umožňuje aplikovat princip darwinovského přirozeného výběru a evoluce na výběr malých molekul v přímé analogii s biologickými zobrazovacími systémy; prostřednictvím kol výběru, zesílení a překladu.
Technologie, které nejsou založeny na evoluci
Kombinatorické knihovny
Kombinatorické knihovny jsou speciální vícesložkové směsné směsi, které jsou syntetizovány v jednom postupném procesu. Liší se od kolekce jednotlivých sloučenin i od řady sloučenin připravených paralelní syntézou. Kombinované knihovny mají důležité rysy.
„Směsi se používají při jejich syntéze. Použití směsí zajišťuje velmi vysokou účinnost procesu. Oba reaktanty mohou být směsi, ale z praktických důvodů se používá postup split-mix: jedna směs se rozdělí na části, které jsou spojeny s BB.[10][11] Směsi jsou tak důležité, že neexistuje kombinatorická knihovna bez použití směsi při syntéze, a pokud je směs použita v procesu, nevyhnutelně se kombinatorická knihovna formuje.
„Komponenty knihoven musí být přítomny v téměř stejném molárním množství. Aby se toho dosáhlo co nejblíže, jsou směsi rozděleny na stejné části a po smíchání je nutné důkladné promíchání.
„Jelikož struktura komponent není známa, je při screeningu nutné použít metody dekonvoluce. Z tohoto důvodu byly vyvinuty metody kódování. Kódující molekuly jsou připojeny k kuličkám pevného podkladu, které zaznamenávají spojené BB a jejich sekvenci. Jednou z těchto metod je kódování DNA oligomery.
„Je pozoruhodnou vlastností kombinatorických knihoven, že lze celou směsnou směs prověřovat v jediném procesu.
Protože jak syntéza, tak screening jsou velmi účinné postupy, vede použití kombinatorických knihoven ve farmaceutickém výzkumu k enormním úsporám.
V kombinatorické syntéze na pevné fázi se v každé kuličce tvoří pouze jedna sloučenina. Z tohoto důvodu nemůže počet komponent v knihovně překročit počet perliček pevné podpory. To znamená, že počet komponent v takových knihovnách je omezený. Toto omezení bylo zcela vyloučeno Harburym a Halpinem. Při jejich syntéze DEL je pevný nosič vynechán a BB jsou připojeny přímo ke kódujícím DNA oligomerům.[12] Tento nový přístup pomáhá prakticky neomezeně zvyšovat počet složek kombinovaných knihoven kódovaných DNA (DECL).
Split - & - Pool DNA kódování
Aby bylo možné se přihlásit kombinatorická chemie pro syntézu chemických knihoven kódovaných DNA byl použit přístup Split - & - Pool.[10][11] Zpočátku sada jedinečných DNA-oligonukleotidy (n) každá obsahující specifickou kódující sekvenci je chemicky konjugována s odpovídající sadou malých organických molekul. V důsledku toho oligonukleotid -konjugované sloučeniny jsou smíchány ("Pool") a rozděleny ("Split") do několika skupin (m). Za vhodných podmínek je druhá sada stavebních bloků (m) spojena s první a další oligonukleotid který kóduje druhou modifikaci, se před opětovným mícháním enzymaticky zavede. Tento „split - & - pool“ krok může být několikrát iterován (r) zvyšování v každém kole kombinačním způsobem velikost knihovny (tj. (n X m)r). Alternativně byly peptidové nukleové kyseliny použity ke kódování knihoven připravených metodou "split - & - pool".[13] Výhodou kódování PNA je, že chemii lze provádět standardním SPPS.[14]
Postupné navázání kódujících fragmentů DNA na rodící se organické molekuly

Slibnou strategii pro konstrukci knihoven kódovaných DNA představuje použití multifunkčních stavebních bloků kovalentně konjugovaný s oligonukleotid slouží jako „základní struktura“ pro syntézu knihovny. Sada „multifunkčních lešení“ podléhá ortogonálním reakcím se sérií vhodných reaktivních partnerů. Po každém reakčním kroku je identita modifikace kódována enzymatickým přidáním segmentu DNA k původní „základní struktuře“ DNA.[15][16] Použití N-chráněný aminokyseliny kovalentně připojené k fragmentu DNA umožňují po vhodném kroku deprotekce další amidová vazba formace s řadou karboxylové kyseliny nebo a redukční aminace s aldehydy. Podobně, dien karboxylové kyseliny používané jako lešení pro konstrukci knihovny na 5'-konci aminoskupiny oligonukleotid, mohl být vystaven a Diels-Alder reakce s různými maleimid deriváty. Po dokončení požadovaného reakčního kroku se přidala identita chemické skupiny oligonukleotid je zřízen žíhání částečně doplňkové oligonukleotid a následným Klenow vyplnit DNA polymerace, čímž se získá dvouvláknový fragment DNA. Syntetické a kódovací strategie popsané výše umožňují snadnou konstrukci knihoven kódovaných DNA až do velikosti 104 členské sloučeniny nesoucí dvě sady „stavebních bloků“. Postupné přidávání alespoň tří nezávislých sad chemických skupin do stavebního bloku trifunkčního jádra pro konstrukci a kódování velmi velké knihovny kódované DNA (obsahující až 106 lze také předpokládat.[15](Obr)
Kombinatorické samo sestavení
Zakódované samo-sestavující se chemické knihovny

Encoded SElf-Asmíchání Chemický (ESAC) Knihovny se spoléhají na princip, že dvě dílčí knihovny o velikosti X členů (např3) obsahující konstantní komplementární hybridizační doménu, může po hybridizaci se složitostí poskytnout výnos kombinační DNA-duplexní knihovny X2 rovnoměrně zastoupení členové knihovny (např.106).[17] Každý člen dílčí knihovny by sestával z oligonukleotid obsahující variabilní kódující oblast ohraničenou konstantní DNA sekvencí, nesoucí vhodnou chemickou modifikaci na konci oligonukleotidu.[17] The ESAC dílčí knihovny lze použít v nejméně čtyřech různých provedeních.[17]
- Subknihovna může být spárována s komplementárním oligonukleotidem a použita jako knihovna kódovaná DNA zobrazující jedinou kovalentně spojenou sloučeninu pro afinitní selekční experimenty.
- Subknihovna může být spárována s oligonukleotidem zobrazujícím známé pojivo k cíli, což umožňuje strategie afinitního zrání.
- Dvě jednotlivé dílčí knihovny lze kombinatoricky sestavit a použít pro de novo identifikace vazebných molekul bindentate.
- Lze sestavit tři různé dílčí knihovny a vytvořit tak kombinatorickou knihovnu triplexů.
Preferenční pojiva izolovaná z afinitního výběru mohou být PCR-amplifikováno a dekódováno na doplňkovém oligonukleotid mikročipy[18] nebo zřetězením kódů, subklonování a sekvenování.[17] Jednotlivé stavební bloky mohou být případně konjugovány za použití vhodných linkerů za vzniku vysoce afinitní sloučeniny podobné léčivu. Vlastnosti linkeru (např. Délka, pružnost, geometrie, chemická podstata a rozpustnost) ovlivňují vazebná afinita a chemické vlastnosti výsledného pojiva. (Obr)
Pokusy o biologické rýžování HSA 600členného ESAC knihovna umožnila izolaci 4- (str-jodfenyl) butanová skupina. Sloučenina představuje základní strukturu řady přenosných albumin vazebných molekul a Albufluoru, který byl nedávno vyvinut fluorescein angiografický kontrastní látka v současné době pod klinickým hodnocením.[19]
ESAC technologie byla použita pro izolaci silných inhibitory skotu trypsin a pro identifikaci románu inhibitory z stromelysin-1 (MMP-3 ), matrix metaloproteináza účastnící se jak fyziologických, tak patologických procesů remodelace tkání, jakož i chorobných procesů, jako je artritida a metastáza.[20]
Technologie založené na evoluci
Směrování DNA
V roce 2004 D.R. Halpin a P.B. Harbury představil novou zajímavou metodu pro konstrukci knihoven kódovaných DNA. Poprvé sloužily šablony konjugované s DNA jak pro kódování, tak pro programování infrastruktury syntézy knihovních komponent „split - & - pool“.[21] Návrh Halpina a Harburyho umožnil střídání kol výběru, Amplifikace PCR a diverzifikace s malými organickými molekulami, zcela analogicky k fágový displej technologie. Stroj pro směrování DNA se skládá z řady spojených sloupců nesoucích antikodony vázané na pryskyřici, které by mohly sekvenčně specificky oddělit populaci šablon DNA na prostorově odlišná místa pomocí hybridizace.[21] Podle tohoto protokolu split-and-pool a peptid kombinatorická knihovna DNA kódovaná 106 členové byli vygenerováni.[22]
Syntéza založená na DNA

V roce 2001 David Liu a spolupracovníci prokázali tuto komplementární DNA oligonukleotidy lze použít k podpoře určitých syntetických látek reakce, které se efektivně nekonají v řešení na nízké úrovni koncentrace.[23][24] K urychlení reakce mezi chemickými skupinami zobrazenými na koncích dvou řetězců DNA byl použit DNA-heteroduplex. Dále se ukázalo, že „efekt blízkosti“, který urychluje bimolekulární reakci, je nezávislý na vzdálenosti (alespoň ve vzdálenosti 30 nukleotidy ).[23][24] Sekvenčně programovaným způsobem byly oligonukleotidy nesoucí jednu skupinu chemických reaktantů hybridizován na komplementární oligonukleotidové deriváty nesoucí jinou reaktivní chemickou skupinu. Blízkost poskytnutá hybridizací DNA drasticky zvyšuje efektivní molarita reakčních činidel připojených k oligonukleotidům, což umožňuje, aby k požadované reakci došlo i ve vodném prostředí v koncentracích, které jsou o několik řádů nižší než koncentrace potřebné pro odpovídající konvenční organickou reakci, která není chráněna DNA.[25] Pomocí nastavení templátu DNA a sekvenčně naprogramované syntézy Liu a spolupracovníci vytvořili 64člennou sloučeninu DNA kódovanou knihovnu makrocykly.[26]
3-dimenzionální technologie založená na blízkosti (technologie YoctoReactor)
YoctoReactor (yR) je 3D přístup založený na přiblížení, který využívá samoobslužnou povahu DNA oligonukleotidů do 3, 4 nebo 5 cest, aby nasměroval syntézu malých molekul do středu spojení. Obrázek 5 ilustruje základní koncept se čtyřcestným spojením DNA.
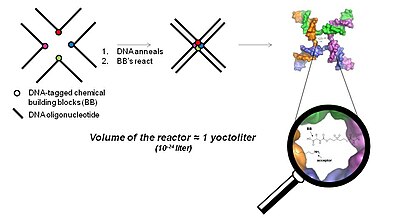
Střed spojení DNA tvoří svazek v řádu a yoktolitr, odtud název YoctoReactor. Tento objem obsahuje reakci s jedinou molekulou, která poskytuje reakční koncentrace ve vysokém rozmezí mM. Efektivní koncentrace usnadněná DNA výrazně urychluje chemické reakce, které by jinak při skutečné koncentraci neproběhly o několik řádů níže.
Budování knihovny yR
Obrázek 6 ilustruje generování knihovny yR pomocí třícestného spojení DNA.
Stručně řečeno, chemické stavební bloky (BB) jsou připojeny prostřednictvím štěpitelných nebo neštěpitelných linkerů ke třem typům bispecifických DNA oligonukleotidů (oligo-BB) představujících každé rameno yR. Pro usnadnění kombinatorické syntézy jsou oligo-BB navrženy tak, že DNA obsahuje (a) kód pro připojený BB na distálním konci oligo (barevné čáry) a (b) oblasti konstantní DNA sekvence (černá řádky), aby došlo k vlastnímu sestavení DNA do třícestného spojení (nezávisle na BB) a následné chemické reakci. Chemické reakce se provádějí po krocích a po každém kroku se DNA liguje a produkt se čistí elektroforézou na polyakryamidovém gelu. Odštěpitelné linkery (BB-DNA) se používají pro všechny polohy kromě jedné, čímž se získá knihovna malých molekul s jedinou kovalentní vazbou na kód DNA. Tabulka 1 uvádí, jak lze pomocí technologie yR generovat knihovny různých velikostí.

Přístup yR design poskytuje neměnné reakční místo s ohledem na (a) vzdálenost mezi reaktanty a (b) prostředí sekvence obklopující reakční místo. Kromě toho důvěrné spojení mezi kódem a BB na oligo-BB částech, které jsou kombinatoricky smíchány v jednom hrnci, poskytuje vysokou věrnost kódování knihovny. Kód syntetizovaných produktů dále není přednastaven, ale je kombinován kombinovaně a syntetizován synchronně s vrozeným produktem.
Homogenní screening knihoven yoctoreactor
Nedávno byla vyvinuta homogenní metoda screeningu knihoven yoctoreactor (yR), která využívá technologii emulze voda v oleji k izolaci jednotlivých komplexů ligand-cíl. Volané obohacení o vazebnou pastu (BTE), ligandy k proteinovému cíli jsou identifikovány zachycením vazebných párů (DNA-značený proteinový cíl a yR ligand) v kapičkách emulze během kinetiky dominující disociaci. Jakmile jsou cíl a DNA ligandu zachyceny, jsou spojeny ligací, čímž jsou zachovány informace o vazbě.
Dále bude identifikace zásahů v zásadě počítáním: informace o vazebných událostech se dešifrují sekvenováním a počítáním spojené DNA - selektivní pojiva se počítají s mnohem vyšší frekvencí než náhodná pojiva. To je možné, protože náhodné zachycení cíle a ligandu je „zředěno“ vysokým počtem vodních kapiček v emulzi. Nízký šum a charakteristika pozadí signálu BTE se připisují „zředění“ náhodného signálu, nedostatku povrchových artefaktů a vysoké věrnosti knihovny yR a screeningové metody. Screening se provádí metodou jedné trubice. Biologicky aktivní zásahy jsou identifikovány v jednom kole BTE charakterizované nízkou mírou falešně pozitivních výsledků.
BTE napodobuje nerovnovážnou povahu interakcí ligand-cíl in vivo a nabízí jedinečnou možnost skríningu cílových specifických ligandů na základě doby zdržení ligand-cíl, protože emulze, která zachycuje vazebný komplex, se tvoří během fáze dynamické disociace.
Dekódování chemických knihoven kódovaných DNA
Po výběru z chemických knihoven kódovaných DNA je strategie dekódování pro rychlou a účinnou identifikaci specifických vazebných sloučenin zásadní pro další vývoj DEL technologie. Zatím, Sangerovo sekvenování - dekódování na základě, microarray metodika založená na a vysoce výkonné sekvenování techniky představovaly hlavní metodiky pro dekódování výběrů knihoven kódovaných DNA.
Dekódování na základě Sangerova sekvenování
Ačkoli mnoho autorů implicitně předpokládalo tradiční Sangerovo sekvenování - dekódování na základě,[6][7][17][22][26] počet sekvenčních kódů jednoduše podle složitosti knihovny je pro tradiční rozhodně nereálný úkol Sangerovo sekvenování přístup. Provádění Sangerovo sekvenování jako první byl popsán dekódování chemických knihoven kódovaných DNA vysoce výkonným způsobem.[17] Po výběru a Amplifikace PCR z DNA značek knihovních sloučenin byly generovány konkatamery obsahující více kódujících sekvencí a ligován do vektor. Následující Sangerovo sekvenování reprezentativního čísla výsledného kolonie odhalily frekvence kódů přítomných ve vzorku knihovny kódované DNA před a po výběru.[17]
Dekódování na základě microarray
DNA microarray je zařízení pro vysoce výkonné vyšetřování široce používané v molekulární biologie a v lék. Skládá se z řady mikroskopických skvrn („funkcí“ nebo „míst“) obsahujících několik pikomolů oligonukleotidy nesoucí specifickou sekvenci DNA. Může to být krátký úsek a gen nebo jiný prvek DNA, který se používá jako sondy hybridizovat DNA nebo RNA vzorek za vhodných podmínek. Terč sondy hybridizace je obvykle detekována a kvantifikována pomocí fluorescence - detekce na základě fluorofor -značené cíle k určení relativní hojnosti cíle nukleová kyselina sekvence. Microarray byl použit pro úspěšné dekódování knihoven kódovaných DNA ESAC[17] a knihovny kódované PNA.[27] Kódování oligonukleotidy představující jednotlivé chemické sloučeniny v knihovně, jsou tečkované a chemicky navázané na microarray sklíčka pomocí robota BioChip Arrayer. Následně oligonukleotid značky vazebných sloučenin izolovaných z výběru jsou PCR amplifikováno používat fluorescenční primer a hybridizován na DNA-microarray skluzavka. Později, mikročipy jsou analyzovány pomocí a laser detekovány a kvantifikovány intenzity skenování a spotu. Obohatení preferenčních vazebných sloučenin je odhaleno porovnáním intenzity skvrn DNA-microarray snímek před a po výběru.[17]
Dekódování pomocí vysoce výkonného sekvenování
Podle složitosti chemické knihovny kódované DNA (obvykle mezi 103 a 106 členů), konvenční Sangerovo sekvenování založené na dekódování je nepravděpodobné, že bude v praxi použitelné, a to jak kvůli vysokým nákladům na základnu pro sekvenování, tak kvůli zdlouhavému postupu.[28] Vysoce výkonné sekvenování technologie využívají strategie, které paralelizují proces sekvenování a nahrazují použití kapilární elektroforéza a produkovat tisíce nebo miliony sekvencí najednou. V roce 2008 byla popsána první implementace a vysoce výkonné sekvenování technika původně vyvinutá pro sekvenování genomu (tj. „454 technologie ") k rychlému a efektivnímu dekódování chemické knihovny kódované DNA obsahující 4 000 sloučenin.[15] Tato studie vedla k identifikaci nových chemických sloučenin s submikromolárními disociační konstanty vůči streptavidin a rozhodně prokázal proveditelnost konstrukce, provádění selekcí a dekódování knihoven kódovaných DNA obsahujících miliony chemických sloučenin.[15]
Viz také
Reference
- ^ Smith GP (červen 1985). "Filamentous fusion phage: new expression vectors that display cloned antigens on the virion surface". Věda. 228 (4705): 1315–7. Bibcode:1985Sci ... 228.1315S. doi:10.1126 / science.4001944. PMID 4001944.
- ^ Hoogenboom HR (2002). "Přehled technologie protilátkového fágového displeje a jejích aplikací". Fágový displej protilátky. Metody v molekulární biologii. 178. s. 1–37. doi:10.1385/1-59259-240-6:001. ISBN 978-1-59259-240-1. PMID 11968478.
- ^ Furka Á. Tanulmány, gyógyászatilag hasznosítható peptidek szisztematikus felkutatásának lehetőségéről. Studie o možnosti systematického vyhledávání farmaceuticky užitečných peptidů) https://mersz.hu/mod/object.php?objazonosito=matud202006_f42772_i2
- ^ Furka Á (2002). Combinatorial Chemistry 20 years on .., Drug DiscovToday 7; 1-4.
- ^ Brenner S, Lerner RA (červen 1992). "Kódovaná kombinatorická chemie". Sborník Národní akademie věd Spojených států amerických. 89 (12): 5381–3. Bibcode:1992PNAS ... 89.5381B. doi:10.1073 / pnas.89.12.5381. PMC 49295. PMID 1608946.
- ^ A b C Nielsen J, Brenner S, Janda KD (1993). "Syntetické metody pro implementaci kódované kombinatorické chemie". Journal of the American Chemical Society. 115 (21): 9812–9813. doi:10.1021 / ja00074a063.
- ^ A b Needels MC, Jones DG, Tate EH, Heinkel GL, Kochersperger LM, Dower WJ, Barrett RW, Gallop MA (listopad 1993). "Generování a screening knihovny syntetických peptidů kódovaných oligonukleotidy". Sborník Národní akademie věd Spojených států amerických. 90 (22): 10700–4. Bibcode:1993PNAS ... 9010700N. doi:10.1073 / pnas.90.22.10700. PMC 47845. PMID 7504279.
- ^ Mukund S. Chorghade (2006). Objev a vývoj drog. New York: Wiley-Interscience. str. 129–167. ISBN 978-0-471-39848-6.
- ^ Heitner TR, Hansen NJ (listopad 2009). „Zefektivnění objevování a optimalizace zásahů pomocí DNA reaktoru v měřítku yoctoliter“. Odborné stanovisko k objevu drog. 4 (11): 1201–13. doi:10.1517/17460440903206940. PMID 23480437.
- ^ A b A. Furka, F. Sebestyén, M. Asgedom, G. Dibó, hojnost peptidů syntézou In Highlights of Modern Biochemistry, Proceedings of the 14. International Congress of Biochemistry, VSP. Utrecht, Nizozemsko, 1988, sv. 5, s. 47.
- ^ A b Furka Á, Sebestyén F, Asgedom M, Dibó G (1991) Obecná metoda pro rychlou syntézu vícesložkových peptidových směsí. Int J Peptide Protein Res 37; 487-93.
- ^ Harbury DR, Halpin DR (2000) WO 00/23458.
- ^ Winssinger N, Damoiseaux R, Tully DC, Geierstanger BH, Burdick K, Harris JL (říjen 2004). „PNA-kódovaná proteázová substrátová mikročipy“. Chemie a biologie. 11 (10): 1351–60. doi:10.1016 / j.chembiol.2004.07.015. PMID 15489162.
- ^ Zambaldo C, Barluenga S, Winssinger N (červen 2015). "Chemické knihovny kódované PNA". Aktuální názor na chemickou biologii. 26: 8–15. doi:10.1016 / j.cbpa.2015.01.005. PMID 25621730.
- ^ A b C d Mannocci L, Zhang Y, Scheuermann J, Leimbacher M, De Bellis G, Rizzi E, Dumelin C, Melkko S, Neri D (listopad 2008). „Vysoce výkonné sekvenování umožňuje identifikaci vazebných molekul izolovaných z chemických knihoven kódovaných DNA“. Sborník Národní akademie věd Spojených států amerických. 105 (46): 17670–5. Bibcode:2008PNAS..10517670M. doi:10.1073 / pnas.0805130105. PMC 2584757. PMID 19001273.
- ^ Buller F, Mannocci L, Zhang Y, Dumelin CE, Scheuermann J, Neri D (listopad 2008). „Návrh a syntéza nové chemické knihovny kódované DNA pomocí cykloadic Diels-Alder“. Dopisy o bioorganické a léčivé chemii. 18 (22): 5926–31. doi:10.1016 / j.bmcl.2008.07.038. PMID 18674904.
- ^ A b C d E F G h i Melkko S, Scheuermann J, Dumelin CE, Neri D (květen 2004). "Zakódované samo-sestavující se chemické knihovny". Přírodní biotechnologie. 22 (5): 568–74. doi:10.1038 / nbt961. PMID 15097996.
- ^ Lovrinovic M, Niemeyer CM (květen 2005). "DNA mikročipy jako dekódovací nástroje v kombinatorické chemii a chemické biologii". Angewandte Chemie. 44 (21): 3179–83. doi:10.1002 / anie.200500645. PMID 15861437.
- ^ Dumelin CE, Trüssel S, Buller F, Trachsel E, Bootz F, Zhang Y, Mannocci L, Beck SC, Drumea-Mirancea M, Seeliger MW, Baltes C, Müggler T, Kranz F, Rudin M, Melkko S, Scheuermann J, Neri D (2008). "A portable albumin binder from a DNA-encoded chemical library". Angewandte Chemie. 47 (17): 3196–201. doi:10.1002/anie.200704936. PMID 18366035.
- ^ Melkko S, Zhang Y, Dumelin CE, Scheuermann J, Neri D (2007). "Isolation of high-affinity trypsin inhibitors from a DNA-encoded chemical library". Angewandte Chemie. 46 (25): 4671–4. doi:10.1002/anie.200700654. PMID 17497616.
- ^ A b Halpin DR, Harbury PB (July 2004). "DNA display I. Sequence-encoded routing of DNA populations". PLOS Biology. 2 (7): E173. doi:10.1371/journal.pbio.0020173. PMC 434148. PMID 15221027.

- ^ A b Halpin DR, Harbury PB (July 2004). "DNA display II. Genetic manipulation of combinatorial chemistry libraries for small-molecule evolution". PLOS Biology. 2 (7): E174. doi:10.1371/journal.pbio.0020174. PMC 434149. PMID 15221028.

- ^ A b Gartner ZJ, Liu DR (July 2001). "The generality of DNA-templated synthesis as a basis for evolving non-natural small molecules". Journal of the American Chemical Society. 123 (28): 6961–3. doi:10.1021/ja015873n. PMC 2820563. PMID 11448217.
- ^ A b Calderone CT, Puckett JW, Gartner ZJ, Liu DR (November 2002). "Directing otherwise incompatible reactions in a single solution by using DNA-templated organic synthesis". Angewandte Chemie. 41 (21): 4104–8. doi:10.1002/1521-3773(20021104)41:21<4104::AID-ANIE4104>3.0.CO;2-O. PMID 12412096.
- ^ Li X, Liu DR (September 2004). "DNA-templated organic synthesis: nature's strategy for controlling chemical reactivity applied to synthetic molecules". Angewandte Chemie. 43 (37): 4848–70. doi:10.1002/anie.200400656. PMID 15372570.
- ^ A b Gartner ZJ, Tse BN, Grubina R, Doyon JB, Snyder TM, Liu DR (September 2004). "DNA-templated organic synthesis and selection of a library of macrocycles". Věda. 305 (5690): 1601–5. Bibcode:2004Sci...305.1601G. doi:10.1126/science.1102629. PMC 2814051. PMID 15319493.
- ^ Harris J, Mason DE, Li J, Burdick KW, Backes BJ, Chen T, Shipway A, Van Heeke G, Gough L, Ghaemmaghami A, Shakib F, Debaene F, Winssinger N (October 2004). "Activity profile of dust mite allergen extract using substrate libraries and functional proteomic microarrays". Chemie a biologie. 11 (10): 1361–72. doi:10.1016/j.chembiol.2004.08.008. PMID 15489163.
- ^ Sanger F, Nicklen S, Coulson AR (December 1977). "DNA sequencing with chain-terminating inhibitors". Sborník Národní akademie věd Spojených států amerických. 74 (12): 5463–7. Bibcode:1977PNAS...74.5463S. doi:10.1073/pnas.74.12.5463. PMC 431765. PMID 271968.
