Horizontální přenos genů v evoluci - Horizontal gene transfer in evolution

Vědci, kteří se pokoušejí rekonstruovat evoluční historii, byli zpochybněni skutečností, že geny se někdy mohou přenášet mezi vzdálenými větvemi na strom života. K tomuto pohybu genů může dojít prostřednictvím horizontální přenos genů (HGT), zakódování informací, na které se biologové spoléhali při rekonstrukci fylogeneze organismů. Naopak, HGT může také pomoci vědcům rekonstruovat a datovat strom života. Ve skutečnosti lze genový přenos použít jako fylogenetický marker nebo jako důkaz soudobosti dárcovských a přijímajících organismů a jako stopa vyhynulé biologické rozmanitosti.
HGT se děje velmi zřídka - na úrovni jednotlivých organismů je velmi nepravděpodobné, že by k takové události došlo. Na velkém rozsahu evoluční historie se však tyto události vyskytují s určitou pravidelností. Na jedné straně to nutí biologové, aby upustili od používání jednotlivých genů jako dobrých značek pro historii života. Na druhou stranu to poskytuje téměř nevyužitý velký zdroj informací o minulosti.
Tři oblasti života
Tři hlavní rané větve stromu života byly intenzivně studovány mikrobiologové protože první organismy byly mikroorganismy. Mikrobiologové (pod vedením Carl Woese ) zavedli termín doména pro tři hlavní větve tohoto stromu, kde doména je fylogenetický termín podobný ve smyslu biologickému království. K rekonstrukci tohoto stromu života, genové sekvence kódující malou podjednotku ribozomální RNA (SSU rRNA, 16s rRNA ) se osvědčil a strom (jak je znázorněno na obrázku) se do značné míry spoléhá na informace z tohoto jediného genu.
Tyto tři domény života představují hlavní evoluční linie raného buněčného života a v současné době zahrnují Bakterie, Archaea (jednobuněčné organismy povrchně podobné bakteriím) a Eukarya. Eukarya zahrnuje pouze organismy, které mají dobře definované jádro, jako např houby, protistů a všechny organismy v rostlinné a živočišné říši (viz obrázek).
Gen nejčastěji používaný pro konstrukci fylogenetických vztahů v mikroorganismy je malá podjednotka ribozomální RNA gen, protože jeho sekvence mají tendenci být konzervovány mezi členy s blízkými fylogenetickými vzdálenostmi, přesto dostatečně variabilní, aby bylo možné měřit rozdíly.[1]SSR rRNA jako měřítko evolučních vzdáleností byla průkopníkem Carl Woese při formulování prvního moderního „stromu života“ a jeho výsledky ho vedly k návrhu Archaea jako třetí doména život. Nedávno se však tvrdilo, že geny rRNA SSU lze také přenášet vodorovně.[2] I když to může být vzácné, tato možnost si vynucuje kontrolu platnosti fylogenetických stromů na základě SSR rRNA.
Nedávné objevy „nekontrolovatelného“ HGT v mikroorganismech a detekce horizontálního pohybu rovnoměrných genů pro malou podjednotku ribozomální RNA přinutily biology zpochybnit přesnost alespoň raných větví stromu a dokonce zpochybnit platnost stromy jako užitečné modely toho, jak probíhá raná evoluce.[3] Ve skutečnosti se má za to, že k rané evoluci došlo od komunity progenoty, schopný vyměňovat velké molekuly, když byl standardem HGT. K tomuto laterálnímu přenosu genů došlo také za Darwinovský práh po zjištění dědičnosti nebo vertikálního přenosu genů.[4][5]
„Porovnání sekvencí naznačuje nedávný horizontální přenos mnoha geny mezi různorodými druh včetně přes hranice fylogenetické "domény". Stanovení fylogenetické historie druhu tedy nelze provést přesvědčivě stanovením evolučních stromů pro jednotlivé geny. “ [6] HGT je tedy potenciál matoucí faktor vyvozovat fylogenetické stromy z sekvence jednoho gen. Například pokud si dvě vzdáleně příbuzné bakterie vyměnily gen, a fylogenetický strom včetně těchto druhů jim ukáže, že spolu úzce souvisejí, i když se většina ostatních genů podstatně rozcházela. Z tohoto důvodu je důležité použít jiné informace k odvození fylogenezí, jako je přítomnost nebo nepřítomnost genů, nebo častěji zahrnout co nejširší škálu genů pro analýzu.
Výběr metafory: strom, síť, pavučina nebo prsten
Ve svém článku Vykořenění stromu života, W. Ford Doolittle pojednává o Poslední univerzální společný předek - kořen Stromu života - a problémy s tímto konceptem, které představuje HGT.[7] Popisuje mikroorganismus Archaeoglobus fulgidus jako anomálie s ohledem na a fylogenetické strom založený na kódu pro enzym HMGCoA reduktáza - organismus je určitě archaean se všemi buněčnými lipidy a transkripčními stroji, které se od archaeanu očekávají, ale jeho geny HMGCoA jsou bakteriálního původu. V článku Doolittle říká, že i když je nyní široce přijímáno, že mitochondrie v eukaryotech odvozených z alfa-proteobakteriálních buněk a podobně chloroplasty pochází z požití sinice,
„... již nelze bezpečně předpokládat, že to byly jediné postranní přenosy genů, ke kterým došlo po vzniku prvních eukaryot. Pouze v pozdějších vícebuněčných eukaryotech známe určit definitivní omezení horizontální genové výměny, jako je příchod oddělených ( a chráněné) zárodečné buňky...
Pokud by nikdy nedošlo k laterálnímu přenosu genů, měly by všechny tyto jednotlivé genové stromy stejnou topologii (stejné pořadí větvení) a geny předků v kořenu každého stromu by byly všechny přítomny v posledním univerzálním společném předku, a jediná starodávná buňka. Ale rozsáhlý přenos znamená, že tomu tak není: genové stromy se budou lišit (i když mnohé budou mít oblasti podobné topologie) a nikdy by neexistovala jediná buňka, kterou lze nazvat posledním univerzálním společným předkem ... “
Doolittle navrhl, že univerzálním společným předkem nemohl být jeden konkrétní organismus, ale musel to být volný a rozmanitý konglomerát primitivních buněk, které se vyvinuly společně. Tyto rané buňky, z nichž každá má relativně málo genů, se v mnoha ohledech lišily a vyměňovaly si je geny volně. Nakonec z těchto eklektických buněk vzešly tři domény života, jak je známe dnes: bakterie, archaea a eukaryot. Tyto domény jsou nyní rozeznatelně odlišné, protože velká část přenosu genů, která stále probíhá, je spíše v těchto doménách než mezi nimi. Biolog Peter Gogarten posílil tyto argumenty a navrhl, že metafora stromu neodpovídá údajům z nedávného výzkumu genomu, a že by biologové měli místo toho použít „metaforu mozaiky k popisu různých dějin kombinovaných v jednotlivých genomech a použít [metaforu] síť pro vizualizaci bohaté výměny a kooperativních účinků HGT mezi mikroby. “[8]
Řešení nejistoty pomocí fylogenomiky
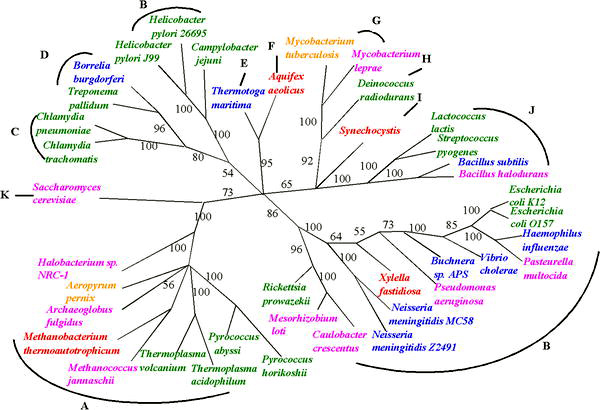
Navzdory nejistotám při rekonstrukci fylogenií zpět do počátků života dochází k pokroku v rekonstrukci stromu života tváří v tvář nejistotám vyvolaným HGT. Nejistotu jakéhokoli odvozeného fylogenetického stromu založeného na jediném genu lze vyřešit použitím několika běžných genů nebo dokonce důkazů z celých genomů.[10] Jeden takový přístup, někdy nazývaný „multi-locus typing“, byl použit k odvození fylogenních stromů pro organismy, které si vyměňují geny, jako jsou bakterie meningitidy.[11]
Jonathan Eisen a Claire Fraser poukázali na to, že:
„Při budování stromu života začala analýza celých genomů doplňovat a v některých případech i vylepšovat studie dříve provedené s jedním nebo několika geny. Například nedávné studie úplných bakteriálních genomů naznačují, že hypertermofilní druhy nejsou hluboce rozvětvené; pokud je to pravda, zpochybňuje to představu, že prvními formami života byly termofily. Analýza genomu eukaryotického parazita Encephalitozoon cuniculi podporuje návrhy, které skupina Microsporidia nejsou hluboce se rozvětvujícími protisty, ale ve skutečnosti jsou členy říše hub. Analýza genomu může dokonce pomoci vyřešit vztahy uvnitř druhů, například poskytnutím nových genetických markerů pro populační genetické studie u bakterií způsobujících antrax nebo tuberkulózu. Ve všech těchto studiích jsou to další data poskytovaná kompletní sekvencí genomu, která umožňuje oddělit fylogenetický signál od šumu. Tím nechci říci, že strom života je nyní vyřešen - odebrali jsme pouze vzorky genomu a mnoho skupin se toho ještě nedotklo “[12]
Tyto přístupy umožňují odhady relativní frekvence HGT; relativně nízké hodnoty, které byly pozorovány, naznačují, že „strom“ je stále platnou metaforou evoluce - ale strom zdobí „pavučiny“ horizontálně přenesených genů. Toto je hlavní závěr studie z roku 2005 o více než 40 úplných mikrobiálních genomových sekvencích od Fan Ge, Li-San Wang a Junhyong Kim. Odhadují frekvenci HGT událostí na přibližně 2% základních genů na genom.[13] Podobné přístupy k hodnocení evoluce celého genomu také umožňují pokrok v identifikaci velmi raných událostí ve stromu života, například návrh, že eukaryoty vznikly fúzí dvou úplných, ale velmi rozmanitých prokaryotických genomů: jednoho z bakterie a druhého z archaální buňky .[3]
Taková fúze organismů hypotéza protože původ složitých jaderných buněk byl předložen Lynn Margulis pomocí zcela odlišného uvažování o symbióza mezi bakterií a archaenem vznikajícím ve starověkém konsorciu mikrobů.[14]
Využití horizontálního přenosu pro evoluční studie
Zatímco HGT je často vnímána jako výzva pro rekonstrukci stromu života, alternativní pohled je, že naopak poskytuje další cenné informace pro jeho rekonstrukci.
Za prvé, pro organismus příjemce je HGT mutace DNA jako ostatní, a jako taková může být modelována a použita při rekonstrukci a zakořenění stromu.[15]
Zadruhé je nutné, aby příjemce získávání genů HGT žil současně nebo v postranní době jako dárce.[16] V důsledku toho existuje informace o načasování diverzifikace v HGT.[17] To je o to pozoruhodnější, že hlavní obvyklý zdroj datování v živém světě, fosilní záznam, chybí právě tam, kde je HGT v mikrobiálním světě hojný.
Za třetí poskytuje informace o zaniklé biologické rozmanitosti, protože přenosy jsou pravděpodobně z vyhynulých druhů.[18]
Viz také
Reference
- ^ Woese C a kol. (1990). „Směrem k přirozenému systému organismů: návrh domén Archaea, Bacteria a Eucarya“. PNAS USA. 87 (12): 4576–9. Bibcode:1990PNAS ... 87,4576 W.. doi:10.1073 / pnas.87.12.4576. PMC 54159. PMID 2112744.
- Woese C, Fox G (1977). "Fylogenetická struktura prokaryotické domény: primární království". PNAS USA. 74 (11): 5088–90. Bibcode:1977PNAS ... 74,5088W. doi:10.1073 / pnas.74.11.5088. PMC 432104. PMID 270744.
- ^ Yap, WH; Zhang, Z; Wang, Y (1999). „V genomu aktinomycety Thermomonospora chromogena existují odlišné typy operonů rRNA a důkazy o horizontálním přenosu celého operonu rRNA“. Journal of Bacteriology. 181 (17): 5201–9. doi:10.1128 / JB.181.17.5201-5209.1999. PMC 94023. PMID 10464188.
- ^ A b Simonson, AB; Servin, JA; Skophammer, RG; Herbold, CW; Rivera, MC; Lake, JA (2005). „Dekódování genomického stromu života“. Sborník Národní akademie věd Spojených států amerických. 102 Suppl 1: 6608–13. Bibcode:2005PNAS..102.6608S. doi:10.1073 / pnas.0501996102. PMC 1131872. PMID 15851667.
- ^ Woese, C. (09.06.1998). „Univerzální předek“. Sborník Národní akademie věd Spojených států amerických. 95 (12): 6854–6859. Bibcode:1998PNAS ... 95,6854W. doi:10.1073 / pnas.95.12.6854. ISSN 0027-8424. PMC 22660. PMID 9618502.
- ^ Woese, Carl R. (2002-06-25). „O vývoji buněk“. Sborník Národní akademie věd Spojených států amerických. 99 (13): 8742–8747. Bibcode:2002PNAS ... 99.8742W. doi:10.1073 / pnas.132266999. ISSN 0027-8424. PMC 124369. PMID 12077305.
- ^ Horizontální přenos genů, stát Oklahoma
- ^ Doolittle, WF (2000). "Vykořenění stromu života". Sci Am. 282 (2): 90–5. Bibcode:2000SciAm.282b..90D. doi:10.1038 / scientificamerican0200-90. PMID 10710791.
- ^ Gogarten JP „Horizontální přenos genů - nové paradigma pro biologii“ Disertační práce
- Zhaxybayeva, O; Gogarten, JP (2004). „Kladogeneze, koalescence a vývoj tří oblastí života“ (PDF). Trendy v genetice. 20 (4): 182–7. doi:10.1016 / j.tig.2004.02.004. PMID 15041172.
- ^ Ge, F .; Wang, L .; Kim, J. (2005). „Pavučina života odhalená odhady horizontálního přenosu genů v měřítku genomu“. PLOS Biology. 3 (10): e316. doi:10.1371 / journal.pbio.0030316. PMC 1233574. PMID 16122348.

- ^ Henz, S .; Huson, D .; Auch, A .; Nieselt-Struwe, K .; Schuster, S. (2005). „Prokaryotická fylogeneze celého genomu“. Bioinformatika. 21 (10): 2329–2335. doi:10.1093 / bioinformatika / bth324. PMID 15166018.
- Fitzpatrick, DA; Logue, ME; Stajich, JE; Butler, G (2006). „Fylogeneze hub založená na 42 kompletních genomech odvozených z analýzy superstromů a kombinovaných genů“. BMC Evoluční biologie. 6: 99. doi:10.1186/1471-2148-6-99. PMC 1679813. PMID 17121679.

- Fitzpatrick, DA; Logue, ME; Stajich, JE; Butler, G (2006). „Fylogeneze hub založená na 42 kompletních genomech odvozených z analýzy superstromů a kombinovaných genů“. BMC Evoluční biologie. 6: 99. doi:10.1186/1471-2148-6-99. PMC 1679813. PMID 17121679.
- ^ Urwin, R; Maiden, MC (2003). „Multi-locus sequence typing: a tool for global epidemiology“. Trends Microbiol. 11 (10): 479–87. doi:10.1016 / j.tim.2003.08.006. PMID 14557031.
- Yang, Z (2002). „Pravděpodobnost a Bayesův odhad velikosti populace předků u hominoidů pomocí dat z více lokusů“. Genetika. 162 (4): 1811–23. PMC 1462394. PMID 12524351.
- Jennings, W. B .; Edwards, S. V. (2005). "Speciační historie australských travních pěnkav (Poephila) odvozených z třiceti genových stromů". Vývoj; International Journal of Organic Evolution. 59 (9): 2033–2047. doi:10.1554/05-280.1. PMID 16261740. S2CID 18665436.
- ^ Eisen, J .; Fraser, C. (2003). "Fylogenomika: průnik evoluce a genomiky". Věda. 300 (5626): 1706–1707. Bibcode:2003Sci ... 300.1706E. doi:10.1126 / science.1086292. PMID 12805538. S2CID 42394233.
- ^ Ge, Fan; Wang, Li-San; Kim, Junhyong (2005). „Pavučina života odhalená odhady horizontálního přenosu genů v měřítku genomu“. PLOS Biology. 3 (10): e316. doi:10.1371 / journal.pbio.0030316. PMC 1233574. PMID 16122348.

- ^ Získávání genomů: teorie původu druhů. Margulis L a Sagan D (2002) Základní knihy ISBN 0-465-04392-5
- ^ Abby, Sophie S .; Tannier, Eric; Gouy, Manolo; Daubin, Vincent (2012-03-27). „Laterální přenos genů jako podpora stromu života“. Sborník Národní akademie věd. 109 (13): 4962–4967. Bibcode:2012PNAS..109,4962A. doi:10.1073 / pnas.1116871109. PMC 3323970. PMID 22416123.
- ^ Huang, Jinling; Gogarten, Johann Peter (2009). Horizontální přenos genů. Metody v molekulární biologii. 532. Humana Press. str. 127–139. doi:10.1007/978-1-60327-853-9_7. ISBN 9781603278522. PMID 19271182.
- ^ Davín, Adrián A .; Tannier, Eric; Williams, Tom A .; Boussau, Bastien; Daubin, Vincent; Szöllősi, Gergely J. (02.04.2018). „Genové transfery mohou datovat strom života“. Ekologie a evoluce přírody. 2 (5): 904–909. doi:10.1038 / s41559-018-0525-3. ISSN 2397-334X. PMC 5912509. PMID 29610471.
- ^ Szöllősi, Gergely J .; Tannier, Eric; Lartillot, Nicolas; Daubin, Vincent (01.05.2013). „Lateral Gene Transfer from the Dead“. Systematická biologie. 62 (3): 386–397. arXiv:1211.4606. doi:10.1093 / sysbio / syt003. ISSN 1063-5157. PMC 3622898. PMID 23355531.
- Tento článek včlení materiál z Citizendium článek "Fylogeneze a evoluce prokaryot ", který je licencován pod Creative Commons Attribution-ShareAlike 3.0 Unported License ale ne pod GFDL.
Další čtení
- Gogarten, JP; Townsend, JP (2005). „Horizontální přenos genů, inovace a vývoj genomu“. Recenze přírody. Mikrobiologie. 3 (9): 679–87. doi:10.1038 / nrmicro1204. PMID 16138096. S2CID 8092665.. Jeden článek v celém čísle časopisu Příroda Recenze Mikrobiologie z velké části věnovaný HGT.
- Daubin, V; Moran, NA; Ochman, H (2003). "Fylogenetika a soudržnost bakteriálních genomů". Věda. 301 (5634): 829–32. Bibcode:2003Sci ... 301..829D. doi:10.1126 / science.1086568. PMID 12907801. S2CID 11268678.
- Eisen, JA; Fraser, CM (2003). "Hledisko fylogenomika: průnik evoluce a genomiky". Věda. 300 (5626): 1706–7. Bibcode:2003Sci ... 300.1706E. doi:10.1126 / science.1086292. PMID 12805538. S2CID 42394233.
- Ge, F; Wang, LS; Kim, J (2005). „Pavučina života odhalená odhadem horizontálního přenosu genů v měřítku genomu“. PLOS Biology. 3 (10): e316. doi:10.1371 / journal.pbio.0030316. PMC 1233574. PMID 16122348.

- Henz, SR; Huson, DH; Auch, AF; Nieselt-Struwe, K; Schuster, SC (2005). „Prokaryotická fylogeneze celého genomu“. Bioinformatika. 21 (10): 2329–35. doi:10.1093 / bioinformatika / bth324. PMID 15166018.
- Lerat, E; Daubin, V; Ochman, H; Moran, NA (2005). „Evoluční počátky genomových repertoárů v bakteriích“. PLOS Biology. 3 (5): e130. doi:10.1371 / journal.pbio.0030130. PMC 1073693. PMID 15799709.

- Urwin, R; Maiden, MC (2003). „Multi-locus sequence typing: a tool for global epidemiology“. Trends Microbiol. 11 (10): 479–87. doi:10.1016 / j.tim.2003.08.006. PMID 14557031.
- Adoutte, A; Balavoine, G; Lartillot, N; Lespinet, O; Prud'homme, B; De Rosa, R (2000). „Nová fylogeneze zvířat: spolehlivost a důsledky“. Proc Natl Acad Sci USA. 97 (9): 4453–6. Bibcode:2000PNAS ... 97.4453A. doi:10.1073 / pnas.97.9.4453. PMC 34321. PMID 10781043.
- Delsuc, F; Brinkmann, H; Philippe, H (2005). „a kol.(2005) Fylogenomika a rekonstrukce stromu života “. Nat Rev Genet. 6 (5): 361–75. CiteSeerX 10.1.1.333.1615. doi:10.1038 / nrg1603. PMID 15861208. S2CID 16379422.
- Steenkamp, ET; Wright, J; Baldauf, SL (2006). „Počátky zvířat a hub v Protistanu“. Mol Biol Evol. 23 (1): 93–106. doi:10.1093 / molbev / msj011. PMID 16151185.
- Thomas, CM; Nielsen, KM. (2005). "Mechanismy a bariéry horizontálního přenosu genů mezi bakteriemi". Nat Rev Microbiol. 3 (9): 711–21. doi:10.1038 / nrmicro1234. PMID 16138099. S2CID 1231127.
