Rsa RNA - Rsa RNA
Rsa RNA jsou nekódující RNA nalezený v bakterii Zlatý stafylokok. Sdílený název pochází z jejich objevu a neznamená homologie. Bioinformatika skeny identifikovaly 16 rodin Rsa RNA pojmenovaných RsaA-K a RsaOA-OG.[1][2] Jiné, RsaOH-OX, byly nalezeny díky přístupu RNomic.[3] Ačkoli RNA vykazovaly různé vzorce exprese, ukázalo se, že mnoho z nově objevených RNA je Hfq -nezávislý a nejvíce nesený a C - bohatý motiv (UCCC).[1]
RsaA
Potlačuje translaci transkripčního regulátoru MgrA vazbou na jeho mRNA, zvyšuje tvorbu biofilmu a snižuje bakteriální virulenci.[4] Další mRNA: včetně enzymů podobných SsaA zapojených do metabolismu peptidoglykanů a vylučovaného protizánětlivého proteinu FLIPr byly ověřeny jako přímé cíle RsaA.[5]
RsaE
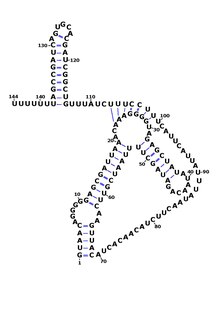
RsaE se vyskytuje u ostatních členů rodu Staphylococcus jako Staphylococcus epidermidis a Staphylococcus saprophyticus a je jedinou Rsa RNA, která se nachází mimo tento rod, v Macrococcus caseolyticus a Bacil. v Bacillus subtilis, RsaE byl dříve identifikován jako ncr22.[8][9] RsaE se také důsledně nalézá po proudu od PepF pro které kódy oligoendopeptidáza F. Funkce RsaE byla objevena pomocí genový knockout analýza a nadměrná exprese genů - bylo zjištěno, že regulují výraz několika enzymů zapojených do metabolismus přes antisense vazba jejich mRNA.[1][3]
Ukázalo se, že RsaE je regulován přítomností oxidu dusnatého (NO). v Bacillus subtilis řídí expresi genů s funkcemi souvisejícími s oxidačním stresem a oxidačně-redukčními reakcemi a byl přejmenován na RoxS (pro související s oxidačním stresem).[10]
RsaF
v S.aureus druh RsaF se nachází ve stejné intergenní oblasti jako RsaE a překrývá se s 3 'koncem RsaE přibližně o 20 bp. Na rozdíl od RsaE byl RsaF a jeho upstream gen identifikován pouze v Druh S.aureus.[1]
RsaK
RsaK se nachází v vedoucí sekvence z glcA mRNA, která kóduje enzym podílí se na specifické pro glukózu fosfotransferázový systém. RsaK také obsahuje konzervované ribonukleové antiterminátor systém, jak je rozpoznán proteinem GclT.[11]
RsaI
RsaOG[2] také přejmenován na RsaI[1] Předpokládá se, že vyladí regulaci toxinů nebo invazních mechanismů S. aureus přes trans-působící mechanismy. Své sekundární struktura obsahuje a pseudoknot vytvořené mezi dvěma vysoce konzervovanými nepárovými sekvencemi.[6]
Vzory výrazů
Bylo zjištěno, že RsaD, EH a I jsou vysoce exprimovány v S. aureus. Hladiny exprese dalších Rsa RNA se měnily za různých podmínek prostředí, například RsaC byl indukován chladný šok a RsaA je indukován v reakci na osmotický stres.[1][2][3]
Geny RsaE a RsaF se překrývají S.aureus ale zdá se, že mají opačné vzorce exprese.[1] Transkripční interference kvůli překrytí mezi a σA rozpoznávací motiv a potenciál σB vazebné místo je navrženo jako mechanismus způsobující diferenciální expresi dvou transkriptů[1][12]
Viz také
Reference
- ^ A b C d E F G h Geissmann T, Chevalier C, Cros MJ, Boisset S, Fechter P, Noirot C, Schrenzel J, François P, Vandenesch F, Gaspin C, Romby P (listopad 2009). „Hledání malých nekódujících RNA ve Staphylococcus aureus odhaluje konzervovaný sekvenční motiv pro regulaci“. Výzkum nukleových kyselin. 37 (21): 7239–7257. doi:10.1093 / nar / gkp668. PMC 2790875. PMID 19786493.
- ^ A b C Marchais A, Naville M, Bohn C, Bouloc P, Gautheret D (červen 2009). „Jednoprůchodová klasifikace všech nekódujících sekvencí v bakteriálním genomu pomocí fylogenetických profilů“. Výzkum genomu. 19 (6): 1084–1092. doi:10.1101 / gr.089714.108. PMC 2694484. PMID 19237465.
- ^ A b C Bohn C, Rigoulay C, Chabelskaya S, Sharma CM, Marchais A, Skorski P, Borezée-Durant E, Barbet R, Jacquet E, Jacq A, Gautheret D, Felden B, Vogel J, Bouloc P (říjen 2010). „Experimentální objev malých RNA ve Staphylococcus aureus odhaluje riboregulátor centrálního metabolismu“. Výzkum nukleových kyselin. 38 (19): 6620–6636. doi:10.1093 / nar / gkq462. PMC 2965222. PMID 20511587.
- ^ Romilly C, Lays C, Tomasini A, Caldelari I, Benito Y, Hammann P, Geissmann T, Boisset S, Romby P, Vandenesch F (březen 2014). „Nekódující RNA podporuje bakteriální perzistenci a snižuje virulenci regulací regulátoru u Staphylococcus aureus“. PLoS patogeny. 10 (3): e1003979. doi:10.1371 / journal.ppat.1003979. PMC 3961350. PMID 24651379.
- ^ Tomasini A, Moreau K, Chicher J, Geissmann T, Vandenesch F, Romby P, Marzi S, Caldelari I (červen 2017). „Cílový RNA staphylococcus aureus nekódující RNA RsaA: dopad na vlastnosti buněčného povrchu a obranné mechanismy“. Výzkum nukleových kyselin. 45 (11): 6746–6760. doi:10.1093 / nar / gkx219. PMC 5499838. PMID 28379505.
- ^ A b Marchais A, Bohn C, Bouloc P, Gautheret D (březen 2010). „RsaOG, nová stafylokoková rodina vysoce transkribované nekódující RNA“. RNA Biology. 7 (2): 116–119. doi:10,4161 / rna.7.2.10925. PMID 20200491.
- ^ Darty K, Denise A, Ponty Y (srpen 2009). "VARNA: Interaktivní kresba a úpravy sekundární struktury RNA". Bioinformatika. 25 (15): 1974–1975. doi:10.1093 / bioinformatika / btp250. PMC 2712331. PMID 19398448.
- ^ Rasmussen S, Nielsen HB, Jarmer H (září 2009). „Transkripčně aktivní oblasti v genomu Bacillus subtilis“. Molekulární mikrobiologie. 73 (6): 1043–1057. doi:10.1111 / j.1365-2958.2009.06830.x. PMC 2784878. PMID 19682248.
- ^ Irnov I, Sharma CM, Vogel J, Winkler WC (říjen 2010). "Identifikace regulačních RNA v Bacillus subtilis". Výzkum nukleových kyselin. 38 (19): 6637–6651. doi:10.1093 / nar / gkq454. PMC 2965217. PMID 20525796.
- ^ Durand S, Braun F, Lioliou E, Romilly C, Helfer AC, Kuhn L, Quittot N, Nicolas P, Romby P, Condon C (únor 2015). „Malá RNA regulovaná oxidem dusnatým řídí expresi genů zapojených do redoxní homeostázy u Bacillus subtilis“. Genetika PLoS. 11 (2): e1004957. doi:10.1371 / journal.pgen.1004957. PMC 4409812. PMID 25643072.
- ^ Langbein I, Bachem S, Stülke J (listopad 1999). „Specifická interakce domény vázající RNA transkripčního antiterminátoru GlcT bacillus subtilis s jeho RNA cílem, RAT“. Journal of Molecular Biology. 293 (4): 795–805. doi:10.1006 / jmbi.1999.3176. PMID 10543968.
- ^ Shearwin KE, Callen BP, Egan JB (červen 2005). „Transkripční interference - rychlokurz“. Trendy v genetice. 21 (6): 339–345. doi:10.1016 / j.tig.2005.04.009. PMC 2941638. PMID 15922833.
Další čtení
- Pichon C, Felden B (říjen 2005). „Malé geny RNA exprimované z genomových a patogenních ostrovů Staphylococcus aureus se specifickou expresí mezi patogenními kmeny“. Sborník Národní akademie věd Spojených států amerických. 102 (40): 14249–14254. doi:10.1073 / pnas.0503838102. PMC 1242290. PMID 16183745.
- Altuvia S (červen 2007). "Identifikace bakteriálních malých nekódujících RNA: experimentální přístupy". Současný názor v mikrobiologii. 10 (3): 257–261. doi:10.1016 / j.mib.2007.05.003. PMID 17553733.
- Morfeldt E, Taylor D, von Gabain A, Arvidson S (září 1995). "Aktivace translace alfa-toxinu u Staphylococcus aureus transkódovanou antisense RNA, RNAIII". Časopis EMBO. 14 (18): 4569–4577. doi:10.1002 / j.1460-2075.1995.tb00136.x. PMC 394549. PMID 7556100.
- Roberts C, Anderson KL, Murphy E, Projan SJ, Mounts W, Hurlburt B, Smeltzer M, Overbeek R, Disz T, Dunman PM (duben 2006). „Charakterizace účinku regulátoru faktoru virulence Staphylococcus aureus, SarA, na biologické poločasy mRNA“. Journal of Bacteriology. 188 (7): 2593–2603. doi:10.1128 / JB.188.7.2593-2603.2006. PMC 1428411. PMID 16547047.
- Anderson KL, Roberts C, Disz T, Vonstein V, Hwang K, Overbeek R, Olson PD, Projan SJ, Dunman PM (říjen 2006). „Charakterizace tepelného šoku ze Staphylococcus aureus, studeného šoku, přísných a SOS reakcí a jejich účinků na logaritmický obrat mRNA“. Journal of Bacteriology. 188 (19): 6739–6756. doi:10.1128 / JB.00609-06. PMC 1595530. PMID 16980476.
- Huntzinger E, Boisset S, Saveanu C, Benito Y, Geissmann T, Namane A, Lina G, Etienne J, Ehresmann B, Ehresmann C, Jacquier A, Vandenesch F, Romby P (únor 2005). „Staphylococcus aureus RNAIII a endoribonukleáza III koordinují expresi lázeňských genů“. Časopis EMBO. 24 (4): 824–835. doi:10.1038 / sj.emboj.7600572. PMC 549626. PMID 15678100.
- Novick RP, Ross HF, Projan SJ, Kornblum J, Kreiswirth B, Moghazeh S (říjen 1993). „Syntéza faktorů stafylokokové virulence je řízena regulační molekulou RNA“. Časopis EMBO. 12 (10): 3967–3975. doi:10.1002 / j.1460-2075.1993.tb06074.x. PMC 413679. PMID 7691599.
- Bohn C, Rigoulay C, Bouloc P (únor 2007). "Žádný detekovatelný účinek nepřítomnosti Hfq proteinu vázajícího RNA na Staphylococcus aureus". Mikrobiologie BMC. 7: 10. doi:10.1186/1471-2180-7-10. PMC 1800855. PMID 17291347.