Nekanonické párování základen - Non-canonical base pairing
Nekanonické párování základen nastane, když nukleové báze vodíková vazba nebo základní pár k sobě navzájem v jiných schématech, než jsou standardní páry bází Watson-Crick (které jsou adenin (A) - tymin (T) v DNA, adenin (A) - uracil (U) v RNA, a guanin (G) - cytosin (C) v DNA i RNA). Existují tři hlavní typy nekanonických párů bází: ty stabilizované polárním Vodíkové vazby, ti, kteří mají interakce mezi C-H a O / N skupinami, a ti, kteří mají vodíkové vazby mezi samotnými bázemi.[1] První objevené nekanonické páry bází jsou Hoogsteenovy páry bází, které poprvé popsal americký biochemik Kras Hoogsteen.
Nekanonické párování bází se běžně vyskytuje v sekundární struktura RNA (např. párování G s U) a in tRNA uznání. Obvykle jsou méně stabilní než standardní párování bází.[2] Přítomnost nekanonických párů bází ve dvouvláknové DNA má za následek narušení dvojitá spirála.[3]
Dějiny
James Watson a Francis Crick zveřejnil dvojitá šroubovice struktura DNA a navrhl kanonické páry bází Watson-Crick v roce 1953.[4] O deset let později, v roce 1963, Kras Hoogsteen hlásil, že použil monokrystalová rentgenová difrakce prozkoumat alternativní struktury párů bází a našel alternativní strukturu pro pár nucelobáz adenin-thymin, ve kterém purin (A) přijímá alternativní konformaci s ohledem na pyrimidin (T).[5] Pět let poté, co Hoogsteen navrhl A-T Hoogsteenův základní pár, optická rotační disperze Byly hlášeny spektra, která poskytla důkazy o páru bází G-C Hoogsteen.[6] Pár bází G-C Hoogsteen byl poprvé pozorován rentgenovou krystalografií o několik let později, v roce 1986, kokrystalizací DNA s triostinem A ( antibiotikum ).[7] Nakonec, po letech studia párů bází Watson-Crick a Hoogsteen, bylo zjištěno, že oba se přirozeně vyskytují v DNA a že existují ve vzájemné rovnováze; podmínky, ve kterých DNA existuje, nakonec určují, která forma bude upřednostňována.[8] Protože byly určeny struktury kanonických párů bází Watson-Crick a nekanonických Hoogsteenů, bylo představeno a popsáno mnoho dalších typů nekanonických párů bází.
Struktura
Základní párování

Odhaduje se, že 60% bází ve strukturované RNA se účastní kanonických párů bází Watson-Crick.[9] Párování bází nastává, když dvě báze tvoří vodíkové vazby. Tyto vodíkové vazby mohou být buď polární, nebo nepolární interakce. Polární vodíkové vazby jsou tvořeny interakcemi N-H ... O / N a / nebo O-H ... O / N. Mezi C-H ... O / N se tvoří nepolární vodíkové vazby.[10]
Interakce hran
Každá základna má tři potenciální hrany, kde může interagovat s jinou základnou. Purinové báze mají 3 hrany, které jsou schopné vodíkové vazby. Ty jsou známé jako hrana Watson-Crick (WC), hrana Hoogsteen (H) a hrana Sugar (S). Pyrimidinové báze mají také tři okraje vázající vodík.[9] Stejně jako pyrimidin existuje hrana Watson-Crick (WC) a hrana cukru (S), ale třetí hrana se označuje jako hrana „C-H“ (H). Tato hrana C-H se pro jednoduchost někdy označuje také jako hrana Hoogsteen. Na obrázku 2 jsou znázorněny různé hrany pro purinové a pyrimidinové báze.[10]

Kromě tří hran interakce se páry bází také liší v různých formách cis / trans. Struktury cis a trans závisí na orientaci ribózového cukru ve srovnání s interakcí vodíkové vazby. Tyto různé orientace jsou znázorněny na obrázku 3. S formami cis / trans a 3 hranami vodíkových vazeb existuje 12 základních typů geometrií párování bází, které lze nalézt ve strukturách RNA. Těchto 12 typů je WC: WC (cis / trans), W: HC (cis / trans), WC: S (cis / trans), H: S (cis / trans), H: H (cis / trans) a S: S (cis / trans).
Klasifikace
Těchto 12 typů lze dále rozdělit do více podskupin, které jsou závislé na směrovosti glykosidických vazeb a stérických prodloužení.[11] Se všemi různými kombinacemi základních párů existuje 169 teoreticky možných kombinací základních párů. Počet skutečných kombinací párů bází je však mnohem nižší, protože některé kombinace vedou k nepříznivým interakcím. Tento počet možných nekanonických párů bází se stále určuje, protože je velmi závislý na kritériích párování bází.[12] Pochopení konfigurace párů bází je obtížné, protože párování je velmi závislé na okolí základen. Tato prostředí se skládají ze sousedních párů bází, sousedních smyček nebo třetí interakce, jako je trojnásobek báze.[13]

Protože různé základny jsou tuhé a rovinné, vazby mezi nimi jsou dobře definované. Prostorové interakce mezi dvěma základnami lze klasifikovat do 6 parametrů tuhého těla nebo parametrů párů v rámci báze (3 translační, 3 rotační), jak je znázorněno na obrázku 4.[14] Tyto parametry popisují základní páry trojrozměrné potvrzení. Tři translační uspořádání jsou známá jako smykové, roztažné a střídavé. Tyto tři parametry přímo souvisejí s blízkostí a směrem dvojic vodíkových vazeb. Rotační uspořádání je spona, vrtule a otevírání. Rotační uspořádání souvisí s nerovinným potvrzením ve srovnání s ideální koplanární geometrií.[10] Parametry intra-base pair se používají ke stanovení struktury a stabilit nekanonických párů bází. Tyto parametry byly původně vytvořeny pro párování bází v DNA, ale mohou také odpovídat nekanonickým základním modelům.[14]
Typy
Nejběžnější nekanonické páry bází jsou trans A: G Hoogsteen / hrana cukru, A: U Hoogsteen / WC a G: U Kolísavé páry.[15]
Hoogsteenovy páry bází

Hoogsteenovy páry bází vyskytují se mezi adeninem (A) a tyminem (T) a guaninem (G) a cytosinem (C), podobně jako páry bází Watson-Crick; purin však přijímá alternativní konformaci vzhledem k pyrimidinu. V páru bází A-U Hoogsteen je adenin otočen o 180 ° kolem glykosidová vazba, což má za následek alternativní schéma vodíkových vazeb, které má jednu vodíkovou vazbu společnou s párem bází Watson-Crick (adenin N6 a thymin N4), zatímco druhý, místo toho, aby se vyskytoval mezi adeninem N1 a thyminem N3 jako v bázi Watson-Crick pár se vyskytuje mezi adeninem N7 a tyminem N3.[8] Pár bází A-U je znázorněn na obrázku 5. V páru bází G-C Watson-Crick, podobně jako pár bází A-T Hoogsteen, se purin (guanin) otáčí o 180 ° kolem glykosidové vazby, zatímco pyrimidin (cytosin) zůstává na svém místě. Jedna vodíková vazba z páru bází Watson-Crick je udržována (guanin O6 a cytosin N4) a druhá se vyskytuje mezi guaninem N7 a protonovaným cytosinem N3 (všimněte si, že pár bází Hoogsteen GC má dvě vodíkové vazby, zatímco Watson-Crick GC základní pár má tři).[8]

Kolísání párů bází
Kolísání párování základen se vyskytují mezi dvěma nukleotidy, které nejsou Watson-Crickovými páry bází. 4 hlavními příklady jsou guanin-uracil (G-U), hypoxanthin-uracil (I-U), hypoxanthin-adenin (I-A) a hypoxanthin-cytosin (I-C). Tyto kolísavé páry bází jsou v tRNA velmi důležité. Většina organismů má méně než 45 molekul tRNA, ale ke kanonickému spárování s kodonem by bylo zapotřebí 61 molekul tRNA. Wobble párování bází bylo navrženo Watsonem v roce 1966. Párování Wobble bází umožňuje 5 'antikodon na nestandardní pár bází. Příklady vratkých párů bází jsou uvedeny na obrázku 6.
3-D struktura
Sekundární a trojrozměrné struktury RNA se tvoří a stabilizuje prostřednictvím nekanonických párů bází. Páry bází tvoří mnoho sekundárních strukturálních bloků, které napomáhají skládání komplexů RNA a trojrozměrných struktur. Celková složená RNA je stabilizována terciárními a sekundárními strukturami kanonicky spárovanými bázemi dohromady.[10] Vzhledem k mnoha nekanonickým párům bází existuje neomezené množství struktur, které umožňují rozmanité funkce RNA.[9] Uspořádání nekanonických bází umožňuje interakce RNA na velké vzdálenosti, rozpoznávání proteinů a dalších molekul a strukturní stabilizační prvky.[14] Mnoho běžných nekanonických párů bází lze přidat do skládaného kmene RNA, aniž by došlo k narušení jeho spirálového charakteru.[1]
Sekundární

Mezi základní sekundární strukturní prvky RNA patří boule, dvojité šroubovice, vlásenky a vnitřní smyčky. Příklad vlásenkové smyčky RNA je uveden na obrázku 7. Jak je znázorněno na obrázku, vlásenkové smyčky a vnitřní smyčky vyžadují náhlou změnu směru páteře. Nekanonické párování základen umožňuje zvýšenou flexibilitu na křižovatkách nebo zatáčkách v sekundární struktuře.[10]
Terciární
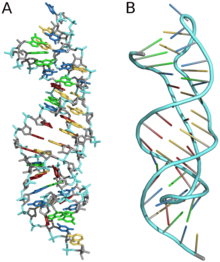
Trojrozměrné struktury se vytvářejí prostřednictvím mezimolekulárních interakcí mezi sekundárními strukturami na velké vzdálenosti. To vede k tvorbě pseudoknotů, ribózových zipů, líbání vlásenkových smyček nebo koaxiálních pseudokontinuálních šroubovic.[10] Trojrozměrné struktury RNA jsou primárně určovány pomocí molekulárních simulací nebo výpočtově řízených měření.[14] Příklad Pseudoknotu je uveden na obrázku 8.
Experimentální metody
Watson-Crickovo kanonické párování bází není jedinou možnou konformací od okraje k okraji pro nukleotid, protože může dojít také k nekanonickému párování. Cukr-fosfátová páteř má iontový charakter, díky čemuž jsou báze citlivé na jejich prostředí, což vede ke konformačním změnám, jako je nekanonické párování.[16][1] Pro tyto konformace existují různé metody predikce, jako např NMR stanovení struktury a Rentgenová krystalografie.[16]
Aplikace
RNA má v celé buňce mnoho účelů, včetně mnoha důležitých kroků genová exprese. Různé konformace párů bází jiných než Watson-Crick umožňují mnoho biologických funkcí, jako je mRNA sestřih, siRNA, transport, rozpoznávání proteinů, vazba proteinů a překlad.[17][18]
Jedním z příkladů biologické aplikace nekanonických párů bází v zatáčce. Zlom se nachází u mnoha funkčních druhů RNA. Skládá se ze tří-nukleotidové podpalubí, které je způsobeno třemi páry bází Hoogsteen. Toto zalomení funguje jako marker, kde se mohou vázat různé proteiny, jako je lidský protein 15-5k nebo proteiny v rodině L7Ae.[19] Podobný scénář je popsán ve vazbě na RNA HIV-1 Rev-response element (RRE). RNA má extra širokou hlubokou rýhu, která je způsobena cis Watson-Crick G: Pár následovaný trans Watson-Crick G: G. Prvek reakce HIV-1 Rev je poté schopen vázat se díky prohloubené drážce.[1]
Viz také
Reference
- ^ A b C d Hermann T, Westhof E (prosinec 1999). „Páry bází jiné než Watson-Crick v rozpoznávání RNA-proteinů“ (PDF). Chemie a biologie. 6 (12): R335-43. doi:10.1016 / s1074-5521 (00) 80003-4. PMID 10631510.
- ^ Lemieux S, major F (říjen 2002). „RNA kanonické a nekanonické typy párování bází: metoda rozpoznávání a úplný repertoár“. Výzkum nukleových kyselin. 30 (19): 4250–63. doi:10.1093 / nar / gkf540. PMC 140540. PMID 12364604.
- ^ Das J, Mukherjee S, Mitra A, Bhattacharyya D (říjen 2006). "Nekanonické páry bází a struktury vyššího řádu v nukleových kyselinách: analýza databáze krystalové struktury". Journal of Biomolecular Structure & Dynamics. 24 (2): 149–61. doi:10.1080/07391102.2006.10507108. PMID 16928138.
- ^ Watson JD, Crick FH (duben 1953). "Molekulární struktura nukleových kyselin; struktura pro deoxyribosovou nukleovou kyselinu". Příroda. 171 (4356): 737–8. Bibcode:1953Natur.171..737W. doi:10.1038 / 171737a0. PMID 13054692.
- ^ Hoogsteen K (10.10.1963). „Krystalová a molekulární struktura komplexu vázaného na vodík mezi 1-methylthyminem a 9-methyladeninem“. Acta Crystallographica. 16 (9): 907–916. doi:10.1107 / S0365110X63002437. ISSN 0365-110X.
- ^ Courtois Y, Fromageot P, Guschlbauer W (prosinec 1968). „Protonované polynukleotidové struktury. 3. Studie optické rotační disperze protonace DNA“. European Journal of Biochemistry. 6 (4): 493–501. doi:10.1111 / j.1432-1033.1968.tb00472.x. PMID 5701966.
- ^ Quigley GJ, Ughetto G, van der Marel GA, van Boom JH, Wang AH, Rich A (červen 1986). „Non-Watson-Crick G.C a A.T párů bází v komplexu DNA-antibiotikum“. Věda. 232 (4755): 1255–8. doi:10.1126 / science.3704650. PMID 3704650.
- ^ A b C Nikolova EN, Zhou H, Gottardo FL, Alvey HS, Kimsey IJ, Al-Hashimi HM (prosinec 2013). „Historický popis párů bází Hoogsteen v duplexní DNA“. Biopolymery. 99 (12): 955–68. doi:10,1002 / bip.22334. PMC 3844552. PMID 23818176.
- ^ A b C Leontis NB, Westhof E (duben 2001). "Geometrická nomenklatura a klasifikace párů bází RNA". RNA. 7 (4): 499–512. doi:10.1017 / S1355838201002515. PMC 1370104. PMID 11345429.
- ^ A b C d E F G Halder S, Bhattacharyya D (listopad 2013). "Struktura a dynamika RNA: perspektiva párování bází". Pokrok v biofyzice a molekulární biologii. 113 (2): 264–83. doi:10.1016 / j.pbiomolbio.2013.07.003. PMID 23891726.
- ^ Sponer JE, Leszczynski J, Sychrovský V, Sponer J (říjen 2005). "Cukrová hrana / párová hrana cukrové hrany v RNA: stability a struktury z kvantově chemických výpočtů". The Journal of Physical Chemistry B. 109 (39): 18680–9. doi:10.1021 / jp053379q. PMID 16853403.
- ^ Sharma P, Sponer JE, Sponer J, Sharma S, Bhattacharyya D, Mitra A (březen 2010). „O úloze cis Hoogsteen: rodina párů bází na hraně cukru a triplety - kvantový chemický pohled na strukturní biologii RNA“. The Journal of Physical Chemistry B. 114 (9): 3307–20. doi:10.1021 / jp910226e. PMID 20163171.
- ^ Heus HA, Hilbers CW (říjen 2003). „Struktury nekanonických párů tandemových bází v helixech RNA: recenze“. Nukleosidy, nukleotidy a nukleové kyseliny. 22 (5–8): 559–71. doi:10.1081 / NCN-120021955. PMID 14565230.
- ^ A b C d Olson WK, Li S, Kaukonen T, Colasanti AV, Xin Y, Lu XJ (květen 2019). „Účinky párování nekanonických bází na skládání RNA: strukturální kontext a prostorová uspořádání párů G · A“. Biochemie. 58 (20): 2474–2487. doi:10.1021 / acs.biochem.9b00122. PMC 6729125. PMID 31008589.
- ^ Roy A, Panigrahi S, Bhattacharyya M, Bhattacharyya D (březen 2008). "Struktura, stabilita a dynamika kanonických a nekanonických párů bází: kvantové chemické studie". The Journal of Physical Chemistry B. 112 (12): 3786–96. doi:10.1021 / jp076921e. PMID 18318519.
- ^ A b Lu XJ, Olson WK (září 2003). „3DNA: softwarový balíček pro analýzu, přestavbu a vizualizaci trojrozměrných struktur nukleových kyselin“. Výzkum nukleových kyselin. 31 (17): 5108–21. doi:10.1093 / nar / gkg680. PMC 212791. PMID 12930962.
- ^ Fernandes CL, Escouto GB, Verli H (2013-06-28). "Strukturální glykobiologie heparinázy II z Pedobacter heparinus". Journal of Biomolecular Structure & Dynamics. 32 (7): 1092–102. doi:10.1080/07391102.2013.809604. PMID 23808670.
- ^ Storz G, Altuvia S, Wassarman KM (01.06.2005). "Hojnost regulátorů RNA". Roční přehled biochemie. 74 (1): 199–217. doi:10,1146 / annurev.biochem.74.082803.133136. PMID 15952886.
- ^ Huang L, Lilley DM (leden 2018). „Zlom ve strukturální biologii RNA“. Čtvrtletní recenze biofyziky. 51: e5. doi:10.1017 / S0033583518000033. PMID 30912490.
