Gaussův síťový model - Gaussian network model

The Gaussův síťový model (GNM) je znázorněním biologického makromolekula jako elastická hmotajaro síť pro studium, porozumění a charakterizaci mechanických aspektů jejího dlouhodobého velkého rozsahu dynamika. Tento model má širokou škálu aplikací od malých proteinů, jako jsou enzymy složené z jednoho doména, na velké makromolekulární sestavy jako a ribozom nebo virový kapsid. Dynamika proteinové domény hraje klíčovou roli v mnoha molekulárních rozpoznávání a buněčná signalizace procesy. Proteinové domény spojené s vnitřní poruchou flexibilní linker domén, vyvolat dalekonosný allostery přes dynamika proteinové domény Výsledné dynamické režimy nelze obecně předpovědět ze statických struktur celého proteinu nebo jednotlivých domén.
Gaussův síťový model je minimalistický, hrubozrnný přístup ke studiu biologických molekul. V modelu jsou proteiny reprezentovány uzly odpovídajícími a-uhlíkům aminokyselinových zbytků. Podobně jsou struktury DNA a RNA zastoupeny jedním až třemi uzly pro každý z nich nukleotid. Model používá harmonickou aproximaci k modelování interakcí. Díky této hrubozrnné reprezentaci jsou výpočty nenákladné.
Na molekulární úrovni je mnoho biologických jevů, jako je katalytická aktivita an enzym, se vyskytují v rozsahu nano- až milisekundových časových řad. Všechny techniky simulace atomů, jako např molekulární dynamika simulace zřídka dosáhnou délky trajektorie mikrosekund, v závislosti na velikosti systému a dostupných výpočetních zdrojích. Analýza normálního režimu v kontextu GNM nebo obecně modelů elastické sítě (EN) poskytuje poznatky o funkčním dynamickém chování makromolekul v delším měřítku. Model zde zachycuje funkční pohyby biomolekuly v nativním stavu za cenu atomových detailů. Závěr získaný z tohoto modelu je komplementární k technikám simulace atomových detailů.
Dalším modelem dynamiky proteinů založeným na elastických sítích hmoty a pružiny je Anizotropní model sítě.
Teorie modelu Gaussovy sítě
Gaussův síťový model navrhli Bahar, Atilgan, Haliloglu a Erman v roce 1997.[1][2] GNM se často analyzuje pomocí analýzy v normálním režimu, která nabízí analytickou formulaci a jedinečné řešení pro každou strukturu. Analýza GNM v normálním režimu se liší od ostatních analýz v normálním režimu v tom, že je založena výhradně na topologii kontaktů mezi zbytky, ovlivněné teorií pružnosti Floryho [3] a Rouse model [4] a nebere v úvahu trojrozměrnou směrovost pohybů.
Reprezentace struktury jako elastické sítě
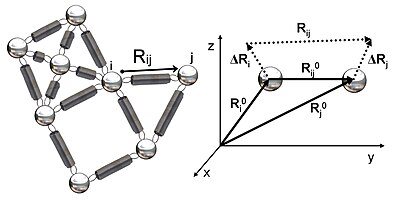
Obrázek 2 ukazuje schematický pohled na elastickou síť studovanou v GNM. Kovové kuličky představují uzly v této gaussovské síti (zbytky proteinu) a pružiny představují spojení mezi uzly (kovalentní a nekovalentní interakce mezi zbytky). Pro uzly i a jvektory rovnovážné polohy, R0i a R0jvektor rovnovážné vzdálenosti, R0ij, vektory okamžitých fluktuací, ΔRi a ΔRja vektor okamžité vzdálenosti, Rij, jsou zobrazeny na obrázku 2. Vektory okamžité polohy těchto uzlů jsou definovány pomocí Ri a Rj. Rozdíl mezi vektorem rovnovážné polohy a vektorem okamžité polohy zbytku i dává okamžitý fluktuační vektor, ΔRi = Ri - R0i. Okamžitý fluktuační vektor mezi uzly i a j je vyjádřena jako ΔRij = ΔRj - ΔRi = Rij - R0ij.
Potenciál gaussovské sítě
Potenciální energie sítě z hlediska ΔRi je
kde y je silová konstantní uniforma pro všechny pružiny a Γij je ijtého prvku Kirchhoff (nebo konektivita) matice kontaktů mezi zbytky, Γ, definován
rC je mezní vzdálenost pro prostorové interakce a je považována za 7 Å pro páry aminokyselin (představované jejich α-uhlíky).
Vyjádření složek X, Y a Z fluktuačních vektorů ΔRi tak jako ΔXT = [ΔX1 ΔX2 ..... ΔXN], ΔYT = [ΔY1 ΔY2 ..... ΔYN], a ΔZT = [ΔZ1 ΔZ2 ..... ΔZN], nad rovnicí se zjednodušuje na
Základy statistické mechaniky
V GNM je rozdělení pravděpodobnosti všech fluktuací, P(ΔR) je izotropní
a Gaussian
kde kB je Boltzmannova konstanta a T je absolutní teplota. str(ΔY) a str(ΔZ) jsou vyjádřeny podobně. Funkce N-rozměrné Gaussovy hustoty pravděpodobnosti s vektorem náhodných proměnných X, střední vektor μ a kovarianční matice Σ je
normalizuje distribuci a | Σ | je determinant kovarianční matice.
Podobně jako Gaussovo rozdělení, normalizované rozdělení pro ΔXT = [ΔX1 ΔX2 ..... ΔXN] kolem rovnovážných poloh lze vyjádřit jako
Normalizační konstanta, také funkce oddílu ZX, darováno
kde je v tomto případě kovarianční matice. ZY a ZZ jsou vyjádřeny podobně. Tato formulace vyžaduje inverzi Kirchhoffovy matice. V GNM je determinant Kirchhoffovy matice nulový, a proto je vyžadován výpočet její inverzní funkce rozklad vlastních čísel. Γ−1 je konstruován pomocí N-1 nenulových vlastních čísel a přidružených vlastních čísel. Výrazy pro str(ΔY) a str(ΔZ) jsou podobné jako u str(ΔX). Stává se pravděpodobné rozdělení všech fluktuací v GNM
U tohoto systému hmotnosti a pružiny je normalizační konstanta v předchozím výrazu celková funkce oddílu GNM, ZGNM,
Očekávané hodnoty fluktuací a korelací
Očekávané hodnoty kolísání reziduí, <ΔRi2> (nazývané také fluktuace střední mocniny, MSFs) a jejich vzájemné korelace, <ΔRi · ΔRj> lze uspořádat jako diagonální a off-diagonální členy kovarianční matice. Na základě statistické mechaniky je kovarianční matice pro ΔX darováno
Poslední rovnost se získá vložením výše uvedeného p (ΔX) a vezmeme (zobecněný Gaussův) integrál. Od té doby,
<ΔRi2> a <ΔRi · ΔRj> následuje
Dekompozice režimu
Normální režimy GNM se nacházejí diagonalizací Kirchhoffovy matice, Γ = UΛUT. Tady, U je unitární matice, UT = U−1vlastních vektorů ui z Γ a Λ je diagonální matice vlastních čísel λi. Frekvence a tvar režimu je reprezentován jeho vlastní hodnotou a vlastním vektorem. Vzhledem k tomu, že Kirchhoffova matice je kladná semitečná, první vlastní hodnota, λ1, je nula a odpovídající vlastní vektor má všechny jeho prvky rovné 1 /√N. To ukazuje, že síťový model je translačně invariantní.
Křížové korelace mezi fluktuacemi reziduí lze zapsat jako součet za nenulové režimy N-1 jako
Z toho vyplývá, že, [ΔRi · ΔRj], příspěvek jednotlivého režimu je vyjádřen jako
kde [uk]i je ith prvek uk.
Vliv místní hustoty balení
Podle definice diagonální prvek Kirchhoffovy matice, Γii, se rovná stupni uzlu v GNM, který představuje koordinační číslo příslušného zbytku. Toto číslo je měřítkem lokální hustoty balení kolem daného zbytku. Vliv lokální hustoty náplně lze posoudit rozšířením série o Γ−1 matice. Γ lze zapsat jako součet dvou matic, Γ = D + Óobsahující diagonální prvky a mimo diagonální prvky Γ.
- Γ−1 = (D + Ó)−1 = [ D (Já + D−1Ó) ]−1 = (Já + D−1Ó)−1D−1 = (Já - D−1Ó + ...)−1D−1 = D−1 - D−1Ó D−1 + ...
Tento výraz ukazuje, že místní hustota balení významně přispívá k očekávaným výkyvům reziduí.[5] Termíny, které následují po inverzi diagonální matice, jsou příspěvky polohových korelací k očekávaným výkyvům.
GNM aplikace

Kolísání rovnováhy
Rovnovážné fluktuace biologických molekul lze experimentálně měřit. v Rentgenová krystalografie B-faktor (také nazývaný Debye-Wallerův nebo teplotní faktor) každého atomu je měřítkem jeho střední kvadratické fluktuace poblíž jeho rovnovážné polohy v nativní struktuře. V NMR experimentech lze toto měřítko získat výpočtem rozdílů mezi střední a druhou mocninou mezi různými modely. V mnoha aplikacích a publikacích, včetně původních článků, se ukázalo, že očekávané fluktuace reziduí získané pomocí GNM jsou v dobré shodě s experimentálně měřené fluktuace nativního stavu.[6][7] Například vztah mezi faktory B a očekávanými fluktuacemi reziduí získanými z GNM je následující
Obrázek 3 ukazuje příklad výpočtu GNM pro katalytickou doménu proteinu Cdc25B, a cyklus buněčného dělení fosfatáza s dvojí specificitou.

Fyzické významy pomalého a rychlého režimu
Diagonalizace Kirchhoffovy matice rozkládá konformační pohyby na spektrum kolektivních režimů. Očekávané hodnoty fluktuací a vzájemných korelací jsou získány z lineárních kombinací fluktuací podél těchto normálních režimů. Příspěvek každého režimu je upraven s inverzní frekvencí těchto režimů. Pomalé (nízkofrekvenční) režimy tedy nejvíce přispívají k očekávaným výkyvům. Spolu s několika nejpomalejšími režimy se pohyby ukázaly jako kolektivní a globální a potenciálně relevantní pro funkčnost biomolekul. Rychlé (vysokofrekvenční) režimy na druhé straně popisují nekorelované pohyby, které neindukují významné změny ve struktuře. Metody založené na GNM neposkytují skutečnou dynamiku, ale pouze aproximaci založenou na kombinaci a interpolaci normálních režimů.[8] Jejich použitelnost silně závisí na tom, jak kolektivní je pohyb.[8][9]
Další specifické aplikace
Existuje několik hlavních oblastí, ve kterých se Gaussův síťový model a další elastické síťové modely ukázaly jako užitečné.[10] Tyto zahrnují:
- Síťový model založený na jarních perličkách: V síťovém modelu založeném na pružinových kuličkách se pružiny a kuličky používají jako komponenty v zesítěné síti. Pružiny jsou zesítěny, aby představovaly mechanické chování modelu materiálu a mostu molekulární dynamiky (MD) a modelu konečných prvků (FE) (viz obrázek 5). Korálky představují hmotnou hmotu shlukových vazeb. Každá pružina se používá k reprezentaci shluku polymerních řetězců, namísto části jediného polymerního řetězce. Toto zjednodušení umožňuje přemostit různé modely v měřítku několika délek a výrazně zvyšuje efektivitu simulace. V každém iteračním kroku simulace jsou síly v pružinách aplikovány na uzly ve středu kuliček a jsou vypočítány ekvilibrované uzlové posuny v celém systému. Na rozdíl od tradiční metody FE pro získávání napětí a přetvoření poskytuje model pružina-patka posunutí uzlů a síly v pružinách. Ekvivalentní přetvoření a deformační energii modelu sítě založeného na pružinách lze definovat a vypočítat pomocí posunů uzlů a charakteristik pružiny. Kromě toho lze výsledky síťového modelu zvětšit a získat tak strukturální odezvu v makrozměrech pomocí analýzy FE.[11][12]
- Rozklad pružných / tuhých oblastí a domén proteinů [13][14][15]
- Charakterizace funkčních pohybů a funkčně důležitých míst / zbytků proteinů, enzymů a velkých makromolekulárních sestav [16][11][17][18][19][20][21][22][23][24][25][26]
- Zdokonalení a dynamika strukturálních dat s nízkým rozlišením, např. Kryoelektronová mikroskopie [27][28][29][30]
- Molekulární náhrada k řešení Rentgenové struktury, když konformační změna došlo ke známé struktuře[31]
- Integrace s atomistickými modely a simulacemi [32][33]
- Výzkum drah skládání / rozkládání a kinetika.[34][35]
- Anotace funkčních implikací v molekulární evoluci [36][37]
Webové servery
V praxi lze provádět dva druhy výpočtů. První druh (GNM jako takový) využívá Kirchhoffova matice.[1][2] Druhý druh (konkrétněji nazývaný model Elastic Network Model nebo Anisotropic Network Model) využívá Hesenská matice spojené s odpovídající sadou harmonických pružin.[38] Oba druhy modelů lze použít online pomocí následujících serverů.
GNM servery
- iGNM: Databáze proteinových funkčních pohybů založená na GNM http://ignm.ccbb.pitt.edu [39]
- oGNM: Online výpočet strukturální dynamiky pomocí GNM https://web.archive.org/web/20070516042756/http://ignm.ccbb.pitt.edu/GNM_Online_Calculation.htm
ENM / ANM servery
- Anizotropní model sítě webový server http://www.ccbb.pitt.edu/anm [40]
- elNemo: Webové rozhraní k modelu Elastic Network http://www.sciences.univ-nantes.fr/elnemo/
- AD-ENM: Analýza dynamiky elastického síťového modelu http://enm.lobos.nih.gov/
- WEBnm @: Webový server pro analýzu proteinů v normálním režimu http://apps.cbu.uib.no/webnma/home
Další relevantní servery
- ProDy: Aplikační programové rozhraní (API) v Pythonu, které integruje analýzy GNM a ANM a několik molekulárních strukturních a sekvenčních analýz a vizualizačních nástrojů: http://prody.csb.pitt.edu [41][42]
- HingeProt: Algoritmus pro predikci závěsu proteinu pomocí elastických síťových modelů http://www.prc.boun.edu.tr/appserv/prc/hingeprot/ nebo http://bioinfo3d.cs.tau.ac.il/HingeProt/hingeprot.html
- DNABindProt: Server pro stanovení potenciálních stránek vázajících proteiny DNA http://www.prc.boun.edu.tr/appserv/prc/dnabindprot/
- MolMovDB: Databáze makromolekulárních pohybů: http://www.molmovdb.org/
Viz také
- Gaussovo rozdělení
- Harmonický oscilátor
- Hookeův zákon
- Molekulární dynamika
- Normální mód
- Analýza hlavních komponent
- Dynamika proteinů
- Pryžová pružnost
- Statistická mechanika
Reference
Primární zdroje
- Bahar, I .; Atilgan, A. R .; Erman, B. (1997). „Přímé vyhodnocení teplotních výkyvů v proteinu pomocí harmonického potenciálu s jedním parametrem“. Skládací a design. 2 (3): 173–181. doi:10.1016 / s1359-0278 (97) 00024-2. PMID 9218955.
- Haliloglu, T. Bahar; Erman, B. (1997). "Gaussova dynamika skládaných proteinů". Phys. Rev. Lett. 79 (16): 3090–3093. Bibcode:1997PhRvL..79.3090H. doi:10,1103 / fyzrevlett 79,3090.
- Cui Q, Bahar I, (2006). Analýza v normálním režimu: Teorie a aplikace v biologických a chemických systémech, Chapman & Hall / CRC, Londýn, Velká Británie
Konkrétní citace
- ^ A b Bahar, I .; Atilgan, A. R .; Erman, B. (1997). „Přímé vyhodnocení teplotních výkyvů v proteinu pomocí harmonického potenciálu s jedním parametrem“. Skládací a design. 2 (3): 173–181. doi:10.1016 / s1359-0278 (97) 00024-2. PMID 9218955.
- ^ A b Haliloglu, T. Bahar; Erman, B (1997). "Gaussova dynamika skládaných proteinů". Phys. Rev. Lett. 79 (16): 3090–3093. Bibcode:1997PhRvL..79.3090H. doi:10,1103 / fyzrevlett 79,3090.
- ^ Flory, P.J. (1976). "Statistická termodynamika náhodných sítí". Proc. Roy. Soc. Lond. A. 351 (1666): 351. Bibcode:1976RSPSA.351..351F. doi:10.1098 / rspa.1976.0146.
- ^ Rouse, P.E. (1953). "Teorie lineárních viskoelastických vlastností zředěných roztoků stočených polymerů". J. Chem. Phys. 21 (7): 1272. Bibcode:1953JChPh..21.1272R. doi:10.1063/1.1699180.
- ^ Halle, B (2002). „Flexibilita a balení proteinů“. Proc. Natl. Acad. Sci. USA. 99 (3): 1274–1279. Bibcode:2002PNAS ... 99.1274H. doi:10.1073 / pnas.032522499. PMC 122180. PMID 11818549.
- ^ Bahar, I .; Wallqvist, A .; Covell, D. G .; Jernigan, R.L. (1998). "Korelace mezi výměnou vodíku v nativním stavu a výkyvy kooperativních zbytků z jednoduchého modelu". Biochemie. 37 (4): 1067–1075. CiteSeerX 10.1.1.551.9055. doi:10.1021 / bi9720641. PMID 9454598.
- ^ Bahar, I .; Atilgan, A. R .; Demirel, M. C .; Erman, B. (1998). "Vibrační dynamika proteinů: Význam pomalého a rychlého režimu ve vztahu k funkci a stabilitě". Phys. Rev. Lett. 80 (12): 2733–2736. Bibcode:1998PhRvL..80,2733B. doi:10.1103 / fyzrevlett.80.2733. S2CID 1070176.
- ^ A b Kmiecik, Sebastian; Kouza, Maksim; Badaczewska-Dawid, Aleksandra E .; Kloczkowski, Andrzej; Kolinski, Andrzej (2018). „Modelování strukturní flexibility proteinů a velké dynamiky: hrubozrnné simulace a elastické síťové modely“. International Journal of Molecular Sciences. 19 (11): 3496. doi:10,3390 / ijms19113496. PMC 6274762. PMID 30404229.
- ^ Yang, Lei; Song, Guang; Jernigan, Robert L. (01.08.2007). „Jak dobře dokážeme pochopit pohyby proteinů ve velkém měřítku pomocí normálních režimů modelů elastické sítě?“. Biofyzikální deník. 93 (3): 920–929. Bibcode:2007BpJ .... 93..920Y. doi:10.1529 / biophysj.106.095927. ISSN 0006-3495. PMC 1913142. PMID 17483178.
- ^ Chennubhotla, C; Rader, AJ; Yang, LW; Bahar, I (2005). „Modely elastické sítě pro porozumění biomolekulárním strojům: od enzymů po supramolekulární sestavy“. Phys. Biol. 2 (4): S173 – S180. Bibcode:2005PhBio ... 2S.173C. doi:10.1088 / 1478-3975 / 2/4 / S12. PMID 16280623.
- ^ A b Zhang, Jinjun (2015). „Optimalizovaný síťově propojený model pro simulaci lineární elastické reakce materiálu inteligentního polymeru“. Journal of Intelligent Material Systems and Structures. 27 (11): 1461–1475. doi:10.1177 / 1045389X15595292.
- ^ Zhang, Jinjun (2015). „Nový statistický síťový model založený na pružinových kuličkách pro samy detekující inteligentní polymerní materiály“. Journal of Intelligent Material Systems and Structures. 24 (8): 085022. Bibcode:2015SMaS ... 24h5022Z. doi:10.1088/0964-1726/24/8/085022. hdl:2286 / R.I.35587.
- ^ Hinsen, K (1999). Msgstr "Analýza pohybů domény přibližnými výpočty normálního režimu". Proteiny. 33 (3): 417–429. doi:10.1002 / (sici) 1097-0134 (19981115) 33: 3 <417 :: aid-prot10> 3.0.co; 2-8. PMID 9829700.
- ^ Rader, AJ .; Anderson, G .; Isin, B .; Khorana, H. G .; Bahar, I .; Klein-Seetharaman, J. (2004). "Identifikace základních aminokyselin stabilizujících rhodopsin". Proc. Natl. Acad. Sci. USA. 101 (19): 7246–7251. Bibcode:2004PNAS..101.7246R. doi:10.1073 / pnas.0401429101. PMC 409904. PMID 15123809.
- ^ Kundu, S .; Sorensen, D.C .; Phillips, G.N. Jr (2004). "Automatický doménový rozklad proteinů Gaussovým síťovým modelem". Proteiny. 57 (4): 725–733. doi:10,1002 / prot.20268. PMID 15478120.
- ^ Zhang, Jinjun (2015). „Nový statistický síťový model založený na pružinových kuličkách pro samy detekující inteligentní polymerní materiály“. Chytré materiály a struktury. 24 (8): 085022. Bibcode:2015SMaS ... 24h5022Z. doi:10.1088/0964-1726/24/8/085022. hdl:2286 / R.I.35587.
- ^ Keskin, O .; et al. (2002). „Vztah molekulární flexibility k funkci: případová studie tubulinu“. Biophys. J. 83 (2): 663–80. Bibcode:2002BpJ .... 83..663K. doi:10.1016 / s0006-3495 (02) 75199-0. PMC 1302177. PMID 12124255.
- ^ Temiz NA & Bahar I, Inhibitor binding alteres the direction of domain motions in HIV-1 reverse transcriptase, Proteins: Structure, Function and Genetics 49, 61-70, 2002.
- ^ Xu, C., Tobi, D. a Bahar, I. 2003 Allosterické změny ve struktuře proteinů vypočítané jednoduchým mechanickým modelem: přechod hemoglobinu T <-> R2, J. Mol. Biol., 333, 153.
- ^ Dror Tobi & Ivet Bahar, Strukturální změny podílející se na vazbě proteinů, korelují s vnitřními pohyby proteinů v nevázaném stavu, Proc Natl Acad Sci (USA) 102, 18908-18913, 2005.
- ^ Shrivastava, Indira H .; Bahar, Ivet (2006). „Společný mechanismus otevírání pórů sdílený pěti různými draslíkovými kanály“. Biophys J.. 90 (11): 3929–3940. Bibcode:2006BpJ .... 90,3929S. doi:10.1529 / biophysj.105.080093. PMC 1459499. PMID 16533848.
- ^ Yang, LW; Bahar, I (2005). „Spojení mezi katalytickým stanovištěm a kolektivní dynamikou: požadavek na mechanochemickou aktivitu enzymů“. Struktura. 13 (6): 893–904. doi:10.1016 / j.str.2005.03.015. PMC 1489920. PMID 15939021.
- ^ Chennubhotla, čakra; Bahar, Ivet (2006). „Markovovy metody pro hierarchické hrubozrnné dynamiky velkých proteinů“. Přednášky z informatiky. 3909: 379–393. doi:10.1007/11732990_32. ISBN 978-3-540-33295-4.
- ^ Wang, Y. Rader; AJ; Bahar, I .; Jernigan, RL. (2004). "Globální pohyby ribozomu odhaleny pomocí modelu elastické sítě". J. Struct. Biol. 147 (3): 302–314. doi:10.1016 / j.jsb.2004.01.005. PMID 15450299.
- ^ Rader, AJ; Vlad, Daniel; Bahar, Ivet (2005). "Dynamika dozrávání bakteriofága HK97 kapsida". Struktura. 13 (3): 413–21. doi:10.1016 / j.str.2004.12.015. PMID 15766543.
- ^ Hamacher, K .; Trylska, J .; McCammon, J.A. (2006). "Mapa závislosti proteinů v malé ribozomální podjednotce". PLOS Comput. Biol. 2 (2): e10. Bibcode:2006PLSCB ... 2 ... 10H. doi:10.1371 / journal.pcbi.0020010. PMC 1364506. PMID 16485038.
- ^ Ming, D .; et al. (2002). "Jak popsat pohyb bílkovin bez aminokyselinové sekvence a atomových souřadnic". Proc. Natl. Acad. Sci. USA. 99 (13): 8620–8625. Bibcode:2002PNAS ... 99.8620M. doi:10.1073 / pnas.082148899. PMC 124334. PMID 12084922.
- ^ Tama, F .; Wriggers, W .; Brooks III, C.L. (2002). „Zkoumání globálních narušení biologických makromolekul a sestav ze strukturální informace s nízkým rozlišením a teorie elastické sítě“. J. Mol. Biol. 321 (2): 297–305. CiteSeerX 10.1.1.457.8. doi:10.1016 / s0022-2836 (02) 00627-7. PMID 12144786.
- ^ Delarue, M .; Dumas, P. (2004). „O použití nízkofrekvenčních normálních režimů k vynucení kolektivních pohybů při zušlechťování makromolekulárních strukturálních modelů“. Proc. Natl. Acad. Sci. USA. 101 (18): 6957–6962. Bibcode:2004PNAS..101.6957D. doi:10.1073 / pnas.0400301101. PMC 406448. PMID 15096585.
- ^ Micheletti, C .; Carloni, P .; Maritan, A. (2004). ""Přesný a efektivní popis proteinové vibrační dynamiky „porovnání molekulární dynamiky a gaussovských modelů“. Proteiny. 55 (3): 635–45. arXiv:cond-mat / 0405145. Bibcode:2004.mat ...5145 mil. doi:10.1002 / prot.20049. PMID 15103627.
- ^ Suhre, K .; Sanejouand, Y.H. (2004). "O potenciálu analýzy v normálním režimu pro řešení složitých problémů s molekulární náhradou". Acta Crystallogr. D. 60 (4): 796–9. doi:10.1107 / s0907444904001982. PMID 15039589.
- ^ Zhang, Z.Y .; Shi, Y.Y .; Liu, H.Y. (2003). „Simulace molekulární dynamiky peptidů a proteinů se zesílenými kolektivními pohyby“. Biophys. J. 84 (6): 3583–93. Bibcode:2003BpJ .... 84,3583Z. doi:10.1016 / s0006-3495 (03) 75090-5. PMC 1302944. PMID 12770868.
- ^ Micheletti, C .; Lattanzi, G .; Maritan, A. (2002). "Elastické vlastnosti proteinů: pohled na proces skládání a evoluční výběr nativních struktur". J. Mol. Biol. 321 (5): 909–21. arXiv:cond-mat / 0204400. Bibcode:2002.mat. Mat. 4400 mil. doi:10.1016 / s0022-2836 (02) 00710-6. PMID 12206770.
- ^ Micheletti, C .; et al. (2002). „Zásadní fáze skládání bílkovin prostřednictvím řešitelného modelu: předpovídání cílových míst pro léky inhibující enzymy“. Věda o bílkovinách. 11 (8): 1878–87. arXiv:cond-mat / 0209325. Bibcode:2002.mat. Mat. 9325 mil. doi:10.1110 / ps.3360102. PMC 2373687. PMID 12142442.
- ^ Portman, J. J.; Takada, S .; Wolynes, P.G. (2001). „Mikroskopická teorie rychlostí skládání proteinů. I. jemná struktura profilu volné energie a cesty skládání z variačního přístupu“. J. Chem. Phys. 114 (11): 5069. arXiv:cond-mat / 0008454. Bibcode:2001JChPh.114.5069P. doi:10.1063/1.1334662.
- ^ Hamacher, K (2008). „Souvisí vývoj sekvence proteázy HIV1 s její základní molekulární mechanikou“. Gen. 422 (1–2): 30–36. doi:10.1016 / j.gene.2008.06.007. PMID 18590806.
- ^ Hamacher, K .; McCammon, J.A. (2006). "Výpočet aminokyselinové specificity fluktuací v biomolekulárních systémech". J. Chem. Theory Comput. 2 (3): 873–8. doi:10.1021 / ct050247s. PMID 26626694.
- ^ Tirion, M.M. (1996). "Elastické pohyby s velkou amplitudou v proteinech z atomové analýzy s jedním parametrem". Phys. Rev. Lett. 77 (9): 1905–1908. Bibcode:1996PhRvL..77.1905T. doi:10.1103 / fyzrevlett.77.1905. PMID 10063201.
- ^ Li, H., Chang, Y. Y., Yang, L. W. a Bahar, I. (2016). iGNM 2.0: Gaussova databáze síťových modelů pro biomolekulární strukturní dynamiku. Nucleic Acids Research, 44 (D1), D415-D422.
- ^ Atilgan, AR; Durrell, SR; Jernigan, RL; Demirel, MC; Keskin, O .; Bahar, I. (2001). „Anizotropie fluktuační dynamiky proteinů s elastickým síťovým modelem“. Biophys. J. 80 (1): 505–515. Bibcode:2001BpJ .... 80..505A. doi:10.1016 / s0006-3495 (01) 76033-x. PMC 1301252. PMID 11159421.
- ^ Bakan, A .; Meireles, L. M .; Bahar, I. (2011). „ProDy: dynamika proteinů odvozená z teorie a experimentů“. Bioinformatika. 27 (11): 1575–1577. doi:10.1093 / bioinformatika / btr168. PMC 3102222. PMID 21471012.
- ^ Bakan, A .; Dutta, A .; Mao, W .; Liu, Y .; Chennubhotla, C .; Lezon, T. R .; Bahar, I. (2014). „Evol a ProDy pro přemostění evoluce proteinových sekvencí a strukturální dynamiky“. Bioinformatika. 30 (18): 2681–2683. doi:10.1093 / bioinformatika / btu336. PMC 4155247. PMID 24849577.

![V_{GNM} = frac{gamma}{2}left[ sum_{i,j}^{N} (Delta R_j-Delta R_i)^2 ight]=
frac{gamma}{2}left[ sum_{i,j}^{N} Delta R_i Gamma_{ij} Delta R_jight]](https://wikimedia.org/api/rest_v1/media/math/render/svg/d5be28f13b07a00cf754058700cee674e9badb75)

![V_{GNM} = frac{gamma}{2} [Delta X^TGamma Delta X + Delta Y^TGamma Delta Y + Delta Z^TGamma Delta Z]](https://wikimedia.org/api/rest_v1/media/math/render/svg/e69f61521a50a6c344b039f8d8c5e74a141c239c)













![<Delta R_i cdot Delta R_j> = frac{3 k_B T}{gamma}[ULambda^{-1}U^T]_{ij}=frac{3 k_B T}{gamma}sum_{k=1}^{N-1}lambda_k^{-1} [u_k u_k^T]_{ij}](https://wikimedia.org/api/rest_v1/media/math/render/svg/cba38d6c9778162a706d15d897151e128670cd23)
![[Delta R_i cdot Delta R_j]_k = frac{3 k_B T}{gamma}lambda_k^{-1} [u_k]_i [u_k]_j](https://wikimedia.org/api/rest_v1/media/math/render/svg/8284898bdd32ed64bd4ca2eb3c5274540809d2b4)
