Riboswitch PreQ1 - PreQ1 riboswitch
| Riboswitch PreQ1 | |
|---|---|
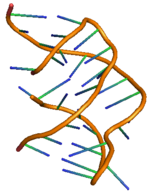
 Předpovězeno sekundární struktura a zachování sekvence PreQ1 | |
| Identifikátory | |
| Symbol | PreQ1 |
| Rfam | RF00522 |
| Další údaje | |
| RNA typ | Cis-reg; riboswitch |
| Domény | Bakterie |
| TAK | SO: 0000035 |
| PDB struktur | PDBe 2L1V |
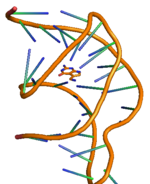
The PreQ1-I riboswitch je cis působící prvek identifikováno v bakterie který reguluje expresi genů zapojených do biosyntéza z nukleosid queuosin (Q) z GTP.[1] PreQ1 (pre-queuosin1) je meziprodukt v cestě queuosinu a preQ1 riboswitch, jako typ riboswitch, je prvek RNA, který váže preQ1. PreQ1 riboswitch se vyznačuje neobvykle malým aptamerem ve srovnání s jinými riboswitchi. Jeho trojrozměrná struktura s atomovým rozlišením byla stanovena pomocí PDB ID 2L1V.[2][3]
PreQ1 Klasifikace
Tři podkategorie PreQ1 riboswitch existují: preQ1-I, preQ1-II a preQ1-III. PreQ1-Mám zřetelně malý aptamer, dlouhý od 25 do 45 nukleotidů,[4] ve srovnání se strukturami PreQ1-II riboswitch a preQ1-III riboswitch. PreQ1-II riboswitch, nalezen pouze v Lactobacillales, má větší a složitější konsensuální sekvence a struktura než preQ1-I riboswitch, s průměrem 58 nukleotidů tvořících jeho aptamer, který tvoří až pět podstruktur spárovaných bází.[5] PreQ1-III riboswitch má odlišnou strukturu a je také větší ve velikosti aptameru než preQ1-I riboswitch, od velikostí od 33 do 58 nukleotidů. PreQ1-III riboswitch má atypicky organizovaný pseudoknot to nejeví, že by začlenilo svoji následnou expresní platformu ribosomové vazebné místo (RBS).[6]
Dějiny
Zatímco preQ1 byl poprvé objeven jako antikodon sekvence tRNA z E-coli v roce 1972,[7] riboswitch preQ1 nebyl poprvé nalezen až do roku 2004[8] a poznal i později.[9] První hlášené preQ1 riboswitch byl umístěn ve vůdci operonu Bacillus subtilis ykvJKLM (queCDEF), který kóduje čtyři geny nezbytné pro produkci queuosinu.[8] V tomto organismu PreQ1 vazba na riboswitch aptamer se předpokládá, že indukuje předčasně transkripce ukončení v rámci vedoucího k down-regulaci exprese těchto genů. Později byl preQ1 riboswitch identifikován jako konzervovaná sekvence na 5 'UTR genů v mnoha grampozitivních bakteriích a bylo prokázáno, že je spojen se syntézou preQ1.[9]
V roce 2008 druhá třída preQ1 riboswitch (Riboswitche PreQ1-II ) byl také nalezen jako zástupce motivu COG4708 RNA z Streptococcus pneumoniae R6.[10] Ačkoli PreQ1-II riboswitch funguje také jako biosyntetický meziprodukt queuosinu, strukturní a molekulární charakteristiky jsou odlišné od riboswitche preQ1-I, což naznačuje, že přirozené aptamery využívající různé struktury k vázání stejného metabolitu mohou být častější, než je v současné době známo.[10]
Struktura a funkce


Riboswitch PreQ1 má dva stonky a tři smyčky a jeho podrobná struktura je zobrazena vpravo.[11] Riboswitching akce preQ1 riboswitche v bakteriích jsou regulovány vazbou metabolitu preQ1 do oblasti aptameru vedoucí ke strukturálním změnám v messengerové RNA (mRNA), která řídí downstream genetickou regulaci.[12] PreQ1 struktura riboswitch využívá kompaktní typ H. pseudoknot, což ho zcela odlišuje od ostatních purin založené riboswitche.[12] PreQ1 ligand je pohřben v jádru pseudoknotu a stabilizovaný interkalace mezi spirálovými vrstvami a interakcí vodíkové vazby s heteroatomy. Při absenci preQ1 je oblast ocasu P2 mimo oblast smyčky P2 a proto je pozorováno, že riboswitch je v neukotveném (částečně ukotveném) stavu, zatímco na vazbě preQ1 k riboswitchi se obě oblasti P2 přiblíží a způsobí úplné ukotvení riboswitche. Tento dokovací a dokovací mechanismus riboswitche se změnou koncentrace ligandu preQ1 je pozorováno, že řídí signalizaci genové regulace, běžně známou jako „ON“ nebo „OFF“ signalizace pro genovou expresi.[11][13] Bylo pozorováno, že dokovací a vyjímací mechanismus je ovlivňován nejen ligandem, ale také dalšími faktory, jako je Mg sůl.[14] Jako každý jiný riboswitch, dva nejběžnější typy regulace genů zprostředkované preQ1 riboswitch jsou skrz transkripce útlum nebo inhibice překlad zahájení. Ligand vazba na transkripční riboswitch v bakteriích způsobuje modifikaci ve struktuře riboswitchové jednotky, což vede k překážce v aktivitě RNA polymeráza způsobující útlum transkripce. Podobně vazba ligandu na translační riboswitch způsobuje modifikaci v sekundární struktuře riboswitchové jednotky, což vede k omezení vazby ribozomu, a tím k inhibici překladový zahájení.
Transkripční regulace
PreQ1 zprostředkovaný transkripční útlum je řízen dynamickým přepínáním anti-terminátorové a terminátorové vlásenky v riboswitchi.[11] Pro preQ1 riboswitch z bakterie Bacillus subtilis (Bsu), předpokládá se, že anti-terminátor bude méně stabilní než terminátor, protože přidání preQ1 významně posouvá rovnováhu směrem k tvorbě terminátoru.[11] V přítomnosti preQ1, 3 'konec adenin páry bohatých ocasních domén se středem vlásenkové smyčky P1 tvoří H-typ pseudoknot.[11] V nativní struktuře mRNA se váže preQ1 do oblasti aptameru v riboswitchi vede k vytvoření vlásenky terminátoru, která způsobí, že RNA polymeráza zastaví transkripci, což je proces, který je obecně známý jako OFF- regulace genetické exprese nebo ukončení transkripce.[13]
Regulace překladu
Překlad bílkovin v prokaryoty je zahájeno vazbou 30S ribozomální podjednotka do Shine-Dalgarno (SD) sekvence v mRNA. PreQ1 zprostředkovaná inhibice translační regulace je řízena blokováním Sekvence Shine-Dalgarno z mRNA zabránit vazbě ribozom na mRNA pro překlad. Vazba preQ1 na aptamerovou doménu podporuje sekvestraci části SD sekvence na 5 'konci k kmeni P2 aptamerové domény, což způsobuje nedostupnost SD sekvence.[11] Translační riboswitch z bakterií Thermoanaerobacter tengcongensis (Tte) bylo pozorováno, že je přechodně uzavřeno (předem ukotven) v nepřítomnosti preQ1, zatímco v přítomnosti preQ1 přijme se plně zakotvený stav. Tato rovnováha dokování / vyjímání doku není regulována pouze koncentrací ligandu, ale také koncentrací Mg soli.[14][15] Nedostupnost sekvence SD kvůli tvorbě pseudoknot v přítomnosti preQ1 ukazuje OFF-regulaci genetické exprese v translačním riboswitchi nebo inhibici translační iniciace.
Fyziologický význam v regulaci bakteriálních genů
PreQ1 činnost riboswitche v Tte bakterie lze měřit hladinami dvou proteinů, které jsou v oblast kódování z Tte mRNA, což jsou TTE1564 a TTE1563.[16] Proteiny po proudu preQ1 riboswitch biosyntetizuje nukleobázi zvanou fronta a nukleosid queuosin jsou inhibovány aktivací riboswitche preQ1. Queuine je zapojena do antikodon sled jistých tRNA.[17] U bakterií zaujímá hypermodifikovaný nukleobázový queuin první antikodonovou pozici viklat pozice v tRNA Asparagin, Kyselina asparagová, Histidin, a Tyrosin.[18] V bakteriích enzym tRNA-guanin transglykosyláza (TGT) katalyzuje výměnu guaninu v pozici 34 tRNA s queuinem do první anitcodonové polohy.[15][16] Eukaryae začleňují queuin do RNA, zatímco eubakterie začleňují preQ1, který poté prochází modifikací za vzniku queuinu.[17] Protože queuosin se vyrábí výlučně v bakteriích, musí si eukaryotické organismy zajistit přísun queuosinu nebo jeho nukleobázového queuinu ze stravy nebo bakterií ze svých střevní mikroflóra. Důsledkem nedostatku queuinu nebo queuosinu je neschopnost vytvořit queuosinem modifikovanou tRNA a dále neschopnost buňky převést fenylalanin na tyrosin.[19]
Viz také
Reference
- ^ Roth A, Winkler WC, Regulski EE, Lee BW, Lim J, Jona I, Barrick JE, Ritwik A, Kim JN, Welz R, Iwata-Reuyl D, Breaker RR (2007). „Riboswitch selektivní pro prekurzor queuosinu preQ1 obsahuje neobvykle malou doménu aptameru“. Nat Struct Mol Biol. 14 (4): 308–317. doi:10.1038 / nsmb1224. PMID 17384645.
- ^ Klein DJ, Edwards TE, Ferré-D'Amaré AR (březen 2009). „Kokrystalická struktura riboswitche třídy I preQ1 odhaluje pseudoknot rozpoznávající základní hypermodifikovanou nukleobázi“. Nat. Struct. Mol. Biol. 16 (3): 343–344. doi:10.1038 / nsmb.1563. PMC 2657927. PMID 19234468.
- ^ Kang M, Peterson R, Feigon J (březen 2009). „Structural Insights into riboswitch control of the biosynthesis of queuosine, a modified nucleotide found in the anticodon of tRNA“. Mol. Buňka. 33 (6): 784–790. doi:10.1016 / j.molcel.2009.02.019. PMID 19285444.
- ^ "RIBOSPÍNAČE: KLASIFIKACE, FUNKCE a INSILICO PŘÍSTUP". International Journal of Pharma Sciences and Research (IJPSR). 1 (9): 414. 2010.
- ^ McCown, PJ; Liang, JJ; Weinberg, Z; Breaker, RR (17. července 2014). „Strukturální, funkční a taxonomická rozmanitost tří tříd riboswitchů PreQ1“. Chemie a biologie. 21 (7): 880–889. doi:10.1016 / j.chembiol.2014.05.015. PMC 4145258. PMID 25036777.
- ^ Liberman, Joseph A .; Suddala, Krishna C .; Aytenfisu, Asaminew; Chan, Dalen; Belashov, Ivan A .; Salim, Mohammad; Mathews, David H .; Spitale, Robert C .; Walter, Nils G. (07.07.2015). „Strukturální analýza riboswitche preQ1 třídy III odhalila aptamer vzdálený od místa vázajícího ribozom regulovaného rychlou dynamikou“. Sborník Národní akademie věd. 112 (27): E3485 – E3494. doi:10.1073 / pnas.1503955112. ISSN 0027-8424. PMC 4500280. PMID 26106162.
- ^ Harada, Fumio; Nishimura, Susumu (leden 1972). "Možné antikodonové sekvence tRNAHis, tRNAAsn a tRNAAsp z Escherichia coli. Univerzální přítomnost nukleosidu O v první poloze antikodonů těchto transferových ribonukleových kyselin." Biochemie. 11 (2): 301–308. doi:10.1021 / bi00752a024. PMID 4550561.
- ^ A b Reader JS, Metzgar D, Schimmel P, de Crécy-Lagard V (2004). "Identifikace čtyř genů nezbytných pro biosyntézu modifikovaného nukleosidového queuosinu". J. Biol. Chem. 279 (8): 6280–6285. doi:10,1074 / jbc.M310858200. PMID 14660578.
- ^ A b Roth, Adam; Winkler, Wade C .; Regulski, Elizabeth E .; Lee, Bobby W. K .; Lim, Jinsoo; Jona, Inbal; Barrick, Jeffrey E .; Ritwik, Ankita; Kim, Jane N. (01.04.2007). Msgstr "Riboswitch selektivní pro prekurzor queuosinu preQ1 obsahuje neobvykle malou doménu aptameru". Přírodní strukturní a molekulární biologie. 14 (4): 308–317. doi:10.1038 / nsmb1224. ISSN 1545-9993. PMID 17384645.
- ^ A b Meyer, Michelle M .; Roth, Adam; Chervin, Stephanie M .; Garcia, George A .; Breaker, Ronald R. (01.04.2008). „Potvrzení druhé třídy přírodních aptamerů preQ1 u bakterií Streptococcaceae“. RNA. 14 (4): 685–695. doi:10,1261 / rna.937308. ISSN 1355-8382. PMC 2271366. PMID 18305186.
- ^ A b C d E F Eichhorn, Catherine D .; Kang, Mijeong; Feigon, Juli (01.10.2014). "Struktura a funkce riboswitchů preQ1". Biochimica et Biophysica Acta (BBA) - regulační mechanismy genů. Riboswitches. 1839 (10): 939–950. doi:10.1016 / j.bbagrm.2014.04.019. PMC 4177978. PMID 24798077.
- ^ A b Serganov, Alexander; Patel, Dinshaw J. (11. 5. 2012). "Principy rozpoznávání metabolitů a molekulární mechanismy, které jsou základem funkce Riboswitch". Roční přehled biofyziky. 41 (1): 343–370. doi:10.1146 / annurev-biophys-101211-113224. ISSN 1936-122X. PMC 4696762. PMID 22577823.
- ^ A b Rieder, Ulrike; Kreutz, Christoph; Micura, Ronald (2010-06-15). "Skládání transkripčně působícího PreQ1 riboswitche". Sborník Národní akademie věd. 107 (24): 10804–10809. doi:10.1073 / pnas.0914925107. ISSN 0027-8424. PMC 2890745. PMID 20534493.
- ^ A b Suddala, Krishna C .; Rinaldi, Arlie J .; Feng, červen; Mustoe, Anthony M .; Eichhorn, Catherine D .; Liberman, Joseph A .; Wedekind, Joseph E .; Al-Hashimi, Hashim M .; Brooks, Charles L. (01.12.2013). „Jednotlivé transkripční a translační preQ1 riboswitche přijímají podobné předem složené soubory, které sledují odlišné skládací dráhy do stejné struktury vázané na ligand“. Výzkum nukleových kyselin. 41 (22): 10462–10475. doi:10.1093 / nar / gkt798. ISSN 0305-1048. PMC 3905878. PMID 24003028.
- ^ A b Suddala, Krishna C .; Wang, Jiarui; Hou, Qian; Walter, Nils G. (11.11.2015). „Mg2 + posouvá skládání Riboswitche zprostředkované ligandem z indukovaného přizpůsobení na konformační výběr“. Journal of the American Chemical Society. 137 (44): 14075–14083. doi:10.1021 / jacs.5b09740. ISSN 0002-7863. PMC 5098500. PMID 26471732.
- ^ A b Rinaldi, Arlie J .; Lund, Paul E .; Blanco, Mario R .; Walter, Nils G. (2016-01-19). „Sekvence Shine-Dalgarno jednotlivých mRNA regulovaných riboswitchem ukazuje shluky přístupnosti závislé na ligandu“. Příroda komunikace. 7: 8976. doi:10.1038 / ncomms9976. ISSN 2041-1723. PMC 4735710. PMID 26781350.
- ^ A b Kittendorf, Jeffrey D .; Sgraja, Tanja; Reuter, Klaus; Klebe, Gerhard; Garcia, George A. (2003-10-24). „Základní role pro aspartát 264 při katalýze pomocí tRNA-guanin-transglykosylázy z Escherichia coli“. Journal of Biological Chemistry. 278 (43): 42369–42376. doi:10,1074 / jbc.M304323200. ISSN 0021-9258. PMID 12909636.
- ^ Jenkins, Jermaine L .; Kručinská, Jolanta; McCarty, Reid M .; Bandarian, Vahe; Wedekind, Joseph E. (2011-07-15). „Srovnání aptameru preQ1 Riboswitch v metabolicky vázaných a volných státech s důsledky pro regulaci genů“. Journal of Biological Chemistry. 286 (28): 24626–24637. doi:10.1074 / jbc.M111.230375. ISSN 0021-9258. PMC 3137038. PMID 21592962.
- ^ Rakovich, Tatsiana; Boland, Coilin; Bernstein, Ilana; Chikwana, Vimbai M .; Iwata-Reuyl, Dirk; Kelly, Vincent P. (06.06.2011). „Nedostatek queuosinu v eukaryotech kompromisuje produkci tyrosinu zvýšenou oxidací tetrahydrobiopterinu“. Journal of Biological Chemistry. 286 (22): 19354–19363. doi:10.1074 / jbc.M111.219576. ISSN 0021-9258. PMC 3103313. PMID 21487017.