Bioimage informatika - Bioimage informatics
Bioimage informatika je podpole z bioinformatika a výpočetní biologie.[1] Zaměřuje se na použití výpočetních technik k analýze bioobrazů, zejména buněčných a molekulárních obrazů, ve velkém měřítku a s vysokou propustností. Cílem je získat užitečné znalosti z komplikovaného a heterogenního obrazu a souvisejících metadata.
Automatizované mikroskopy jsou schopny sbírat velké množství obrázků s minimálním zásahem. To vedlo k datové explozi, která bezpodmínečně vyžaduje automatické zpracování. Navíc a překvapivě pro některé z těchto úkolů existují důkazy, že automatizované systémy mohou fungovat lépe než lidé.[2][3] Automatizované systémy jsou navíc nestranné, na rozdíl od lidské analýzy, jejíž hodnocení může (i nevědomě) ovlivnit požadovaný výsledek.
Stále více se zaměřuje na vývoj románu zpracování obrazu, počítačové vidění, dolování dat, databázové a vizualizační techniky pro extrakci, srovnání, vyhledávání a správu biologických znalostí v těchto problémech náročných na data.[4][5]
Datové modality
Používá se několik systémů a platforem pro sběr dat, které vyžadují optimální zpracování různých metod.
Fluorescenční mikroskopie
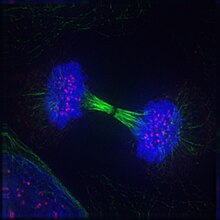
Fluorescenční mikroskopie umožňuje přímou vizualizaci molekul na subcelulární úrovni, v živé i pevný buňky. Molekuly zájmu jsou označeny buď zelený fluorescenční protein (GFP), jiný fluorescenční protein nebo fluorescenčně značený protilátka. Pravidelně se používá několik typů mikroskopů: widefield, konfokální nebo dva fotony. Většina mikroskopických systémů bude také podporovat sběr časových řad (filmů).
Obecně se filtry používají tak, aby se každé barvivo zobrazovalo samostatně (například k zobrazení se používá modrý filtr Hoechst, poté rychle přepnut na zelený filtr na obrázek GFP). Kvůli spotřebě se obrázky často zobrazují v falešná barva zobrazením každého kanálu v jiné barvě, ale nemusí to ani souviset s původními použitými vlnovými délkami. V některých případech mohl být původní obraz získán i v neviditelných vlnových délkách (běžná je infračervená oblast).
Volby ve fázi pořizování obrazu ovlivní analýzu a často vyžadují speciální zpracování. Konfokální hromádky budou vyžadovat 3D zpracování a širokopásmové pseudo-hromádky budou často těžit digitální dekonvoluce k odstranění rozostřeného světla.
Příchod automatizovaných mikroskopů, které mohou automaticky získávat mnoho obrazů, je jedním z důvodů, proč nelze provést analýzu okem (jinak by se anotace rychle stala překážkou výzkumu). Použití automatizovaných mikroskopů znamená, že některé obrázky mohou být rozostřené (automatizované systémy pro zjišťování zaostření mohou být někdy nesprávné), obsahovat malý počet buněk nebo být naplněny nečistotami. Proto bude generování obrazů obtížnější analyzovat než obrazy pořízené operátorem, protože by si vybral jiné umístění pro správné zobrazení a zaostření. Na druhou stranu by operátor mohl při svém výběru zavést nevědomé zkreslení výběrem pouze buněk, jejichž fenotyp se nejvíce podobá fenotypu očekávanému před experimentem.
Histologie

Histologie je aplikace pro mikroskopii, kde jsou tkáňové řezy obarveny a pozorovány pod mikroskopem (obvykle světelný mikroskop, ale používá se také elektronová mikroskopie).
Při použití světelného mikroskopu se obrázky, na rozdíl od fluorescenčního zobrazování, obvykle získávají pomocí standardních barevných kamerových systémů. To částečně odráží historii pole, kde lidé často interpretovali obrazy, ale také skutečnost, že vzorek může být osvětlen bílým světlem a veškeré světlo shromážděno, než aby muselo vzrušovat fluorofory. Pokud je použito více než jedno barvivo, nezbytným krokem předzpracování je smíchání kanálů a obnovení odhadu čistých intenzit specifických pro barvivo.
Ukázalo se, že subcelulární umístění obarvených proteinů lze identifikovat z histologických obrazů.
Pokud je cílem lékařská diagnostika, pak histologické aplikace často spadnou do říše digitální patologie nebo automatizovaná analýza tkáňového obrazu, což jsou sesterská pole bioimage informatiky. Často jsou použitelné stejné výpočetní techniky, ale cíle jsou spíše medicínské než výzkumné.
Důležité problémy
Analýza subcelulární polohy

Subcelulární lokalizační analýza byla jedním z počátečních problémů v této oblasti. V režimu s dohledem je problém naučit se klasifikátor, který dokáže rozpoznat obrázky z hlavní buňky organely na základě obrázků.
Použité metody jsou založeny na strojové učení, budova a diskriminační klasifikátor na základě numerické funkce vypočítané z obrázku. Funkce jsou buď obecné funkce z počítačové vidění, jako Funkce textury Haralick nebo vlastnosti speciálně navržené k zachycení biologických faktorů (např. typickým příkladem je společná lokalizace s jaderným markerem).
U základního problému identifikace organel lze dosáhnout velmi vysokých hodnot přesnosti, včetně lepších než? Výsledek.[2] Tyto metody jsou užitečné při základním výzkumu buněčné biologie, ale byly také použity při objevování proteinů, jejichž umístění se mění v rakovinných buňkách.[6]
Klasifikace do organel je však omezenou formou problému, protože mnoho proteinů se bude lokalizovat na více míst současně (smíšené vzory) a lze rozlišit mnoho vzorů, i když se nejedná o odlišné komponenty vázané na membránu. V této oblasti existuje několik nevyřešených problémů a výzkum pokračuje.
Screening s vysokým obsahem

Obrazovky s vysokou propustností využívající technologii automatizovaného zobrazování (někdy nazývané vysoce obsahový screening ) se staly standardní metodou jak pro objevování drog, tak pro základní biologický výzkum. Pomocí vícejamkových destiček, robotiky a automatizované mikroskopie lze stejný test aplikovat na velkou knihovnu možných reagencií (obvykle buď malé molekuly nebo RNAi ) velmi rychle a v krátkém čase získáte tisíce obrázků. Vzhledem k velkému objemu generovaných dat je automatická analýza obrazu nutností.[7]
Pokud jsou k dispozici pozitivní a negativní kontroly, lze k problému přistupovat jako k problému klasifikace a lze použít stejné techniky výpočtu a klasifikace prvků, které se používají pro analýzu subcelulární lokalizace.
Segmentace
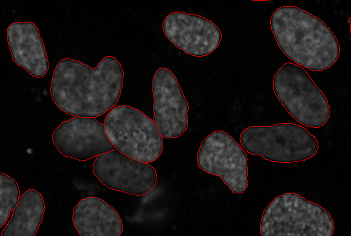
Segmentace buněk je důležitým dílčím problémem v mnoha polích níže (a někdy užitečná sama o sobě, pokud je cílem pouze získat počet buněk v test životaschopnosti ). Cílem je identifikovat hranice buněk v obrazu více buněk. To umožňuje zpracování každé buňky jednotlivě pro měření parametrů. U 3D dat musí být segmentace provedena ve 3D prostoru.
Vzhledem k tomu, že zobrazování jaderného markeru je běžné u mnoha obrazů, je široce používaným protokolem segmentace jader. To může být užitečné samo o sobě, pokud jsou zapotřebí jaderná měření, nebo to může sloužit k nasazení a povodí což rozšiřuje segmentaci na celý obraz.
Všechny hlavní metody segmentace byly hlášeny na obrázcích buněk, od jednoduchých prahování k metodám nastavení úrovní. Protože existuje více obrazových modalit a různé typy buněk, z nichž každá implikuje různé kompromisy, neexistuje pro tento problém jediné přijaté řešení.
Segmentace buněčného obrazu jako důležitý postup se často používá ke studiu genové exprese a kolokačního vztahu atd. Jednotlivých buněk. V takových případech analýzy jednotlivých buněk je často nutné jednoznačně určit identitu buněk při segmentaci buněk. Takový úkol rozpoznávání je často výpočetně netriviální. U modelových organismů, jako je C. elegans, které mají dobře definované buněčné linie, je možné explicitně rozpoznat identitu buněk pomocí obrazové analýzy kombinací jak segmentace obrazu, tak metod rozpoznávání vzorů.[9] Současná segmentace a rozpoznávání buněk[10] bylo také navrženo jako přesnější řešení tohoto problému, když je k dispozici „atlas“ nebo jiné předchozí informace o buňkách. Protože genovou expresi při rozlišení jedné buňky lze získat pomocí těchto typů přístupů založených na zobrazování, je možné kombinovat tyto metody s jinými metodami kvantifikace genové exprese jedné buňky, jako je RNAseq.
Sledování
Sledování je další tradiční problém se zpracováním obrazu, který se objevuje v bioimage informatice. Problém je v souvislosti s objekty, které se objevují v následujících snímcích filmu. Stejně jako u segmentace lze problém představovat jak ve dvojrozměrné, tak v trojrozměrné formě.[11]
V případě fluorescenčního zobrazování musí být sledování často prováděno na obrázcích s velmi nízkým kontrastem. Protože získání vysokého kontrastu se provádí zářením více světla, které poškodí vzorek a ničí barvivo, osvětlení je udržováno na minimu. Často je užitečné myslet na rozpočet fotonů: počet fotonů, které lze použít pro zobrazování před poškozením vzorku, je tak velký, že datům již nelze důvěřovat. Pokud tedy chcete získat vysoce kontrastní obrázky, lze použít pouze několik snímků; zatímco u dlouhých filmů bude mít každý snímek velmi nízký kontrast.
Registrace
Když vezmeme v úvahu vzorky obrazových dat různé povahy, například ty, které odpovídají různým metodám označování, různým jednotlivcům, vzorkům v různých časových bodech atd., Je pro lepší srovnání často nutné obrázky zaregistrovat. Jedním z příkladů je, že jsou shromažďována data časových průběhů, obrázky v následujících rámcích musí být často registrovaný aby bylo možné opravit menší posuny v poloze kamery. Dalším příkladem je, že když mnoho obrázků modelového zvířete (např. C. elegans nebo Mozek Drosophila nebo a myší mozek ) jsou shromážděny, často existuje značná potřeba registrovat tyto obrázky pro srovnání jejich vzorů (např. ty, které odpovídají stejné nebo odlišné populaci neuronů, ty, které sdílejí nebo se liší v genové expresi atd.).
Softwarové balíčky pro registraci lékařských obrazů byly prvními pokusy o použití pro aplikace pro registraci mikroskopických obrazů. Vzhledem k často mnohem větší velikosti obrazového souboru a mnohem většímu počtu vzorků v experimentech je však v mnoha případech nutné vyvinout nový software pro registraci 3D obrazu. The BrainAligner[12] je software, který byl použit k automatizaci procesu 3D deformovatelné a nelineární registrace pomocí strategie spolehlivého porovnávání orientačních bodů. Primárně se používá ke generování více než 50 000 3D standardizovaných obrazů mozku ovocných mušek na farmě Janelia Farm of HHMI s dalšími aplikacemi, včetně vážek a myší.
Důležitá místa
Konsorcium vědců z univerzit a výzkumných ústavů pořádalo výroční setkání o informatice v oblasti bioimage[13] od roku 2005. The ISMB konference měla Bioimaging a vizualizace dat sledovat od roku 2010. Časopis Bioinformatika také představil a Bioimage Informatika trať v roce 2012. Časopis OpenAccess BMC bioinformatika má sekci věnovanou analýze bioobrazů, vizualizaci a souvisejícím aplikacím. jiný časopisy o výpočetní biologii a bioinformatice také pravidelně publikovat práci s informatikou bioimage. Akční akce Evropské unie nazvaná NEUBIAS (síť evropských analytiků bioimage) organizuje od roku 2017 výroční konference i školicí školy a taggathony analytiků bioimage.
Software
Existuje několik balíčků, které zpřístupňují metody informatiky bioimage prostřednictvím grafického uživatelského rozhraní, jako je Obrázek J., FIJI, CellProfiler nebo Ledový. Vizualizační a analytické platformy, jako je Vaa3D se objevily v posledních letech a byly použity v obou velkých projektech, zejména pro neurovědy a desktopové aplikace.

Jiní vědci vyvíjejí své vlastní metody, obvykle založené na programovacím jazyce s dobrou podporou počítačového vidění, jako je Krajta, C ++ nebo MATLAB. The Mahotas knihovna pro Python je jedním z populárních příkladů. Ačkoli příklady výzkumného pracovníka vyvinuly metody v programovacích jazycích s menší podporou počítačového vidění jako R existují (např. trackdem [14]).
Viz také
- Stohování zaostření Technika kombinování více obrazů s rozdílnou vzdáleností zaostření do jednoho.
- Promítání s vysokým obsahem
- digitální patologie
- Lékařské zobrazování
externí odkazy
- Vaa3D: Vysoce výkonná vícerozměrná vizualizace a analýza obrazu
- Bioformáty IO engine obrazového souboru, který podporuje desítky formátů
Reference
- ^ Peng, H; Bateman A; Valencia A; Wren JD (2012). „Bioimage informatika: nová kategorie v bioinformatice“. Bioinformatika. 28 (8): 1057. doi:10.1093 / bioinformatika / bts111. PMC 3324521. PMID 22399678.
- ^ A b Murphy, Robert; Velliste, M .; Porreca, G. (2003). "Robustní numerické funkce pro popis a klasifikaci subcelulárních lokačních vzorů na obrázcích fluorescenčního mikroskopu". The Journal of VLSI Signal Processing. 35 (3): 311–321. CiteSeerX 10.1.1.186.9521. doi:10.1023 / b: vlsi.0000003028.71666.44. S2CID 8134907.
- ^ Nattkemper, Tim; Thorsten Twellmann; Helge Ritter; Walter Schubert (2003). "Člověk vs. stroj: vyhodnocení fluorescenčních mikrofotografií". Počítače v biologii a medicíně. 33 (1): 31–43. CiteSeerX 10.1.1.324.4664. doi:10.1016 / S0010-4825 (02) 00060-4. PMID 12485628.
- ^ Peng H (září 2008). „Bioimage informatika: nová oblast inženýrské biologie“. Bioinformatika. 24 (17): 1827–36. doi:10.1093 / bioinformatika / btn346. PMC 2519164. PMID 18603566.
- ^ „Hledání kvantitativní mikroskopie“. Přírodní metody. 9 (7): 627. 2012. doi:10.1038 / nmeth.2102. PMID 22930824.
- ^ Sláva, Estelle; Justin Newberg; Robert F. Murphy (2008). "Automatizované srovnání vzorů subcelulárního umístění proteinů mezi obrazy normálních a rakovinných tkání". Biomedical Imaging: From Nano to Macro, 2008. ISBI 2008. 5. IEEE International Symposium on.
- ^ Shariff, Aabid; Joshua Kangas; Luis Pedro Coelho; Shannon Quinn; Robert F Murphy (2010). „Automatizovaná analýza obrazu pro screening a analýzu obsahu s vysokým obsahem“. Journal of Biomolecular Screening. 15 (7): 726–734. doi:10.1177/1087057110370894. PMID 20488979.
- ^ Coelho, Luis Pedro; Aabid Shariff; Robert F. Murphy (2009). „Jaderná segmentace v obrazech buněk mikroskopu: ručně segmentovaná datová sada a srovnání algoritmů“. Biomedical Imaging: From Nano to Macro, 2009. ISBI'09. IEEE International Symposium on. IEEE. doi:10.1109 / ISBI.2009.5193098. PMC 2901896.
- ^ Long, Fuhui; Peng, H .; Liu, X .; Kim, S .; Myers, E.W (září 2009). „3D digitální atlas C. elegans a jeho aplikace na jednobuněčné analýzy“. Přírodní metody. 6 (9): 667–672. doi:10.1038 / nmeth.1366. PMC 2882208. PMID 19684595.
- ^ Qu, Lei; Long, F .; Liu, X .; Kim, S .; Myers, E.W .; Peng, H. (2011). "Simultánní rozpoznávání a segmentace buněk: aplikace v C. elegans". Bioinformatika. 27 (20): 2895–2902. doi:10.1093 / bioinformatika / btr480. PMC 3187651. PMID 21849395.
- ^ Dufour, Alexandre; Vasily Shinin; Shahragim Tajbakhsh; Nancy Guillén-Aghion; J-C. Olivo-Marin; Christophe Zimmer (2005). „Segmentace a sledování fluorescenčních buněk v dynamické 3-D mikroskopii se spojenými aktivními povrchy“ (PDF). Zpracování obrazu, IEEE transakce 14., č. 9. str. 1396–1410. doi:10.1109 / TIP.2005.852790. Archivovány od originál (PDF) dne 02.03.2014..
- ^ Peng, Hanchuan; Chung, P .; Long, F .; Qu, L .; Jenett, A .; Seeds, A .; Myers, E.W .; Simpson, J.H (2011). „BrainAligner: 3D atlasy registrace mozků Drosophila“. Přírodní metody. 8 (6): 493–498. doi:10.1038 / nmeth.1602. PMC 3104101. PMID 21532582.
- ^ „Výroční zasedání Informatiky o bioimage“.
- ^ Bruijning, Marjolein; Visser, Marco D .; Hallmann, Caspar A .; Jongejans, Eelke; Golding, Nick (2018). „trackdem: Automatické sledování částic k získání počtu populací a distribucí velikostí z videí v r“. Metody v ekologii a evoluci. 9 (4): 965–973. doi:10.1111 / 2041-210X.12975. ISSN 2041-210X.
