Složení pseudoaminokyseliny - Pseudo amino acid composition
Tento článek má několik problémů. Prosím pomozte vylepši to nebo diskutovat o těchto otázkách na internetu diskusní stránka. (Zjistěte, jak a kdy tyto zprávy ze šablony odebrat) (Zjistěte, jak a kdy odstranit tuto zprávu šablony)
|
Složení pseudoaminokyselinynebo PseAAC, byl původně představen Kuo-Chen Chou v roce 2001 reprezentovat protein vzorky pro vylepšení predikce subcelulární lokalizace proteinu a membránový protein predikce typu.[1] Stejně jako metoda složení vanilkových aminokyselin (AAC) charakterizuje protein hlavně pomocí matice frekvencí aminokyselin, což pomáhá při zacházení s proteiny bez významné postupné homologie k jiným proteinům. Ve srovnání s AAC jsou do matice zahrnuty také další informace, které představují některé místní rysy, jako je korelace mezi zbytky určité vzdálenosti.[2]Při řešení případů PseAAC Chouova věta o invariance byl často používán.
Pozadí
Předvídat subcelulární lokalizace proteinů a dalších atributů na základě jejich sekvence se k reprezentaci proteinových vzorků obecně používají dva druhy modelů: (1) sekvenční model a (2) nesekvenční model nebo diskrétní model.
Nejtypičtější sekvenční reprezentace vzorku proteinu je celá aminokyselina (AA) sekvence, která může obsahovat nejúplnější informace. To je zjevná výhoda sekvenčního modelu. K získání požadovaných výsledků se k provedení predikce obvykle používají nástroje založené na vyhledávání sekvenční podobnosti.
Vzhledem k proteinové sekvenci P s aminokyselinové zbytky, tj.
kde R1 představuje první zbytek proteinu P, R.2 druhý zbytek atd. Toto je zastoupení proteinu v sekvenčním modelu.
Tento druh přístupu však selže, když dotazovaný protein nemá významnou homologii se známým proteinem (proteiny). Byly navrženy různé diskrétní modely, které se nespoléhají na pořadí. Nejjednodušší diskrétní model využívá složení aminokyselin (AAC) k reprezentaci proteinových vzorků. Podle modelu AAC protein P z Rovnice 1 lze také vyjádřit pomocí
kde jsou normalizované frekvence výskytu 20 nativních aminokyselin v P, a T provádějící operátor. AAC proteinu je triviálně odvozen od primární struktura bílkovin známý jako uveden v Rovnice 1; je to také možné hydrolýzou bez znalosti přesné sekvence a takový krok ve skutečnosti často je předpoklad pro sekvenování proteinů.[3]
Díky své jednoduchosti byl model aminokyselinového složení (AAC) široce používán v mnoha dřívějších statistických metodách pro predikci proteinových atributů. Všechny informace o pořadí sekvencí jsou však ztraceny. To je jeho hlavní nedostatek.
Pojem
Aby se zabránilo úplné ztrátě informací o pořadí sekvencí, koncept PseAAC (pseudo Amino Acid Composition).[1] Na rozdíl od konvenčního složení aminokyselin (AAC), které obsahuje 20 složek, z nichž každá odráží frekvenci výskytu jedné z 20 nativních aminokyselin v proteinu, obsahuje PseAAC soubor více než 20 samostatných faktorů, přičemž prvních 20 představuje komponenty jeho konvenční aminokyselina složení, zatímco další faktory začleňují některé informace o pořadí podle různých pseudo komponent.
Mezi další faktory patří řada hodnotově odlišných korelačních faktorů podél proteinového řetězce, ale mohou to být také jakékoli kombinace dalších faktorů, pokud mohou odrážet některé druhy efektů pořadí sekvencí tak či onak. Podstatou PseAAC je tedy to, že na jedné straně pokrývá AA složení, ale na druhé straně obsahuje informace mimo AA složení, a proto může lépe odrážet rys proteinové sekvence prostřednictvím diskrétního modelu.
Mezitím byly také vyvinuty různé režimy pro formulování vektoru PseAAC, jak je shrnuto v článku z roku 2009.[2]
Algoritmus
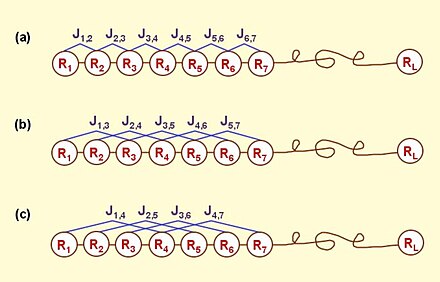
Podle modelu PseAAC protein P z Rovnice 1 lze formulovat jako
Kde () komponenty jsou dány
kde je váhový faktor a the -tý stupeň korelační faktor, který odráží korelaci pořadí sekvencí mezi všemi -té nejvíce sousedící zbytky formulované
s
kde je -tá funkce aminokyseliny , a celkový počet uvažovaných funkcí. Například v původním článku od Chou,[1] , a jsou hodnota hydrofobicity, hodnota hydrofility a hmotnost postranního řetězce aminokyseliny ; zatímco , a odpovídající hodnoty pro aminokyselinu . Celkový počet funkcí, které se zde uvažují, proto je . Je to vidět z Rovnice 3 že prvních 20 komponent, tj. jsou spojeny s konvenčním AA složením proteinu, zatímco zbývající složky jsou korelační faktory, které odrážejí 1. úroveň, 2. úroveň,… a vzory korelace pořadí pořadí tier (Obrázek 1). Prostřednictvím těchto dalších faktory, které jsou zahrnuty některé důležité efekty pořadí pořadí.
v Rovnice 3 je parametr celého čísla a ten pro něj zvolí jiné celé číslo povede k rozměrově odlišnému složení PseAA.[4]
Použitím Rovnice 6 je jen jedním z mnoha režimů pro odvození korelačních faktorů v PseAAC nebo jeho komponentách. Ostatní, například režim fyzikálně-chemické vzdálenosti[5] a režim amfifilního vzoru,[6] lze také použít k odvození různých typů PseAAC, jak je shrnuto v revizním článku z roku 2009.[2] V roce 2011 byla formulace PseAAC (Rovnice 3) byla rozšířena na formu obecného PseAAC, jak je dáno:[7]
kde dolní index je celé číslo, jeho hodnota a komponenty bude záviset na tom, jak extrahovat požadovanou informaci z aminokyselinové sekvence P v Rovnice 1.
Obecný PseAAC lze použít k vyjádření jakýchkoli požadovaných funkcí podle cílů výzkumu, včetně těch základních funkcí, jako je funkční doména, sekvenční vývoj, a genová ontologie zlepšit kvalitu predikce pro subcelulární lokalizaci proteinů.[8][9] stejně jako jejich mnoho dalších důležitých atributů.
Reference
- ^ A b C Chou KC (květen 2001). "Predikce buněčných atributů proteinu pomocí pseudoaminokyselinového složení". Proteiny. 43 (3): 246–55. doi:10,1002 / prot. 1035. PMID 11288174.
- ^ A b C Chou KC (2009). "Pseudoaminokyselinové složení a jeho aplikace v bioinformatice, proteomice a systémové biologii". Současná proteomika. 6 (4): 262–274. doi:10.2174/157016409789973707.
- ^ Michail A. Alterman; Peter Hunziker (2. prosince 2011). Analýza aminokyselin: Metody a protokoly. Humana Press. ISBN 978-1-61779-444-5.
- ^ Chou KC, Shen HB (listopad 2007). Msgstr "Nedávný pokrok v predikci subcelulárního umístění proteinu". Anální. Biochem. 370 (1): 1–16. doi:10.1016 / j.ab.2007.07.006. PMID 17698024.
- ^ Chou KC (listopad 2000). "Predikce proteinových subcelulárních lokací začleněním kvazi-sekvenčního efektu". Biochem. Biophys. Res. Commun. 278 (2): 477–83. doi:10,1006 / bbrc.2000.3815. PMID 11097861.
- ^ Chou KC (leden 2005). „Použití amfifilního pseudoaminokyselinového složení k předpovědi tříd podrodin enzymů“. Bioinformatika. 21 (1): 10–9. doi:10.1093 / bioinformatika / bth466. PMID 15308540.
- ^ Chou KC (březen 2011). „Některé poznámky k predikci atributů bílkovin a složení pseudoaminokyselin“. Journal of Theoretical Biology. 273 (1): 236–47. doi:10.1016 / j.jtbi.2010.12.024. PMC 7125570. PMID 21168420.
- ^ Chou KC, Shen HB (2008). „Cell-PLoc: balíček webových serverů pro predikci subcelulární lokalizace proteinů v různých organismech“. Nat Protoc. 3 (2): 153–62. doi:10.1038 / nprot.2007.494. PMID 18274516. Archivovány od originál dne 2007-08-27. Citováno 2008-03-24.
- ^ Shen HB, Chou KC (únor 2008). "PseAAC: flexibilní webový server pro generování různých druhů složení bílkovinných pseudoaminokyselin". Anální. Biochem. 373 (2): 386–8. doi:10.1016 / j.ab.2007.10.012. PMID 17976365.








![p_ {u} = {egin {cases} {dfrac {f_ {u}} {sum _ {{i = 1}} ^ {{20}} f_ {i}, +, wsum _ {{k = 1}} ^ {{lambda}} au _ {k}}}, & (1leq uleq 20) [10pt] {dfrac {w au _ {{u-20}}} {sum _ {{i = 1}} ^ { {20}} f_ {i}, +, wsum _ {{k = 1}} ^ {{lambda}} au _ {k}}}, a (20 + 1 leq uleq 20 + lambda) končí {případy}} qquad {ext {(4)}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/9e51062076d0296d9b652c39f3ba5e010aabeb2b)




![{mathrm {J}} _ {{i, i + k}} = {frac {1} {Gamma}} součet _ {{q = 1}} ^ {{Gamma}} zbývá [Phi _ {{q}} left ({mathrm {R}} _ {{i + k}} ight) -Phi _ {{q}} left ({mathrm {R}} _ {{i}} ight) ight] ^ {2} qquad { ext {(6)}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/95f4c0827bc12464e0e8ca739608a3a42081c0d2)

















