Dokonalá fylogeneze - Perfect phylogeny - Wikipedia
Dokonalá fylogeneze je termín používaný v výpočetní fylogenetika označit a fylogenetický strom ve kterém mohou být všechny vnitřní uzly označeny tak, že se všechny znaky vyvinou dolů ve stromu homoplazmy. To znamená, že vlastnosti nedrží evoluční konvergence, a nemají analogický struktur. Statisticky to lze reprezentovat jako předchůdce se stavem „0“ ve všech charakteristikách, kde 0 představuje nedostatek této charakteristiky. Každá z těchto charakteristik se mění z 0 na 1 přesně jednou a nikdy se nevrátí do stavu 0. Je zřídka, že skutečná data dodržují koncept dokonalé fylogeneze.[1][2]
Budova
Obecně existují dva různé datové typy, které se používají při konstrukci fylogenetického stromu. Ve výpočtech založených na vzdálenosti je fylogenetický strom vytvořen analýzou vztahů mezi vzdáleností mezi druhy a délkami okrajů odpovídajícího stromu. Použití znakově založeného přístupu využívá charakterové stavy napříč druhy jako vstup ve snaze najít nejvíce „dokonalý“ fylogenetický strom.[3][4]
Statistické komponenty dokonalého fylogenetického stromu lze nejlépe popsat takto:[3]
Perfektní fylogeneze pro n X m matice stavu znaků M je zakořeněný strom T s n listy uspokojující:
i. Každá řada M označí přesně jeden list T
ii. Každý sloupec M označí přesně jednu hranu T
iii. Každá vnitřní hrana T je označen alespoň jedním sloupcem Miv. Znaky spojené s okraji podél jedinečné cesty od kořene k listu proti přesně určit znakový vektor proti, tj. znakový vektor má ve všech sloupcích 1 položku odpovídající znakům spojeným s hranami cesty a jinak 0.
Stojí za zmínku, že je velmi vzácné najít skutečná fylogenetická data, která dodržují zde popsané koncepty a omezení. Proto se často stává, že jsou vědci nuceni dělat kompromisy vývojem stromů, které se jednoduše snaží minimalizovat homoplasii, nalezení sady maximálních mohutností kompatibilních znaků nebo konstrukcí fylogenií, které se co nejvíce shodují s oddíly implikovanými postavami.
Příklad
Obě tyto datové sady ilustrují příklady stavu znaků matice. Používání matice M '1 je možné pozorovat, že výsledný fylogenetický strom může být vytvořen tak, že každá z postav označuje přesně jeden okraj stromu. Naproti tomu při pozorování matice M '2, je vidět, že neexistuje způsob, jak nastavit fylogenetický strom tak, aby každý znak označil pouze jednu délku okraje.[3] Pokud vzorky pocházejí z údajů o variantní alelické frekvenci (VAF) populace studovaných buněk, jsou položky v matici znaků frekvencemi mutací a mají hodnotu mezi 0 a 1. Jmenovitě, pokud představuje pozici v genomu, pak záznam odpovídající a vzorek udrží frekvence genomů ve vzorku s mutací v poloze .[5][6][7][8][9]
- Matice stavu znaků
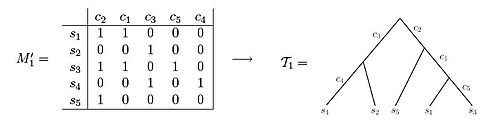
Příklad matice znaků, kterou lze znázornit jako dokonalou fylogenezi
Používání
Dokonalá fylogeneze je teoretický rámec, který lze také použít v praktičtějších metodách. Jedním z takových příkladů je neúplná řízená dokonalá fylogeneze. Tento koncept zahrnuje využití dokonalých fylogenií se skutečnými, a proto neúplnými a nedokonalými datovými soubory. Taková metoda využívá SINE k určení evoluční podobnosti. Tyto krátce proložené prvky jsou přítomny v mnoha genomech a lze je identifikovat podle jejich sousedních sekvencí. SINE poskytují informace o dědictví určitých rysů u různých druhů. Bohužel, pokud chybí SINE, je obtížné zjistit, zda tyto SINE byly přítomny před odstraněním. Použitím algoritmů odvozených z dokonalých fylogenetických dat jsme schopni se pokusit o rekonstrukci fylogenetického stromu i přes tato omezení.[10]
Dokonalá fylogeneze se také používá při stavbě mapy haplotypu. Použitím konceptů a algoritmů popsaných v dokonalé fylogenezi lze určit informace týkající se chybějících a nedostupných dat haplotypu.[11] Za předpokladu, že soubor haplotypů, které jsou výsledkem mapování genotypů, odpovídá a dodržuje koncept dokonalé fylogeneze (stejně jako další předpoklady, jako je dokonalá Mendelianova dědičnost a skutečnost, že na SNP existuje pouze jedna mutace), je možné odvodit chybí data haplotypu.[12][13][14] [15]
Odvodit fylogenezi od hlučných dat VAF v rámci PPM je těžký problém.[5] Většina nástrojů pro odvození obsahuje určitý heuristický krok, který umožňuje výpočet odvodit. Mezi příklady nástrojů, které odvozují fylogeneze z hlučných dat VAF, patří AncesTree, Canopy, CITUP, EXACT a PhyloWGS.[5][6][7][8][9] Zejména EXACT provádí přesnou inference pomocí GPU k výpočtu zadní pravděpodobnosti na všech možných stromech pro problémy malé velikosti. Rozšíření PPM byla provedena pomocí doprovodných nástrojů.[16][17] Například nástroje jako MEDICC, TuMult a FISHtrees umožňují zvýšit nebo snížit počet kopií daného genetického prvku nebo ploidie, což účinně umožňuje odstranění mutací.[18][19][20]
externí odkazy
- Seznam fylogenetického softwaru
- Jeden z několika programů dostupných pro analýzu a tvorbu fylogenetických stromů
- Další takový program pro analýzu fylogenetických stromů
- Další program pro analýzu stromů
- Článek popisující příklad toho, jak lze dokonalou fylogenezi využít mimo oblast genetiky, jako v jazykové asociaci
- Github pro „Algoritmus pro rekonstrukci klonálního stromu z údajů o sekvenování rakoviny u více vzorků“ (AncesTree)
- Github pro „Přístup k intra-nádorové heterogenitě a sledování podélné a prostorové klonální evoluční historie sekvenováním nové generace“ (Canopy)
- Github pro „odvození klonality u nádorů pomocí fylogeneze“ (CITUP)
- Github pro „Přesný závěr podle dokonalého modelu fylogeneze“ (PŘESNÝ)
- Github pro „Rekonstrukce subklonálního složení a evoluce z celogenomomového sekvenování nádorů“ (PhyloWGS)
Reference
- ^ Fernandez-Baca D. „Dokonalý problém s fylogenezí“ (PDF). Kluwer Academic Publishers. Citováno 30. září 2012.
- ^ Nakhleh L, Ringe D, Warnow T. „Dokonalé fylogenetické sítě: nová metodika rekonstrukce evoluční historie přirozených jazyků“ (PDF). Citováno 1. října 2012.
- ^ A b C Uhler C.. „Hledání dokonalé fylogeneze“ (PDF). Archivovány od originál (PDF) dne 4. března 2016. Citováno 29. září 2012.
- ^ Nikaido M, Rooney AP, Okada N (srpen 1999). „Fylogenetické vztahy mezi cetartiodaktyly založené na vložení krátkých a dlouhých vzájemně narušených prvků: hrochy jsou nejbližší existující příbuzní velryb“. Sborník Národní akademie věd Spojených států amerických. 96 (18): 10261–6. Bibcode:1999PNAS ... 9610261N. doi:10.1073 / pnas.96.18.10261. PMC 17876. PMID 10468596.
- ^ A b C El-Kebir M, Oesper L, Acheson-Field H, Raphael BJ (červen 2015). "Rekonstrukce klonálních stromů a složení nádoru z vícevzorkových sekvenčních dat". Bioinformatika. 31 (12): i62-70. doi:10.1093 / bioinformatika / btv261. PMC 4542783. PMID 26072510.
- ^ A b Satas G, Raphael BJ (červenec 2017). „Odvození fylogeneze nádoru pomocí vzorkování důležitosti omezené stromem“. Bioinformatika. 33 (14): i152 – i160. doi:10.1093 / bioinformatika / btx270. PMC 5870673. PMID 28882002.
- ^ A b Malikic S, McPherson AW, Donmez N, Sahinalp CS (květen 2015). „Odvození klonality ve více vzorcích nádorů pomocí fylogeneze“. Bioinformatika. 31 (9): 1349–56. doi:10.1093 / bioinformatika / btv003. PMID 25568283.
- ^ A b Ray S, Jia B, Safavi S, van Opijnen T, Isberg R, Rosch J, Bento J (2019-08-22). Msgstr "Přesný závěr podle dokonalého modelu fylogeneze". arXiv:1908.08623v1. Bibcode:2019arXiv190808623R. Citovat deník vyžaduje
| deník =(Pomoc) - ^ A b Deshwar AG, Vembu S, Yung CK, Jang GH, Stein L, Morris Q (únor 2015). „PhyloWGS: rekonstrukce subklonálního složení a evoluce z genomového sekvenování nádorů“. Genome Biology. 16 (1): 35. doi:10.1186 / s13059-015-0602-8. PMC 4359439. PMID 25786235.
- ^ Pe'er I, Pupko T, Shamir R, Sharan R. „Neúplná řízená dokonalá fylogeneze“. Tel-Aviv University. Archivovány od originálu dne 20. října 2013. Citováno 30. října 2012.CS1 maint: BOT: stav původní adresy URL neznámý (odkaz)
- ^ Eskin E, Halperin E, Karp RM (duben 2003). "Efektivní rekonstrukce struktury haplotypu pomocí dokonalé fylogeneze" (PDF). Journal of Bioinformatics and Computational Biology. University of California, Berkeley. 1 (1): 1–20. doi:10.1142 / S0219720003000174. PMID 15290779. Citováno 30. října 2012.
- ^ Gusfield D. „Přehled výpočetních metod pro odvození haplotypu“ (PDF). University of California, Davis. Citováno 18. listopadu 2012.
- ^ Ding Z, Filkov V, Gusfield D. „Lineární časový algoritmus pro perfektní problém fylogenetické haplotypizace“. University of California, Davis. Citováno 18. listopadu 2012.
- ^ Bafna V, Gusfield D, Lancia G, Yooseph S (2003). "Haplotypování jako dokonalá fylogeneze: přímý přístup". Journal of Computational Biology. 10 (3–4): 323–40. doi:10.1089/10665270360688048. PMID 12935331.
- ^ Seyalioglu H. „Haplotypování jako dokonalá fylogeneze“ (PDF). Archivovány od originál (PDF) dne 30. září 2011. Citováno 30. října 2012.
- ^ Bonizzoni P, Carrieri AP, Della Vedova G, Trucco G (říjen 2014). „Vysvětlení evoluce prostřednictvím omezené trvalé perfektní fylogeneze“. BMC Genomics. 15 Suppl 6 (S6): S10. doi:10.1186 / 1471-2164-15-S6-S10. PMC 4240218. PMID 25572381.
- ^ Hajirasouliha I, Raphael BJ (2014), Brown D, Morgenstern B (eds.), „Rekonstrukce historie mutace v nádorech s více vzorky pomocí dokonalých směsí fylogeneze“, Algoritmy v bioinformaticeSpringer Berlin Heidelberg, 8701, str. 354–367, doi:10.1007/978-3-662-44753-6_27, ISBN 9783662447529
- ^ Schwarz RF, Trinh A, Sipos B, Brenton JD, Goldman N, Markowetz F (duben 2014). Beerenwinkel N (ed.). "Fylogenetická kvantifikace intra-nádorové heterogenity". PLoS výpočetní biologie. 10 (4): e1003535. arXiv:1306.1685. Bibcode:2014PLSCB..10E3535S. doi:10.1371 / journal.pcbi.1003535. PMC 3990475. PMID 24743184.
- ^ Letouzé E, Allory Y, Bollet MA, Radvanyi F, Guyon F (2010). „Analýza profilů počtu kopií několika vzorků nádorů od stejného pacienta odhaluje postupné kroky v tumorigenezi“. Genome Biology. 11 (7): R76. doi:10.1186 / gb-2010-11-7-r76. PMC 2926787. PMID 20649963.
- ^ Gertz EM, Chowdhury SA, Lee WJ, Wangsa D, Heselmeyer-Haddad K, Ried T a kol. (2016-06-30). „FISHtrees 3.0: Fylogenetika nádorů pomocí ploidní sondy“. PLOS ONE. 11 (6): e0158569. Bibcode:2016PLoSO..1158569G. doi:10.1371 / journal.pone.0158569. PMC 4928784. PMID 27362268.



