NAIL-MS - NAIL-MS
NAIL-MS (zkratka pro nukleová kyselina izotop označení spojené hmotnostní spektrometrie ) je technika založená na hmotnostní spektrometrii používaná pro výzkum nukleových kyselin a jejich modifikace. Umožňuje různým návrhům experimentů studovat základní mechanismus biologie RNA in vivo. Například dynamické chování nukleových kyselin v živých buňkách, zejména z RNA modifikace, lze sledovat podrobněji.[1]
Teorie
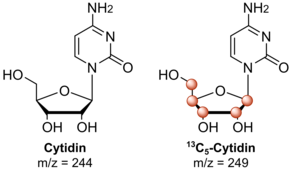
NAIL-MS se používá ke studiu mechanismů modifikace RNA. Proto jsou buňky v kultuře nejprve napájeny stabilními izotopem značenými živinami a buňky je začleňují do svých biomolekul. Po čištění nukleových kyselin nejčastěji RNA, analýza se provádí hmotnostní spektrometrií. Hmotnostní spektrometrie je analytická technika, která měří poměr hmotnosti k náboji z ionty. Páry chemicky identických nukleosidů různého stabilního izotopového složení lze diferencovat v hmotnostním spektrometru kvůli jejich hmotnostnímu rozdílu. Neoznačené nukleosidy lze proto odlišit od značeného stabilního izotopu izotopologové. U většiny přístupů NAIL-MS je zásadní, aby značených nukleosidů bylo více než 2 Da těžší než neoznačené. Je to proto, že 1,1% přirozeně se vyskytujících atomů uhlíku je 13C izotopy. V případě nukleosidů to vede k nárůstu hmotnosti o 1 Da u ~ 10% nukleosidů. Tento signál by narušil konečné vyhodnocení měření.
NAIL-MS lze použít ke zkoumání dynamiky modifikace RNA změnou značených živin odpovídajícího růstového média během experimentu. Kromě toho lze buněčné populace navzájem přímo porovnávat bez účinků zkreslení čištění. Dále může být použit pro výrobu biosyntetických izotopologů většiny nukleosidů, které jsou potřebné pro kvantifikaci pomocí hmotnostní spektrometrie a dokonce pro objev dosud neznámých RNA modifikací.[2][3][4]
Obecný postup

Obecně jsou buňky kultivovány v neznačeném nebo stabilním (neradioaktivním) izotopem značeném médiu. Médium může například obsahovat glukózu označenou šesti uhlík-13 atomy (13C) namísto normálu uhlík-12 (12C). Buňky rostoucí v tomto médiu začlení v závislosti na modelovém organismu těžkou glukózu do všech svých molekul RNA. Poté vše nukleotidy jsou o 5 Da těžší než jejich neoznačené izotopologové díky úplnému uhlíkovému označení ribózy. Po kultivaci a vhodném značení buněk se buňky obvykle sklidí za použití fenolu / chloroformu / guanidiniumisothiokyanátu. Jiné extrakční metody jsou možné a někdy nutné (např. Pro kvasinky). RNA je poté izolována Extrakce fenol-chloroformem a iso-Propanol srážky. Další čištění specifických druhů RNA (např. RRNA, tRNA) se obvykle provádí pomocí vylučovací chromatografie (SEC), ale jsou k dispozici i jiné přístupy. U většiny aplikací musí být konečný produkt před analýzou pomocí enzymaticky štěpen na nukleosidy LC-MS. Proto trávicí enzymy, jako je benzonáza, NP1 a CIP Jsou používány.[5][6] Typicky, a trojitý kvadrupól v režimu MRM se používá pro měření.
Označování buněk
Způsob, jakým je dosaženo značení molekul RNA, závisí na modelovém organismu. Pro E-coli (bakterie ) lze použít minimální médium M9 a doplnit je o stabilní izotopem označené varianty potřebných solí. To umožňuje označování pomocí 13C-uhlík, 15N-dusík, 34S-síra a 2H-vodík.[7] v S. cerevisiae (droždí ) v současné době existují dvě možnosti: Za prvé, použití komerčně dostupného kompletního růstového média, které umožňuje značení pomocí 13C-uhlík a / nebo 15N-dusík a za druhé použití minimálního YNB média, které musí být doplněno několika aminokyseliny a glukóza které lze přidat jako stabilní izotopem označené varianty, aby bylo dosaženo 13C-uhlík, 15N-dusík a 2H-vodíkové značení RNA.[8]
Při označování na modelových organismech jako E-coli a S. cerevisiae je poměrně jednoduché a stabilní označení izotopů v systému Windows buněčná kultura je mnohem náročnější, protože složení růstového média je složitější. Ani doplnění stabilní izotopem značené glukózy, ani doplnění stabilních izotopem značených variant jednoduchých prekurzorů biosyntézy nukleosidů, jako je glutamin a / nebo aspartát mít za následek definované zvýšení hmotnosti vyšší než 2 Da. I když již byly nalezeny vhodné sloučeniny pro úplné značení v buněčné kultuře, tyto výsledky ještě nejsou publikovány.
Aplikace
S NAIL-MS jsou možné různé návrhy experimentů.
Výroba SILIS
NAIL-MS lze použít k výrobě stabilních izotopem značených vnitřních standardů (ISTD). Proto se buňky pěstují v médiu, které vede k úplnému značení všech nukleosidů. Vyčištěná směs nukleosidů může být poté použita jako ISTD, která je nutná pro přesnou absolutní kvantifikaci nukleosidů hmotnostní spektrometrií. Tato směs značených nukleosidů se také označuje jako SILIS (stabilní izotopem značený vnitřní standard).[9] Výhodou tohoto přístupu je, že všechny modifikace přítomné v organismu mohou být biosyntetizovány jako značené sloučeniny. Výroba SILIS byla provedena již před vznikem termínu NAIL-MS.
Srovnávací experimenty
Srovnávací experiment NAIL-MS je velmi podobný a SILAC experiment, ale místo RNA místo proteinů. Nejprve se kultivují dvě populace příslušných buněk. Jedna z populací buněk je krmena růstovým médiem obsahujícím neznačené živiny, zatímco druhá populace je krmena růstovým médiem obsahujícím stabilní izotopem značené živiny. Buňky pak začleňují příslušné izotopology do svých molekul RNA. Jedna z populací buněk slouží jako kontrolní skupina, zatímco druhá je předmětem souvisejícího výzkumu (např. Kmen KO, stres). Po sklizni dvou buněčných populací se smísí a společně zpracují, aby se vyloučilo zkreslení purifikace. Vzhledem k odlišné hmotnosti zabudovaných živin do nukleosidů je možná diferenciace dvou populací buněk pomocí hmotnostní spektrometrie.
Pulse-Chase experimenty
Po zahájení experimentu s pulzním chase se médium přepne ze média (1) na médium (2). Obě média se musí lišit pouze obsahem izotopů. Tím je možné rozlišovat mezi molekulami RNA, které již existovaly před zahájením experimentu (= molekuly RNA pěstované v médiu (1)) a molekulami RNA, které jsou nově transkribovány po zahájení experimentu (= molekuly RNA pěstované v médiu (2)). To umožňuje podrobné studium dynamiky modifikací in vivo. Doplnění značeného methioninu buď v médiu (1), nebo v médiu (2) umožňuje sledování methylačních procesů. Jiné izotopově značené metabolity potenciálně umožňují další analýzu modifikací.
NAIL-MS celkem umožňuje zkoumání dynamiky modifikace RNA hmotnostní spektrometrií. S touto technikou byla pozorována enzymatická demetylace pro několik poškození RNA uvnitř živých bakterií.[4][7]
Objev nových modifikací RNA
Pro objev necharakterizovaných modifikací se buňky pěstují v neznačeném stavu nebo 13Označeno písmenem C. nebo 15Označeno N nebo 2Označeno písmenem H. nebo 34Médium označené značkou S. Neznámé signály vyskytující se během hmotnostní spektrometrie jsou poté kontrolovány ve všech odlišně označených kulturách. Pokud jsou retenční časy neznámých sloučenin s vhodně odlišnými m / z hodnoty se překrývají, lze vypočítat souhrnný vzorec sloučeniny výpočtem hmotnostních rozdílů překrývajícího se signálu v odlišně značených kulturách. S touto metodou bylo možné objevit několik nových modifikací RNA. Tento experimentální design byl také počáteční myšlenkou, která odstartovala koncept NAIL-MS.
Oligonukleotid NAIL-MS
NAIL-MS lze také použít k analýze oligonukleotidů hmotnostní spektrometrií. To je užitečné, pokud mají být zachovány informace o sekvenci.[10]
Reference
- ^ Reichle, Valentin F .; Kaiser, Steffen; Heiss, Matthias; Hagelskamp, Felix; Borland, Kayla; Kellner, Stefanie (1. března 2019). „Překonání limitů statické analýzy modifikace RNA s dynamickým NAIL-MS“. Metody (San Diego, Kalifornie). 156: 91–101. doi:10.1016 / j.ymeth.2018.10.025. ISSN 1095-9130. PMID 30395967.
- ^ Kellner, Stefanie; Neumann, Jennifer; Rosenkranz, David; Lebedeva, Svetlana; Ketting, René F .; Zischler, Hans; Schneider, Dirk; Helm, Mark (4. dubna 2014). "Profilování modifikací RNA multiplexovaným stabilním značením izotopů". Chemická komunikace (Cambridge, Anglie). 50 (26): 3516–3518. doi:10.1039 / c3cc49114e. ISSN 1364-548X. PMID 24567952.
- ^ Dal Magro, Christina; Keller, Patrick; Kotter, Annika; Werner, Stephan; Duarte, Victor; Marchand, Virginie; Ignarski, Michael; Freiwald, Anja; Müller, Roman-Ulrich; Dieterich, Christoph; Motorin, Yuri (25. června 2018). „Výrazně zvýšená chemická rozmanitost modifikací RNA obsahující thioacetální strukturu“. Angewandte Chemie International Edition v angličtině. 57 (26): 7893–7897. doi:10.1002 / anie.201713188. ISSN 1521-3773. PMID 29624844.
- ^ A b Reichle, Valentin F .; Petrov, Dimitar P .; Weber, Verena; Jung, Kirsten; Kellner, Stefanie (6. prosince 2019). „NAIL-MS odhaluje opravu 2-methylthiocytidinu AlkB v E. coli“. Příroda komunikace. 10 (1): 5600. Bibcode:2019NatCo..10.5600R. doi:10.1038 / s41467-019-13565-9. ISSN 2041-1723. PMC 6898146. PMID 31811240.
- ^ Quinlivan, Eoin P .; Gregory, Jesse F. (15. února 2008). „Digesce DNA na deoxyribonukleosid: zjednodušený jednostupňový postup“. Analytická biochemie. 373 (2): 383–385. doi:10.1016 / j.ab.2007.09.031. ISSN 0003-2697. PMC 2239294. PMID 18028864.
- ^ Crain, P. F. (1990). "Příprava a enzymatická hydrolýza DNA a RNA pro hmotnostní spektrometrii". Metody v enzymologii. 193: 782–790. doi:10.1016 / 0076-6879 (90) 93450 let. ISSN 0076-6879. PMID 1706062.
- ^ A b Reichle, Valentin F .; Weber, Verena; Kellner, Stefanie (18. prosince 2018). „NAIL-MS v E. coli určuje zdroj a osud methylace v tRNA“. Chembiochem: A European Journal of Chemical Biology. 19 (24): 2575–2583. doi:10.1002 / cbic.201800525. ISSN 1439-7633. PMC 6582434. PMID 30328661.
- ^ Heiss, Matthias; Reichle, Valentin F .; Kellner, Stefanie (2. září 2017). "Pozorování osudu tRNA a jejích modifikací pomocí hmotnostní spektrometrie značení izotopů nukleové kyseliny: NAIL-MS". RNA Biology. 14 (9): 1260–1268. doi:10.1080/15476286.2017.1325063. ISSN 1555-8584. PMC 5699550. PMID 28488916.
- ^ Kellner, Stefanie; Ochel, Antonia; Thüring, Kathrin; Spenkuch, Felix; Neumann, Jennifer; Sharma, Sunny; Entian, Karl-Dieter; Schneider, Dirk; Helm, Mark (16. srpna 2014). „Absolutní a relativní kvantifikace modifikací RNA prostřednictvím biosyntetických izotopomerů“. Výzkum nukleových kyselin. 42 (18): e142. doi:10.1093 / nar / gku733. ISSN 1362-4962. PMC 4191383. PMID 25129236.
- ^ Hagelskamp, Felix; Borland, Kayla; Ramos, Jillian; Hendrick, Alan G .; Fu, Dragony; Kellner, Stefanie (21. února 2020). „Široce použitelná hmotnostní spektrometrie oligonukleotidů pro analýzu zapisovačů a mazadel RNA in vitro“. Výzkum nukleových kyselin. 48 (7): e41. doi:10.1093 / nar / gkaa091. ISSN 1362-4962. PMC 7144906. PMID 32083657.
