MA spiknutí - MA plot
tento článek poskytuje nedostatečný kontext pro ty, kteří danému tématu nejsou obeznámeni. (Říjen 2009) (Zjistěte, jak a kdy odstranit tuto zprávu šablony) |
An MA spiknutí je aplikace a Bland – Altmanova zápletka pro vizuální znázornění genomický data. Graf zobrazuje rozdíly mezi měřeními provedenými ve dvou vzorcích transformací dat na M (logaritmický poměr) a A (průměrný průměr ) váhy, pak tyto hodnoty vykreslí. Ačkoli původně aplikován v kontextu dvou kanálů DNA microarray data genové exprese, MA grafy se také používají k vizualizaci vysoce výkonné sekvenování analýza.[1][2]
Vysvětlení
Microarray data jsou často normalizována v polích ke kontrole systematických předsudků v účinnosti spojování a hybridizace barviv, jakož i dalších technických předsudků v DNA sondách a tiskové špičce používané k vyhledání pole.[3] Minimalizací těchto systematických variací lze nalézt skutečné biologické rozdíly. K určení, zda je nutná normalizace, lze vykreslit Cy5 (R) intenzity proti Cy3 (G) intenzity a zjistěte, zda je sklon čáry kolem 1. Vylepšená metoda, která je v podstatě zmenšená, 45 stupňová rotace grafu R vs. G, je grafem MA.[4] Graf MA je graf distribuce poměru červené / zelené intenzity („M“) vynesený průměrnou intenzitou („A“). M a A jsou definovány následujícími rovnicemi.
M je tedy binární logaritmus poměru intenzity (nebo rozdílu mezi logovými intenzitami) a A je průměrná logaritmická intenzita pro tečku v grafu. MA grafy se poté používají k vizualizaci poměru intenzity nezpracovaných dat microarray (mikročipy zde obvykle vykazují zkreslení, přičemž vyšší A vede k vyššímu | M |, tj. Čím světlejší je bod, tím je pravděpodobnější pozorovaný rozdíl mezi vzorkem a kontrolou). Graf MA vloží proměnnou M na y- osa a A na X- osa a poskytuje rychlý přehled o rozdělení údajů.
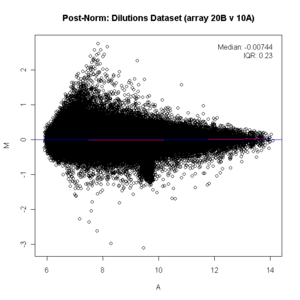
V mnoha experimentech s genovou expresí microarray je základním předpokladem to, že většina genů by neviděla žádnou změnu v jejich expresi; proto většina bodů na internetu y- osa (M) by byl umístěn na 0, protože log (1) je 0. Pokud tomu tak není, pak a normalizace metoda jako MNOHO by měly být použity na data před statistickou analýzou. (Na níže uvedeném diagramu viz červená čára běžící pod nulovou značkou před normalizací, měla by být přímá. Protože to není přímé, data by měla být normalizována. Po normalizaci je červená čára rovně na nulové čáře a ukazuje jako růžová / černá.)
Balíčky
Několik Biovodič balíčky pro R software, poskytnout zařízení pro vytváření MA pozemků. Patří mezi ně affy (ma.plot, mva.pair), limma (plotMA), marray (maPlot) a edgeR (maPlot)
Podobný "RA" pozemky lze generovat pomocí funkce raPlot v karolině CRAN R balík.
Příklad v programovacím jazyce R.
knihovna(afy)-li (vyžadovat(affydata)) { data(Ředění)}y <- (exprs(Ředění)[, C(„20B“, „10A“)])x11()ma.plot( řádek znamená(log2(y)), log2(y [, 1])-log2(y [, 2]), cex=1 )titul(„Datová sada ředění (pole 20B v 10A)“)knihovna(preprocesCore)#do normalizace kvantilX <- normalizovat. kvantity(y)x11()ma.plot( řádek znamená(log2(X)), log2(X[, 1])-log2(X[, 2]), cex=1 ) titul(„Post Norm: Dilutions Dataset (pole 20B v 10A)“)
Viz také
Reference
- ^ Robinson, M. D .; McCarthy, D. J .; Smyth, G. K. (11. listopadu 2009). „edgeR: balíček Bioconductor pro diferenciální analýzu exprese dat digitální genové exprese“. Bioinformatika. 26 (1): 139–140. doi:10.1093 / bioinformatika / btp616. PMC 2796818. PMID 19910308.
- ^ Láska, Michael I; Huber, Wolfgang; Anders, Simon (5. prosince 2014). „Moderovaný odhad změny násobení a disperze pro data RNA-seq s DESeq2“. Genome Biology. 15 (12): 550. doi:10.1186 / s13059-014-0550-8. PMC 4302049. PMID 25516281.
- ^ YH Yang, S Dudoit, P Luu, DM Lin, V Peng, J Ngai, Rychlost TP. (2002). Normalizace pro data cDNA microarray: robustní kompozitní metoda zaměřená na systematické variace jednoho a více snímků. Výzkum nukleových kyselin sv. 30 (4), str. E15.
- ^ Dudoit, S, Yang, YH, Callow, MJ, Rychlost, TP. (2002). Statistické metody pro identifikaci rozdílně exprimovaných genů v replikovaných cDNA microarray experimentech. Stat. Hřích. 12:1 111–139


